利用vcftools比较两个vcf文件
Posted 天使不设防
tags:
篇首语:本文由小常识网(cha138.com)小编为大家整理,主要介绍了利用vcftools比较两个vcf文件相关的知识,希望对你有一定的参考价值。
因为最近有一项工作是比较填充准确性的,中间有用到vcftools比较两个vcf文件。
使用命令也很简单:
1 vcftools --vcf file1.snp.vcf --diff file2.snp.vcf --diff-site --out Diff.site
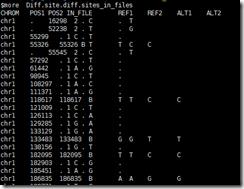
运行结束会生成一个名为Diff.site.diff.sites_in_files的文件:
pso1,ref1和alt1代表file1.snp.vcf文件中位点信息,IN_FILE列代表含有当前snp位置信息的文件,1代表file1.snp.vcf含有此位点,2代表file2.snp.vcf含有此位点,B代表两个文件都含有此位点。根据此结果就可以做一些基本的统计了。
以上是关于利用vcftools比较两个vcf文件的主要内容,如果未能解决你的问题,请参考以下文章