使用mafft进行多序列比对
Posted
tags:
篇首语:本文由小常识网(cha138.com)小编为大家整理,主要介绍了使用mafft进行多序列比对相关的知识,希望对你有一定的参考价值。
参考技术A多序列比对(多序列联配,Multiple sequence alignment, MSA) 是指把多条(3 条或以上)有系统进化关系的蛋白质分子的氨基酸序列或核酸序列进行比对,尽可能地把相同的碱基或氨基酸残基排在同一列上。这样做的意义是,对齐的碱基或氨基酸残基在进化上是同源的,即来自共同祖先(common ancestor)。
多序列比对的软件有很多,最常用的多的序列比对软件是 clustalw 。 有文献报道:比对速度(Muscle>MAFFT>ClustalW>T-Coffee),比对准确性(MAFFT>Muscle>T-Coffee>ClustalW)。因此,推荐使用 MAFFT 软件进行多序列比对。
使用了之前注释的水稻基因组数据,将其转换成蛋白质文件,
使用conda安装mafft,输入mafft,显示版本信息
按照软件要求输入信息,
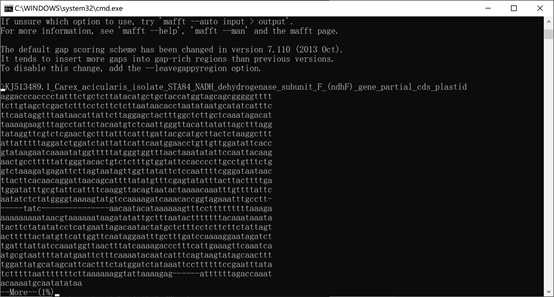
成功运行
How to alignment by MAFFT
MAFFT比对快速且准确,常用做序列之间比对。
MAFFT官方下载地址
https://mafft.cbrc.jp/alignment/software/
下载适合自己电脑的MAFFT版本,打开mafft.bat,操作界面如下:
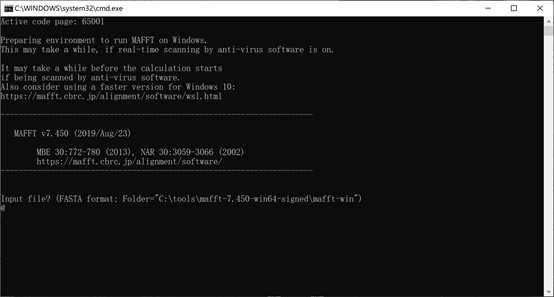
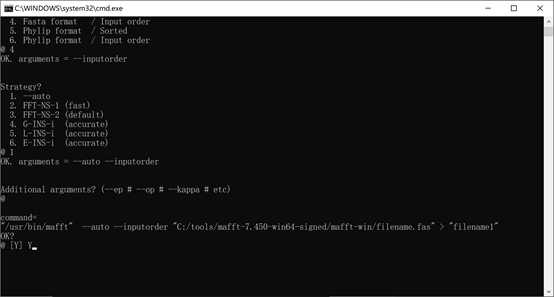
将需要比对的FASTA格式的文件(以filename.fas为例)放入MAFFT根目录文件夹(我的是mafft-win)中,在@后输入文件名filename.fas,如下:
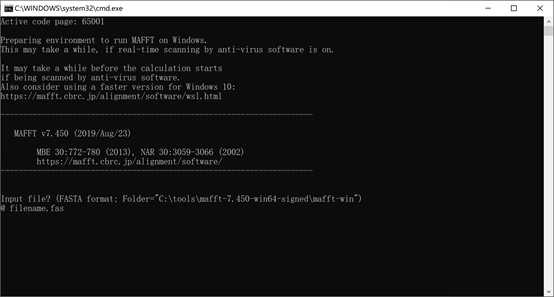
回车,输入 输出的文件名(这里命名为filename1.fas),如下:

回车,输入4(设置输出文件格式为Fasta format),如下:
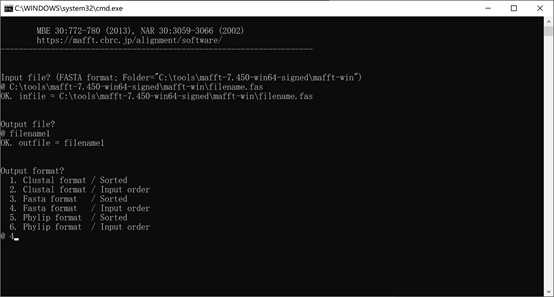
回车,输入1(一般设置alignment策略为自动),如下:

回车。接着设置附加参数,但是一般不设置附加参数,可直接再回车,如下:
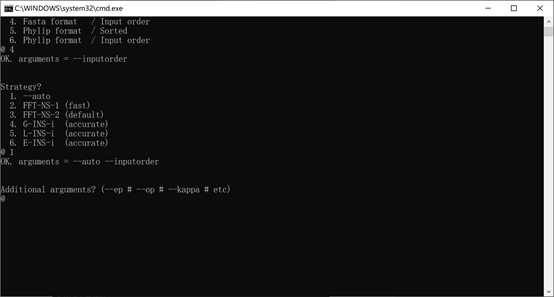

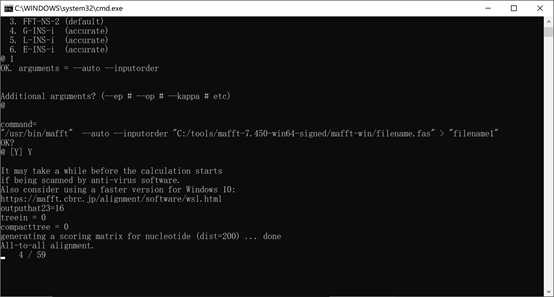
输入Y后,回车,开始比对。中途若停止,按住Enter键,直至比对完。比对完的文件也保存于根目录中。
以上是关于使用mafft进行多序列比对的主要内容,如果未能解决你的问题,请参考以下文章