jbrowse和NGL展示基因组
Posted BH4LM
tags:
篇首语:本文由小常识网(cha138.com)小编为大家整理,主要介绍了jbrowse和NGL展示基因组相关的知识,希望对你有一定的参考价值。
最近朋友介绍了一个项目,用jbrowse和NGL来展示基因组,对于没有生物基础的我来说是个不小的挑战,但是咱10岁就开始用学习机写程序,这点问题还是难不倒我的(吹点小牛,开始正题)。
jbrowse和NGL都是用javascript开发,所以有web开发经验的同学应该都比较好上手。
jbrowse安装
我使用的是JBrowse-1.12.1-dev.zip这个版本,不要使用不带dev的版本,这样在开发中会缺少一些js包。
安装过程比较简单,我使用的CentOS7,首先准备安装所需要的环境
yum groupinstall "Development Tools" yum install gcc gcc-c++ libpng-devel gd-devel zlib-devel perl-ExtUtils-MakeMaker
然后运行解压后的目录中的setup.sh就可以了,直接访问url就可以了。如果安装过程中出现问题,可以查看setup.log文件。
我们在首次安装好以后就有过一个错误提示,提示我们找不到data/seq/refSeqs.json文件。这并不是jbrowse安装的不好,是因为安装完后样例放在sample_data目录下,可以吧sample_data/json/volvox下的内容拷贝到jbrowse安装目录下的data目录下(如果没有data可以手工建一个)
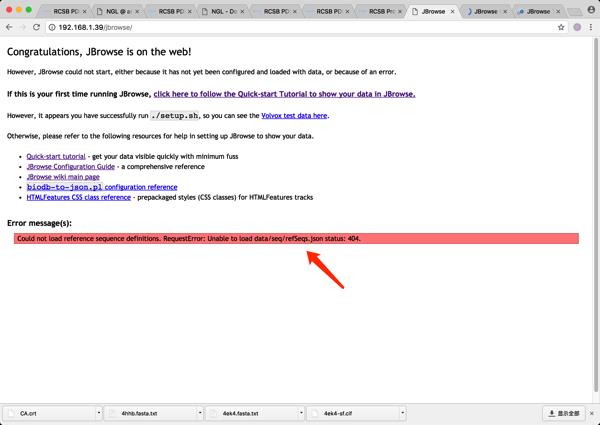
安装完成后会自带几个样例基因组,可以通过访问url来指定其他的data目录所在位置,例如样例提供的http://192.168.1.39/jbrowse/?data=sample_data/json/volvox
加上自己的基因组数据
虽然写这篇文章的时候把这步放在前面,但是之前走了很多弯路。jbrowse很多操作都是基于某个特定基因组的(也就是我们需要把我们的设置放在特定的trackList.conf,他只针对对应的data起作用)。
引用手册文件
Before any feature or image tracks can be displayed in JBrowse, the reference sequences must be defined using the prepare-refseqs.pl formatting tool.
上面安装步骤介绍的时候我们是吧volvox数据拷贝到了data下,这里简单说一下如何创建自己的data
参考官网文档http://gmod.org/wiki/JBrowse_Configuration_Guide#Reference_Sequences
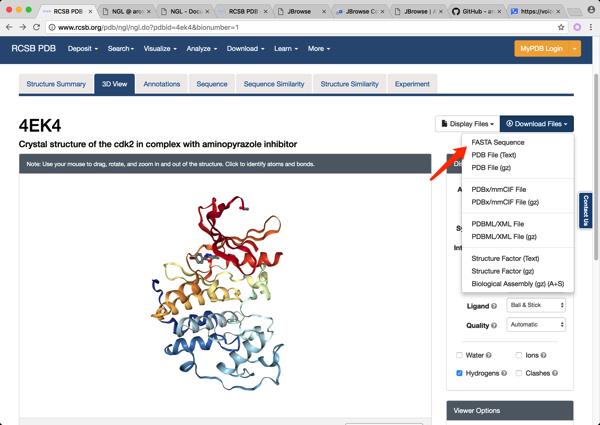
从RCSB网站下载FASTA文件,然后运行命令
bin/prepare-refseqs.pl --fasta [options]
如果不指定options的话就会在目录下生成一个4EK4对应的data目录
找到控制jbrowse的核心文件
所谓的核心文件也就是控制jbrowse的一些操作函数所在的js文件,文件位于{jbrowse_root}/src/JBrowse/Browser.js
例如我们要自己实现一个按钮,控制jbrowse的视窗右移,我们可以在代码中找到下面这一段
var moveRight = document.createElement("img");
//moveRight.type = "image";
moveRight.src = this.resolveUrl( "img/Empty.png" );
moveRight.id="moveRight";
moveRight.className = "icon nav";
navbox.appendChild(moveRight);
dojo.connect( moveRight, "click", this,
function(event) {
dojo.stopEvent(event);
this.view.slide(-0.9);
});
最重要的就是this.view.slide(-0.9)这句话了,我们在其他地方写js的时候只要能得到view这个对象,那么我们就可以直接调用他的slide方法进行平移操作了。
这个目前可以实现控制jbrowse上的navigation导航栏
玩一下plugin
玩jbrowse时候遇到的一些坑
在调试的时候发现改了代码没有起作用,首先要清除一下缓存,然后再刷新。例如在写插件的时候,发现view自带一个zoomOut,但是this.view.zoomOut报找不到view(this等于plugin当然找不到~),改为args.browser.view感觉应该能找到,但是怎么刷新都提示找不到view,后台清除缓存后解决此问题。
在写好了一个plugin的时候发现无论如何就加不上(后来发现这是一个连环坑),因为如果不提供url中data=sample_data/xxxxx/xxx那么jbrowse会去找根目录下的data目录,但是一开始setup.sh完成后并没有这个目录,所以造成在jbrowse.conf就算配置上plugin也看不到,但是如果加上url中的data,那么jbrowse.conf就不起作用了,直接定位到sample_data下的trackList.json。
NGL安装
同样
参考文档
jbrowse官网
http://jbrowse.org
官方配置向导
http://gmod.org/wiki/JBrowse_Configuration_Guide
参考了这个网站的全屏按钮的代码
https://phytozome.jgi.doe.gov/jbrowse/sample_data/
一个不错的jbrowse网站,介绍了一些官网没有的概念
https://www.araport.org/jbrowse/user-guide#navigation
以上是关于jbrowse和NGL展示基因组的主要内容,如果未能解决你的问题,请参考以下文章