生物信息学ATAC-seq流程及代码分析复现文章
Posted 爱做饭的电饭煲
tags:
篇首语:本文由小常识网(cha138.com)小编为大家整理,主要介绍了生物信息学ATAC-seq流程及代码分析复现文章相关的知识,希望对你有一定的参考价值。
1:ATAC-seq的定义及用途 what is

ATAC-seq 全称是 Assay for Transposase-Accessible Chromatin sequencing 即借助Tn5转座酶对开放染色质区域进行高通量测序
-
主要原理:
Tn5 转座酶可以对染色质开放区域DNA切割并添加测序接头,然后进行高通量测序就取得了开放染色质区域的测序数据。与其他技术比较(DNase-Seq, FAIRE-Seq) ATAC-seq 需要的细胞数目更少,同时实验步骤更简单耗时更少,高通量也是一个优点一次性取得了所有的开放染色质区域。 -
大致流程

2: ATAC-seq的用途
- ATAC-seq是一种用来检测染色质可及性的的测序方法,即确定基因表达调控机制的方法。它有以下常见用途:
-
1.帮助识别启动子区域、潜在的增强子或抑制子。
(1)启动子是靠近转录起始点(TSS)的DNA区域。它包含转录因子的结合位点,这些转录因子将招募RNA聚合酶。
(2)增强子是可位于启动子下游或上游1Mb的DNA区域。当 转录因子与增强子结合,并与启动子区域接触时,该基因的转录增加。相反,抑制子会减少或抑制基因的表达。 -
2.生成表观基因组图谱
-
3.得到在不同组织或不同条件下对应可及性区域。
-
4.得到核小体位置,鉴定重要转录因子
-
5.生成转录因子结合区域的特征(footprinting)
3:实操ATAC-seq(代码分析及复现文章) how to do
1.Data analysis
2.Quality control
3.Alignment and filter
4.Mitochondrial reads
5.PCR duplicates
6.Merging BAMs
峰的解析:
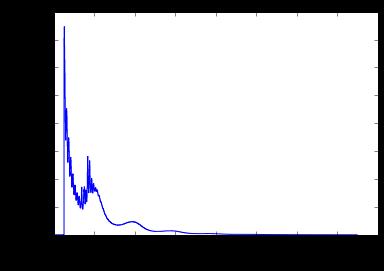
In a normal ATAC-seq library, you should expect to see a sharp peak at the <100 bp region (open chromatin), and a peak at ~200bp region (mono-nucleosome), and other larger peaks (multi-nucleosomes).
以上是关于生物信息学ATAC-seq流程及代码分析复现文章的主要内容,如果未能解决你的问题,请参考以下文章