可视化工具之 IGV 使用方法
Posted Digital-LI
tags:
篇首语:本文由小常识网(cha138.com)小编为大家整理,主要介绍了可视化工具之 IGV 使用方法相关的知识,希望对你有一定的参考价值。
整合基因组浏览器(IGV)是一种高性能的可视化工具,用来交互式地探索大型综合基因组数据。它支持各种数据类型,包括array-based的和下一代测序的数据和基因注释。IGV这个工具很牛,发了NB:
使用IGV的基本思路
安装和配置
要用linux上的IGV(首先需要去官网下载一个Linux下的IGV),然后需要一个远程控制工具:
这里使用 VNC,网上下载破解版,安装(输入注册码即可破解,文章底部有注册码)。
运行IGV
开启 linux 的 VNCserver,开启了之后,linux上会显示节点和端口,用于Windows端的连接。
vncserver -geometry 1366x768 #调节分辨率
开启服务后,Linux会提示可以登录的节点,登录VNC需要你的ip、端口及VNC密码。
运行 VNC 客户端(VNC viewer)里的 IGV.sh 开启可视化程序(打开Windows的VNC会自动跳转到Linux下的IGV安装目录,双击IGV.sh即可打开)。
1.载入参考基因组数据
可以点击选择一个,也可以通过菜单栏Genomes载入文件。
2.载入排序后的bam文件
通过菜单栏的File,load from file。。。
IGV可视化数据准备
不是什么数据都可以拿IGV看的,参考基因组必须为FASTA格式;
IGV只是负责将比对结果可视化,并没有比对过程,所以不能直接载入reads;
需要将待比对的reads与前面指定的参考基因组用bwa进行比对;
比对后的sam文件也不能直接载入(麻烦),要转bam;bam排序;bam建索引(可以一步完成);
命令流程如下:
BWA出来的sam文件无法直接被 IGV 利用的,必须经过 samtools 处理后才能被 IGV 显示出来。
samtools view -@ 10 -bS -F 4 ./contigs_sequence_align_to_public_genome.sam > ./contigs_sequence_align_to_public_genome.bam
samtools sort -@ 10 ./contigs_sequence_align_to_public_genome.bam contigs_sequence_align_to_public_genome.sorted
samtools index contigs_sequence_align_to_public_genome.sorted.bam contigs_sequence_align_to_public_genome.sorted.bai
samtools depth contigs_sequence_align_to_public_genome.sorted.bam >depth_reads.txt
wc -l depth_reads.txt > Coverage-aln_reads.txt
更多的使用细节参照 samtools 使用方法。
IGV 需要设置内存,在看大型基因组时很吃内存.
示例:
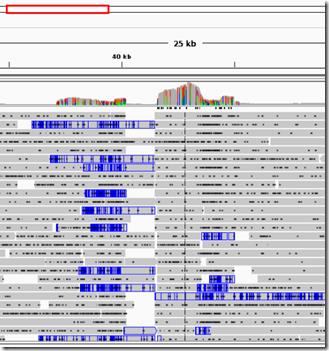
下面显示的是组装异常区,这是手动连接的地方,Pacbio全部不能顺利跨过去,说明组装有问题。
以下是单细胞转录组比对可视化:
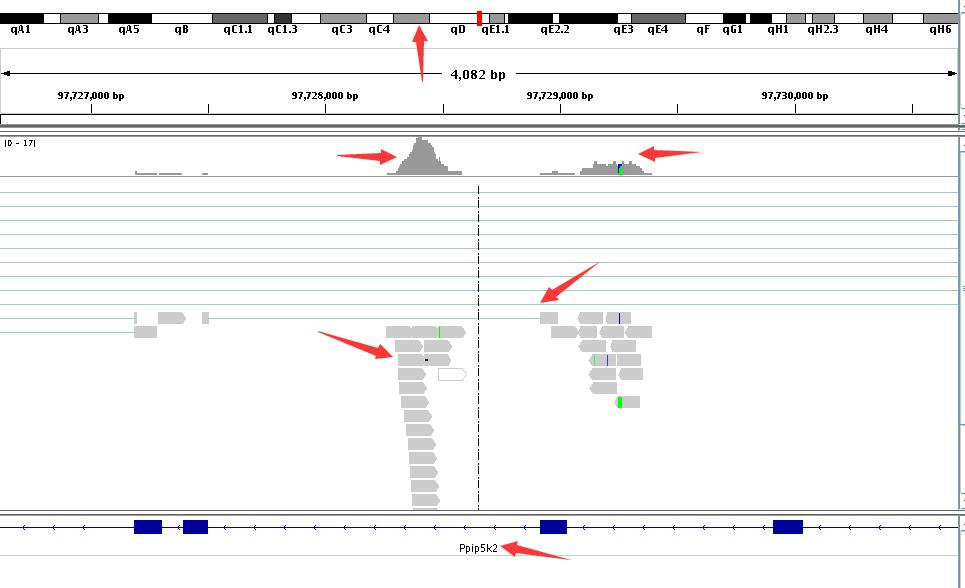
IGV基本操作
如何去掉右边自动弹出的框框?
蓝色框起来的reads是啥?
…
IGV用户指南
目录:
- 用户界面
- 开始使用IGV
- 导航
- 加载一个基因组
- 外部控制IGV
- 查看参考基因组
- 加载数据和属性
- 查看数据
- 查看比对(核心)
- 查看Variants
- 基因列表视图
- 感兴趣的区域
- 样本的属性
- 排序、分组和过滤
- 保存和恢复会话
- 服务器配置
- Motif查找器
- igvtools
- BLAT 搜索
1.用户界面
1.1 Main Window(主窗口)
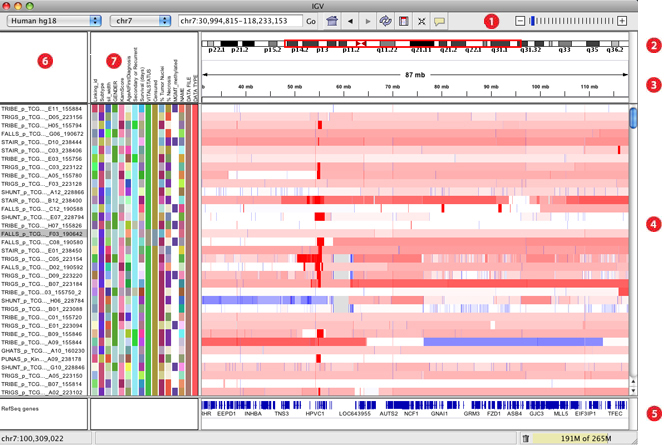
1.tool bar(工具栏),menu bar(菜单栏),pop-up menus(弹出式菜单)。
2.染色体上的红色盒子表示显示这部分染色体,显示完整染色体是红框会消失。
3.尺度显示了染色体的可见部分,刻度线显示了染色体的位置,跨度列表显示了当前显示的碱基的数量。
4.IGV在水平行显示的数据称为tracks。通常,每个tracks代表一个样本或实验。这个例子展示了甲基化、基因表达、拷贝数,LOH和突变数据。
5.IGV也显示某些特性,比如在tracks中的基因。默认情况下,IGV在一个面板显示数据,在另一个面板显示数据特性。拖放一个track名称,将一个track从一个面板移动到另一个地方。
6.Track名称列在最左边面板。名字的易读性取决于 tracks的高度,例如,track越小,它的名字的可读性越小。
7.属性名称被列在顶部的属性面板。彩色块代表属性值,每个独特的值被都有一个独特的颜色。鼠标放在一个颜色块的附近来查看其属性值。
参考资料
Viewing Alignments(查看比对,核心文档)
IGV软件使用指南(博客)
软件安装:
RealVNC是一款免费的远程控制程序,它是VNC (Virtual Network Computing)众多操作平台版本中的一员,这里小编提供的RealVNC适用于windows操作平台,软件采用2048 位 RSA 服务器验证和 128 位 AES 会话加密术,安装此软件的电脑可在全球各地远程操控自己的电脑,所操作平台没有任何限制。
功能特色
1.Windows 验证 —— 允许访问 VNC 服务器以便控制基于标准的 Windows 用户证书, 无论其是本地的、NT 域或者基于 Active Directory。
2.单一登录 —— 允许在一个域内部访问 VNC 服务器而不需要重新输入用户的名称和口令。
3.简单但有效的部署 —— 用 VNC Tool,即 VNC Deployment Tool for Windows 安装和 管理 VNC Enterprise Edition 服务器。
4.桌面缩放 —— 通过精确的比例缩放,或动态缩放为任何大小。
5.Windows 防火墙集成 —— 使 VNC Server 可以更直接的部署。
6.文件传输 —— 与 Windows 剪贴板结合,允许通过 VNC 连接在服务器和查看器计算机之间 复制文件而不需要额外的配置。
最好的就是,与 VNC Enterprise 系列的其他产品一样,VNC Enterprise Edition for Windows是由原始 VNC team 设计、开发和维护的。
RealVNC注册码
3YHED-MNEHC-RMJT5-4UAAK-6A5HA
FBV9V-7Z3V9-MED3U-47SEU-85T3A
HA7MG-J8R3J-R3528-HC6P5-HTQ6A
B7SLM-7MAX5-B4M74-UTDBE-K5WFA
7736G-H4YBP-6ZCEU-AM3H4-QBTWA
NUQQ6-UP89V-9XGPU-GUYU9-U6VTA
以上是关于可视化工具之 IGV 使用方法的主要内容,如果未能解决你的问题,请参考以下文章