疑问1
Posted mohuishou-love
tags:
篇首语:本文由小常识网(cha138.com)小编为大家整理,主要介绍了疑问1相关的知识,希望对你有一定的参考价值。
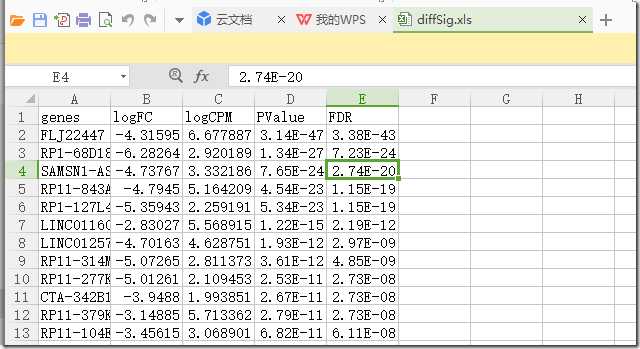
开题报告:筛选同时符合P值<0.05及FoldChange>2的lncRNA数据
程序里:diffSig = diff[(diff$FDR < padj & (diff$logFC>foldChange | diff$logFC<(-foldChange))),]#筛选有显著差异的#
foldChange=1;
diff$logFC 意思
https://blog.csdn.net/tuanzide5233/article/details/88785486
生物信息学入门 使用 RNAseq counts数据进行差异表达分析(DEG)——edgeR 算法 数据 代码 结果解读
这篇博客下面的几个链接看下
edgeR-trainresult.csv datExpr
allTraits.csv datTraits
save(datExpr, datTraits, file = "WGCNA01.RData")
把一些可调参数弄懂它,没条语句什么意思大概搞懂,
训练集和测试集
1. 概念
我们训练机器学习模型,目的是使用已有数据来预测未知的数据,通常我们称模型对未知数据的预测能力称为泛化能力。为了评估一个模型的泛化能力,通常我们会将数据分成训练集和测试集,训练集用来训练模型,测试集用来评估模型的泛化能力。
WGCNA 是不是不需要分训练集和测试集 ,那个赵志洪的没分吧。 那个中文指南是不是也没分?
cox回归没有训练集合和测试集
下面的对这个问题找了几个文献和博客
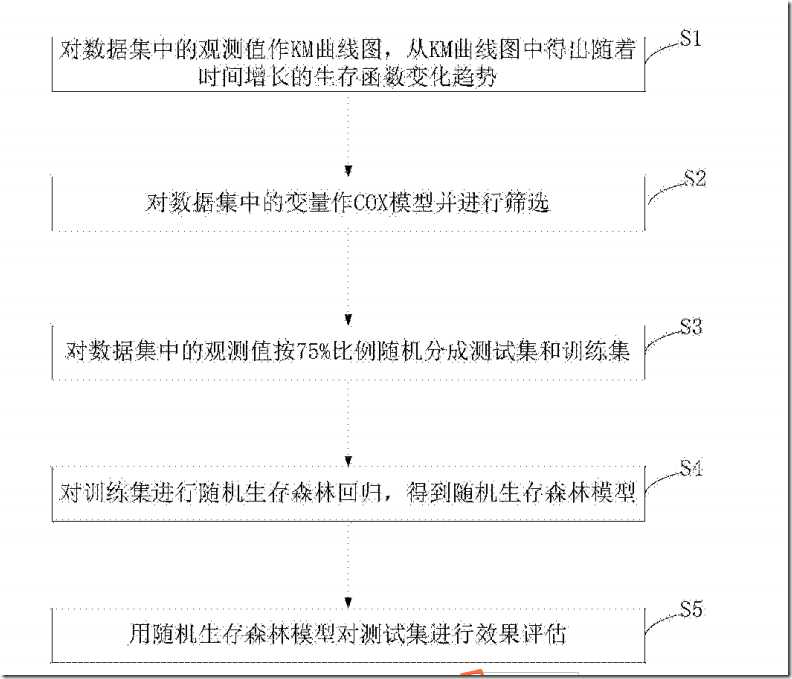
http://www.xjishu.com/zhuanli/55/201610797710.html
一种基于COX模型和随机生存森林的数据预测分析方法与流程 这是个专利,我下载下来了,下面是个流程图
https://wenku.baidu.com/view/5ecdb1728f9951e79b89680203d8ce2f00666593.html
这个文库里面coxph 虽然这里分了训练集和测试集,但是在下面没用。
在下面对测试集和训练集做了ROC分析?好像是。
你给我发的
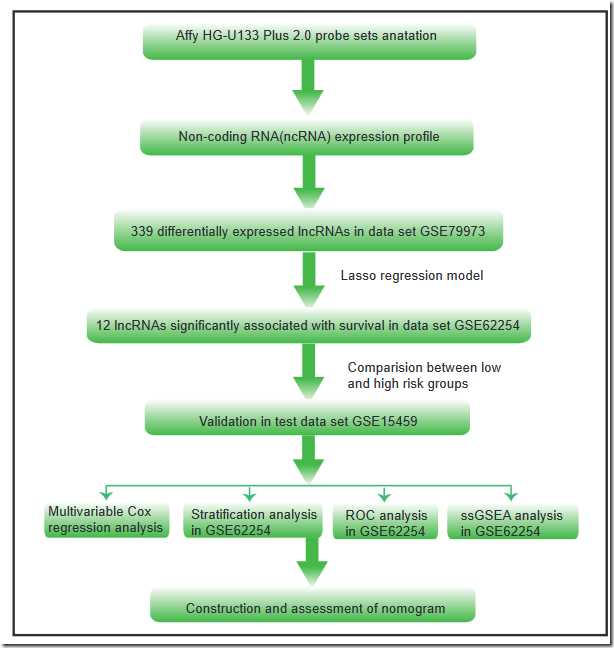
lasso-cox回归,文章里有这个,
Supplementary Material
Supplementary figures.
http://www.jcancer.org/v08p2575s1.pdf
以上是关于疑问1的主要内容,如果未能解决你的问题,请参考以下文章