promoter, exon, intron和UTR的关系
Posted
tags:
篇首语:本文由小常识网(cha138.com)小编为大家整理,主要介绍了promoter, exon, intron和UTR的关系相关的知识,希望对你有一定的参考价值。
参考技术A 启动子 :RNA聚合酶特异性识别和结合的DNA序列。promoter自然不属于intron和Exon的任何一个,属于noncodingsequence。
(1) 启动子位于转录起始位点TSS上游,没有明确的长度范围,但是有几个保守序列 :启动子是RNA 聚合酶识别、结合和开始转录的一段DNA 序列,它含有RNA 聚合酶特异性结合和转录起始所需的保守序列,多数位于结构基因转录起始点的上游,启动子本身不被转录。但有一些启动子(如tRNA启动子)位于转录起始点的下游,这些DNA序列可以被转录。启动子一般位于转录起始位点的上游。
(2) 启动子区中的保守序列 :在真核生物基因中,①Hogness等先在 珠蛋白基因 中发现了类似Pribrow区的Hogness区(Hogness box),这是位于转录起始点上游 -25 ~~ -30 bp 处的共同序列似TAAA,也称为TATA区。②另外,在起始位点上游 -70 ~~ -78 bp 处还育另一段共同序列CCAAT,这是与 原核生物 中-35bp区相对应的序列.称为CAAT区(CAAT box)。
noncoding RNA是现在研究的热点之一。我们常见的MiRNA,SiRNA,antisense RNA tech,这些都是属于ncRNA的范围。只要你在进一步问下:这些RNA是哪里来的?你就知道部分答案,跟那些看似跟编码蛋白没有关系的DNA序列有关系。这部分DNA有个统称就junk DNA,垃圾DNA或者冗余DNA,他们编码的RNA就属于 ncRNA.RNAi就是迄今最经典的ncRNA功能典范。
An exon is asequence of DNA that is expressed (transcribed) into RNA and then often, butwith many noteworthy exceptions[1] , translated into protein. Adjacent exons maybe separated by an intron, which is later removed from the RNAtranscript via the splicing mechanism. (From Wikipedia)
UTR(UntranslatedRegions)即非翻译区,是信使RNA(mRNA)分子两端的非编码片段。
5-UTR从mRNA起点的甲基化鸟嘌呤核苷酸帽延伸至AUG起始密码子,3-UTR从编码区末端的终止密码子延伸至多聚A尾巴(Poly-A)的末端。
转录(Transcription)是遗传信息从DNA到RNA的转移。即以双链DNA中的一条链为模板,以ATP、CTP、GTP和UTP4种核苷三磷酸为原料,在RNA聚合酶催化下合成RNA的过程。
常见问题:
问:Promoter在DNA序列中是算内含子还是外显子?
答:都不是。属于noncoding sequence。
问:UTR在DNA序列中是算内含子还是外显子?
答:外显子。
问:起始密码子ATG在gene的哪个位置?
答:外显子内,CDS以ATG开始。
问:每个基因之间都有一定的间隔序列,这些基因间的间隔序列应该不属于内含子吧?
答:是的。属于noncoding sequence。
问:如何寻找启动子区域和预测转录因子结合位点?
答:见帖子 http://www.dxy.cn/bbs/topic/23466429
Novel LncRNA的实时定量PCR引物设计教程
Novel LncRNA的选择
novel LncRNA选择的原则:
1.不能选与其他基因exon重叠的lncRNA,否则,RT-qPCR结果就混合了两个基因的表达量。
2.最好选择基因间的lncRNA,即lincRNA,跟其他基因没有重叠。
3.其次选择位于其他基因intron区域的lncRNA。
4.再次,如果lncRNA的一部分与其他基因的exon重叠,另一部分不重叠,就选不重叠的那部分。
Novel LncRNA的引物设计
这里我们选取的novel LncRNA是LNC_000332,LNC_000332的fasta序列如下:
>LNC_000332:
TGGCAGCTTCAAGAACGAGTGATTTAAGAACAGCCCTCCCATCTTAGCAATGTCCCGGGGTGGCTGGAGCCACGGTCACTTCTTGGTCCTGGTCCAGAACTGTCGGTAGCGCTCCACATGCAAGTCATCACTGAGCTCCTGCTCGTACTCCTTCCTCGACTGGAGCTGAGCCTCACGCTTCCGCAGCCTGGCCCGGCGCTCCTCAGGAGACAGGGTGTCCAGATCTATGGGCATCGCCAGCATGCGCCGTGGCTCCCTGTAGCGGATGTGGATGGTGGAGCCATCCTGCTTCACCAGCAGCACGGGGTAGAGTCGTGCATAAGCCTGGCGGTGCACACGAGTGAGTGAGGCCCTGCTGCTGTCAGCTCGCCAGGAGGATGTGTGCAGGCGGCGGAGTGCAGGTCCGGTGGCCTTCACGGTGCTCTGCCTCAGCCGGCCAAGCAGGCTGCCCACGGCCGCCATTCCTCCTTACAGCCCCAGTGGCACGTGGAGGTGGTCAGTACAGGCCCTGGCGTGCAACTGCGTTGGGTGCTGCTGCGCTGCAGCCCACGACGTCACTGGCAGGCGCTGCGTCGCGCGGTATGACGTGTTCCATGGC
根据LncRNA的染色体位置相应位置的DNA序列
LNC_000332的染色体位置为:chr1:228106685-228109338,在染色体的负链上,我们通过UCSC来获取相应位置的DNA序列。UCSC的官网http://genome.ucsc.edu
然后选择Genomes,进入后点击View,然后选择DNA
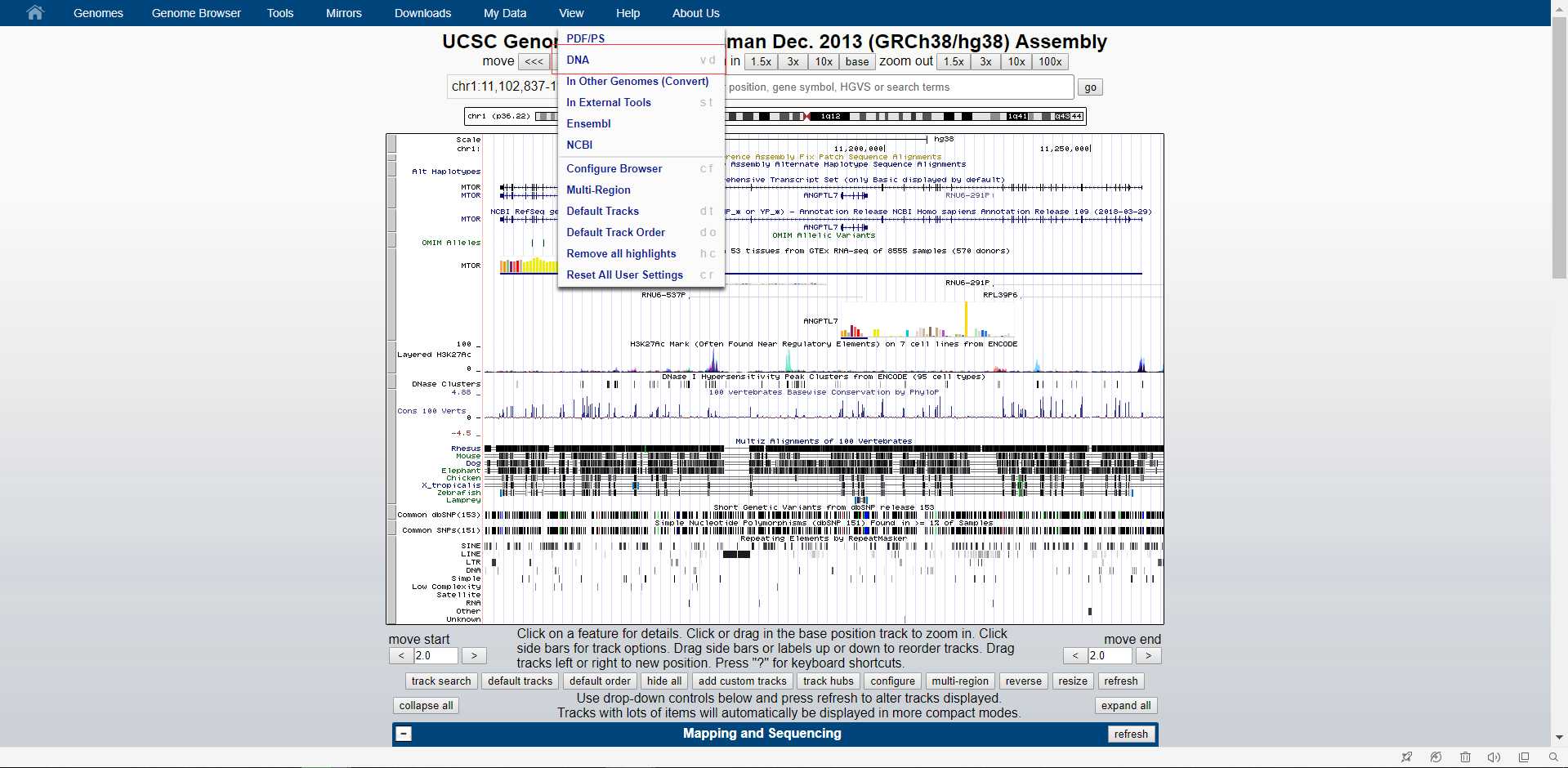
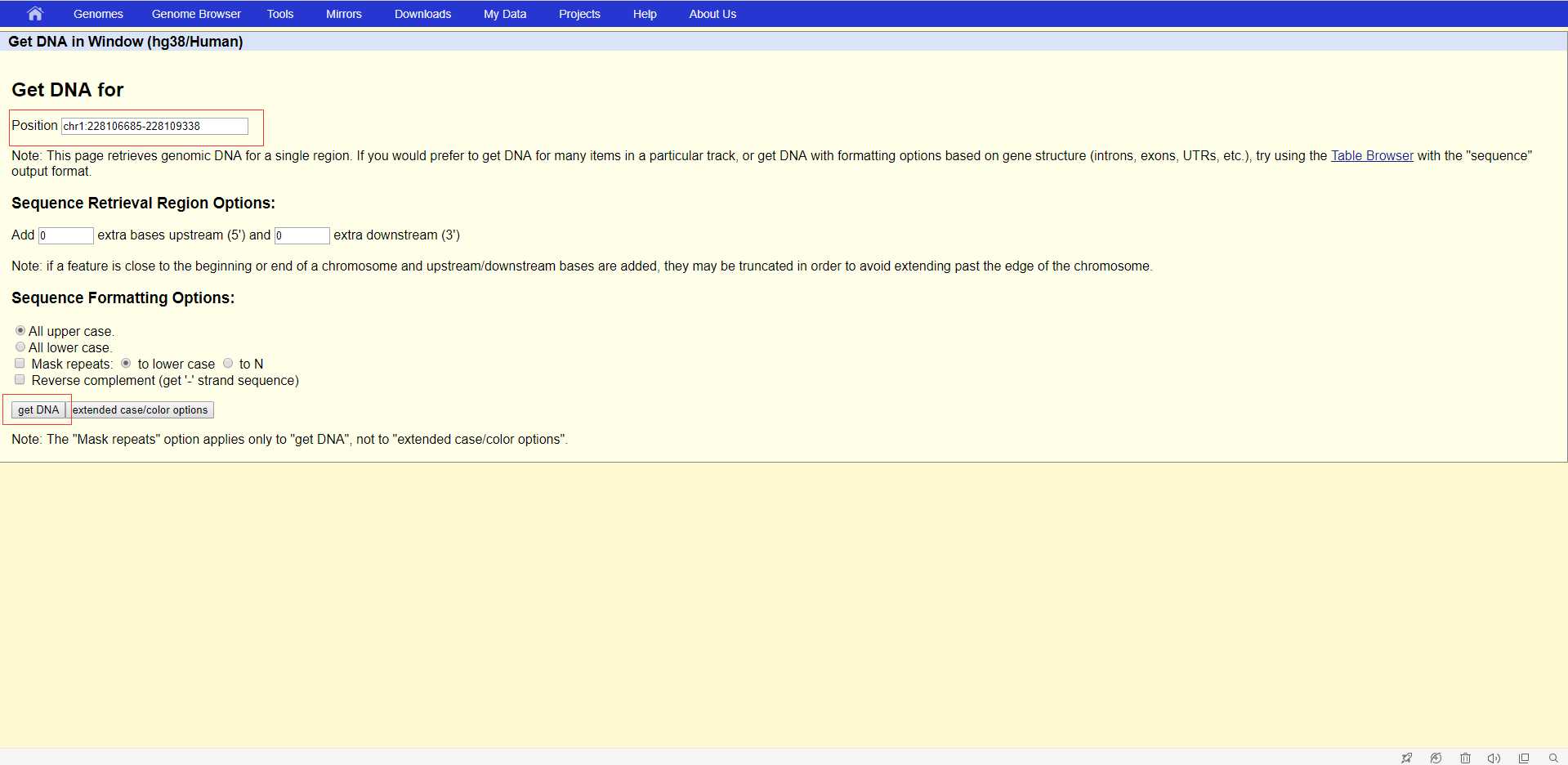
然后在上面的position输入基因的坐标,由于我们的坐标在DNA的负链,所以这里也要勾选Reverse complement,最后点击get DNA,就得到我们想要的DNA序列,下图为得到的DNA序列

通过AnnoLnc获取novel LncRNA的外显子坐标
AnnoLnc是北大生物信息中心开发出的一款分析novel LncRNA的网页工具,网址:http://annolnc.cbi.pku.edu.cn/index.jsp
我们输入LNC_000332的fasta序列,然后点击GO
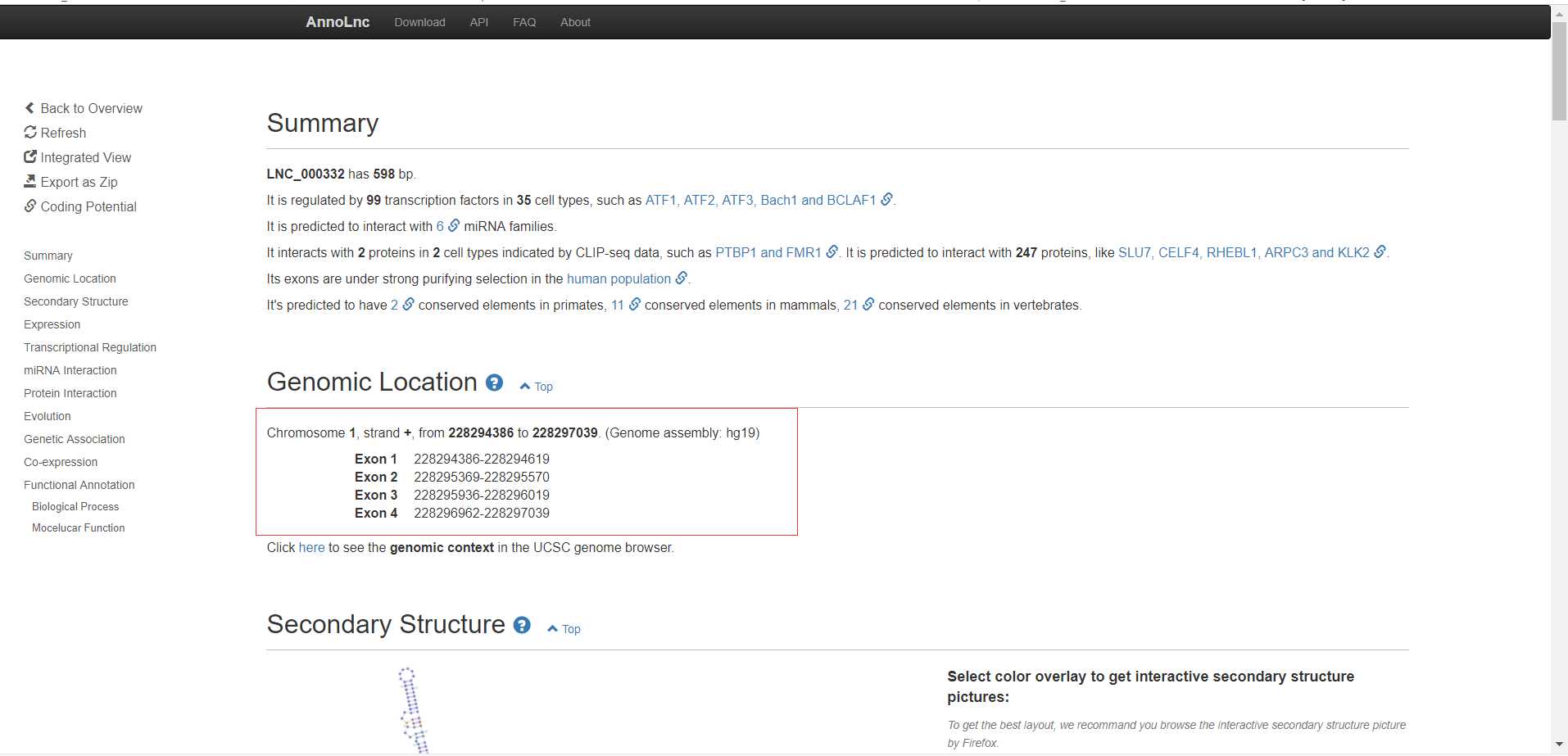
信息很多,这里只截取一部分,可以看到LNC_000332包含4个外显子区域,不过这是hg19的染色体位置,我们需要用网页版的LiftOver来进行hg19和hg38的坐标转换,网址:http://genome.ucsc.edu/cgi-bin/hgLiftOver
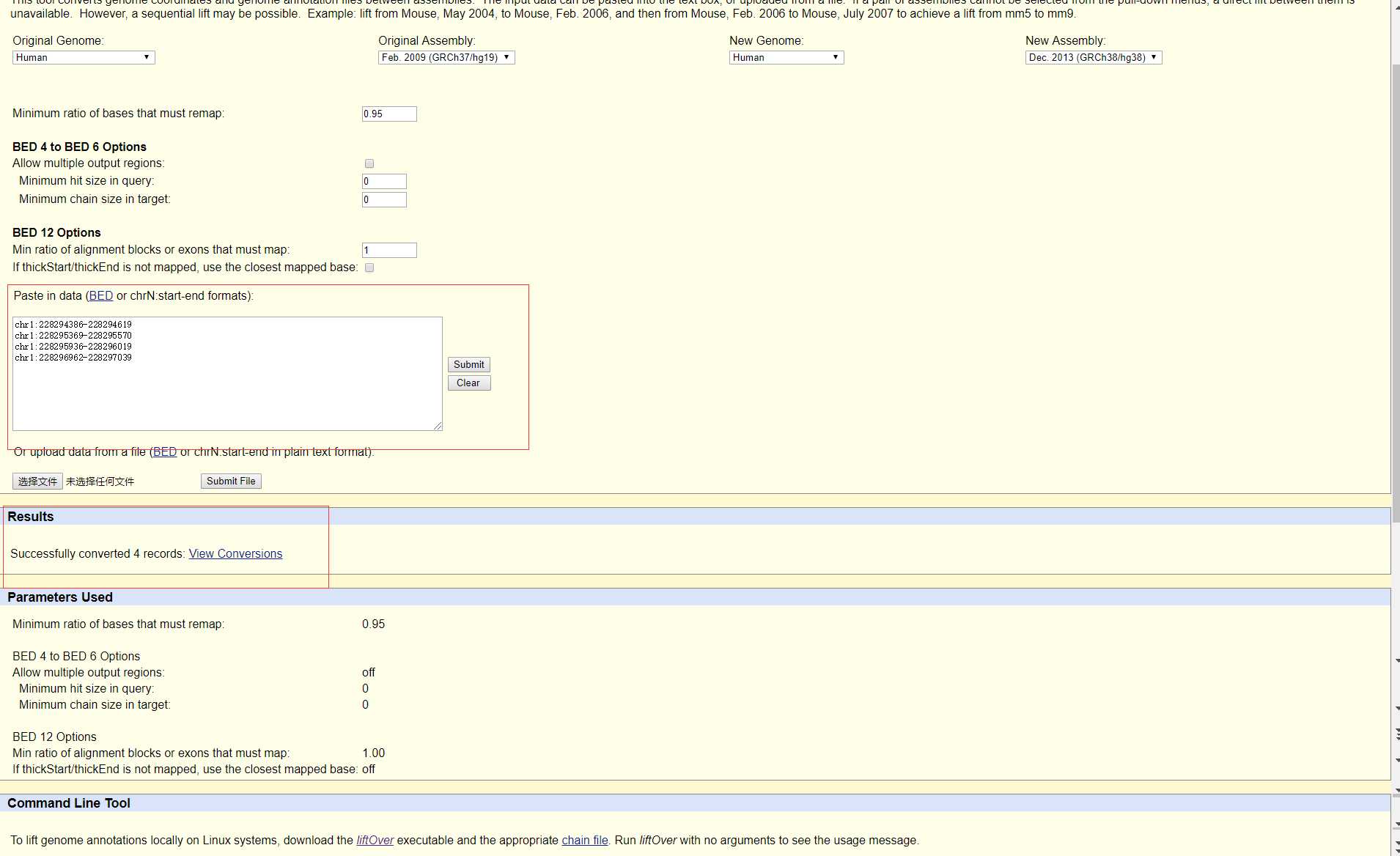
可以看到4个外显子坐标成功转换完成,点击View conversions就可以得到转换完成的hg38的外显子坐标,外显子坐标为:
chr1:228106685-228106918
chr1:228107668-228107869
chr1:228108235-228108318
chr1:228109261-228109338
将相应位置的DNA序列与LncRNA序列比对
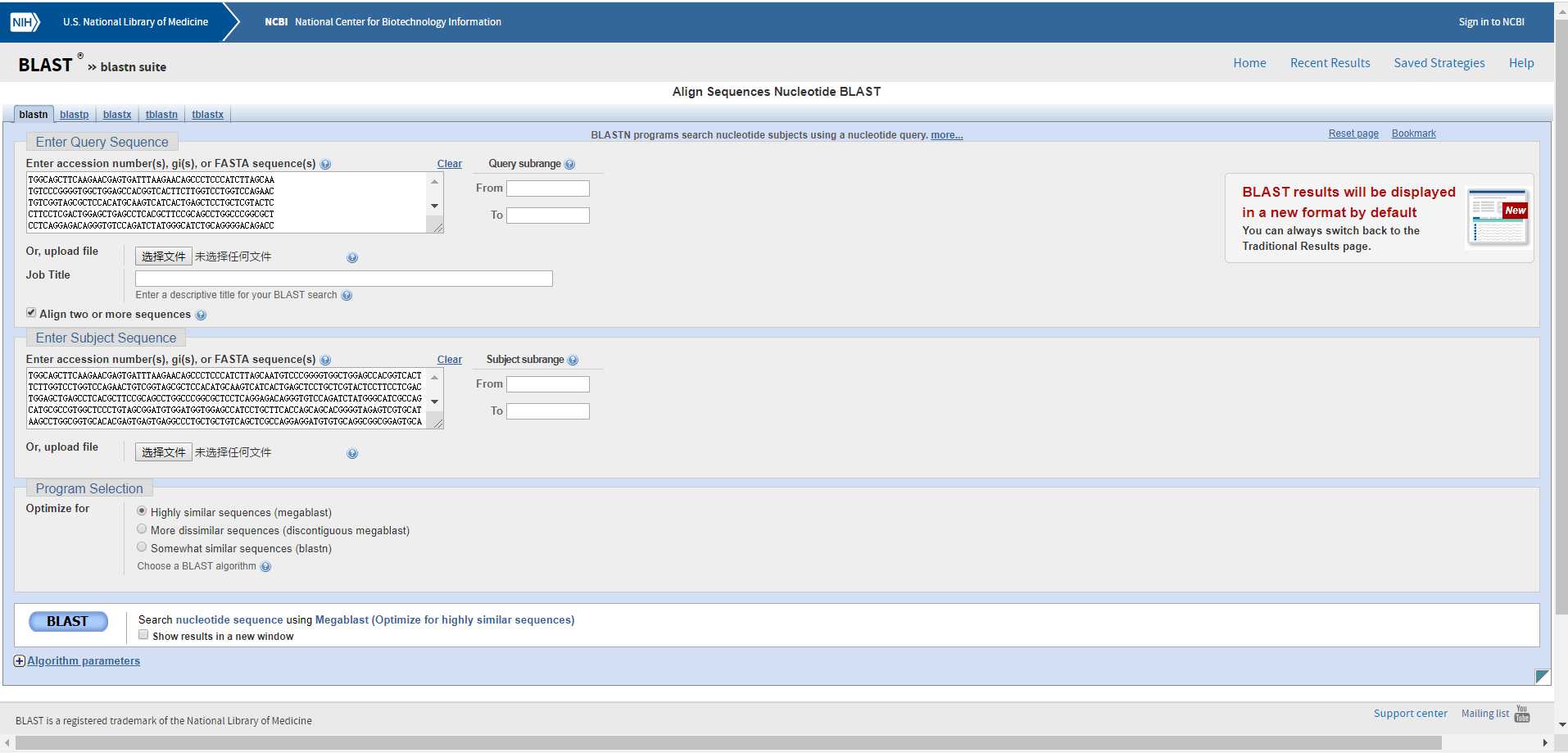
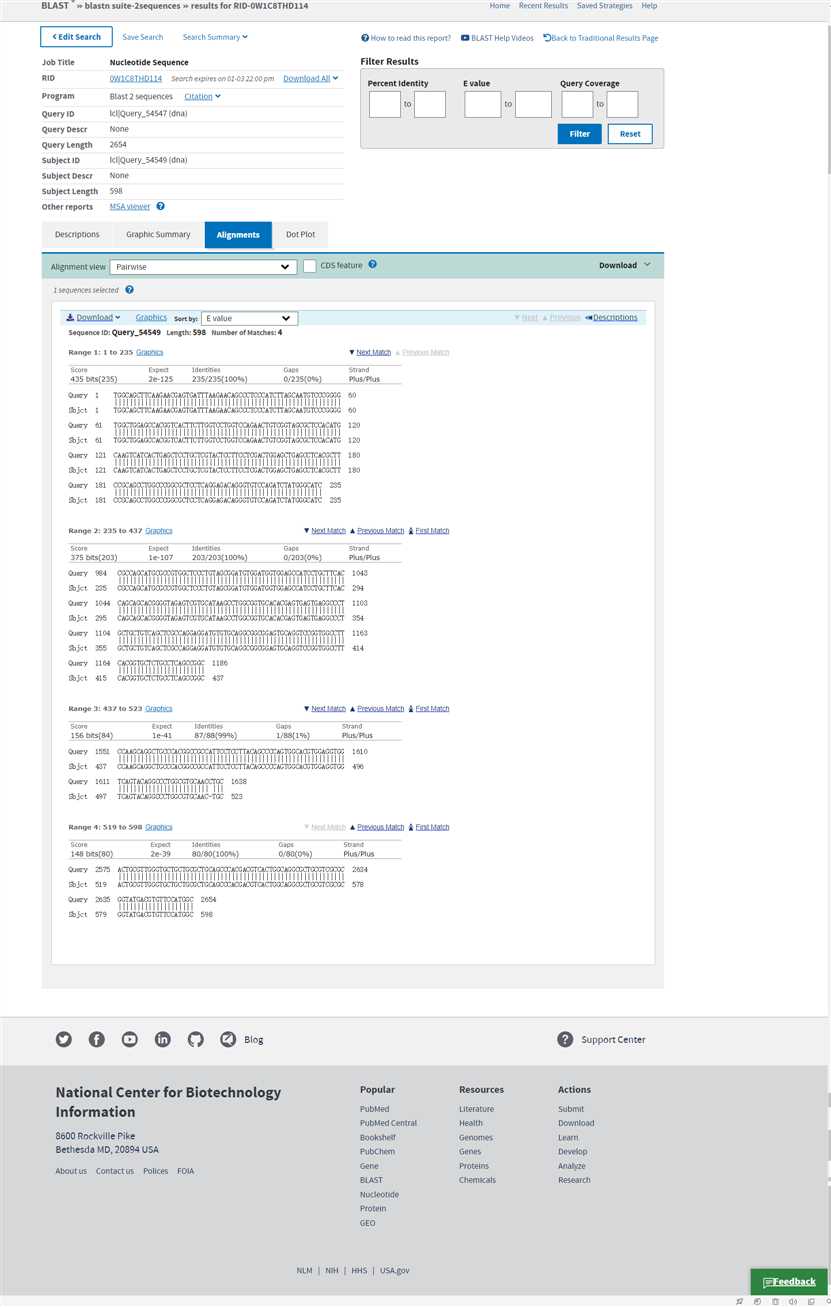
可以看到LNC_000332一共匹配了4段,染色体坐标分别为chr1:228106685-228106919,chr1:228107668-228107870,chr1:228108235-228108332,chr1:228109259-228109938
而外显子的染色体坐标分别为chr1:228106685-228106918,chr1:228107668-228107869,chr1:228108235-228108318,chr1:228109261-228109338
这里可以看到LNC_000332的序列基本都在外显子里面,所以不适合选择这个LncRNA来做PCR验证
以上是关于promoter, exon, intron和UTR的关系的主要内容,如果未能解决你的问题,请参考以下文章