实用技能如何从SRA数据库下载二代测序数据?
Posted 上海交大系统生物医学研究院
tags:
篇首语:本文由小常识网(cha138.com)小编为大家整理,主要介绍了实用技能如何从SRA数据库下载二代测序数据?相关的知识,希望对你有一定的参考价值。
欢迎关注 上海交大系统生物医学研究院
很多课题组有进行二代测序的需求,但苦于经费问题最后都退而求其次选择从数据库中挖掘已有数据,今天我们就来讲一讲如何从SRA数据库下载测序原始数据。

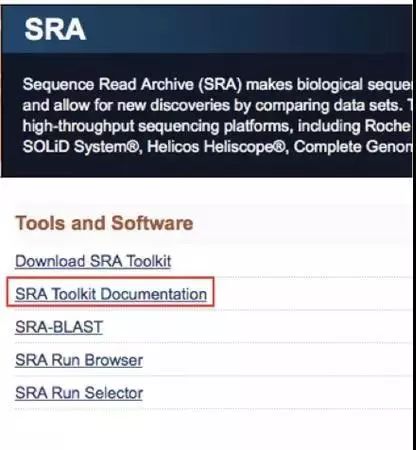
1、登入NCBI的SRA数据库
https://www.ncbi.nlm.nih.gov/sra/点选SRA Toolkit Documentation。
2、在SRA Toolkit Documentation页面点选SRA Toolkit Installation and Configuration Guide。

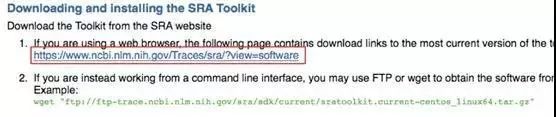
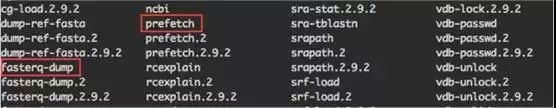
3、通过链接或命令行方式下载SRA Toolkit。解压后可以看到里面有一个bin文件夹,该文件夹中存有各种测序下载和数据格式转换工具。此处展示了bin文件夹中的部分工具,红框中的prefetch和fasterq-dump工具稍后会用到,prefetch用来下载数据,fasterq-dump将数据转换为fastq格式,方便后续分析。
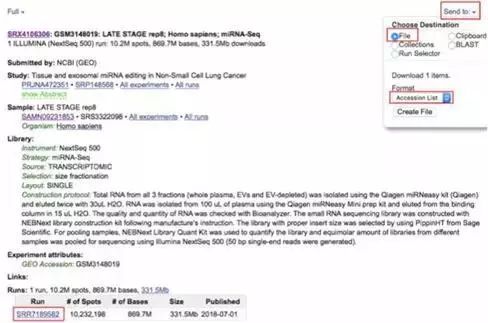
4、载工具准备完毕,现在可以来搜索一下感兴趣的研究内容。此处我挑选了一个人类肺癌样本的小RNA测序数据SRR7189582。

5、在测序信息页面点击右上角Send to -> File -> Accession List之后创建文件,会生成并下载一个名为SraAccList.txt的文本文件,里面包含的信息就是左下角红框中的SRR开头的编号,把这个文件保存在你想要稍后用来存放测序数据的路径下。
6)打开命令行界面,在存储有SraAccList.txt文件的路径下调用SRA Toolkit中的prefetch命令按照下图中设置参数下载数据。数据下载需要一定的时间,下载过程中没有进度提示,下载成功后会提示成功
7)下载完成后,调用fastq-dump命令处理下载好的数据,参数设置如图示。格式转换过程较快,处理完成后会在现有路径下找到文件fastq格式(FASTQ是一种存储了生物序列(通常是核酸序列)以及相应的质量评价的文本格式)的测序数据文件。
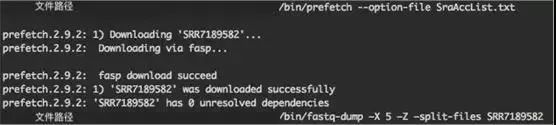
到这里为止,我们就成功的从SRA数据库下载到了一个测序数据。小伙伴们可以赶快尝试起来。有兴趣的同学还可以用fastqc等质控软件分析一下下载数据的质量,做些初步的处理,帮助后续的分析和挖掘。
信息来源:转载实验万事屋
信息采集:王华瑶 系统小Q
图文编辑:王华瑶 系统小Q
以上是关于实用技能如何从SRA数据库下载二代测序数据?的主要内容,如果未能解决你的问题,请参考以下文章