NCBI SRA数据库
Posted yahengwang
tags:
篇首语:本文由小常识网(cha138.com)小编为大家整理,主要介绍了NCBI SRA数据库相关的知识,希望对你有一定的参考价值。
简介
SRA数据库是美国国立卫生研究院(NIH)的高通量测序数据的主要归档,是国际核苷酸序列数据库协作(INSDC)的一部分,其中包括NCBI序列读取存档(SRA),欧洲生物信息学研究所(EBI)和DNA数据库 日本(DDBJ)。 提交给三个组织中的任何一个的数据都是共享的。
SRA数据库数据来自高通量测序平台(Roche 454 GSSystem®,Illumina GenomeAnalyzer®,Applied Biosystems SOLiDSystem®,HelicosHeliscope®,CompleteGenomics®和Pacific BiosciencesSMRT®)的原始测序数据和比对信息,储存的测序数据在研究团体间可重复使用,并通过比较数据集来实现新发现。
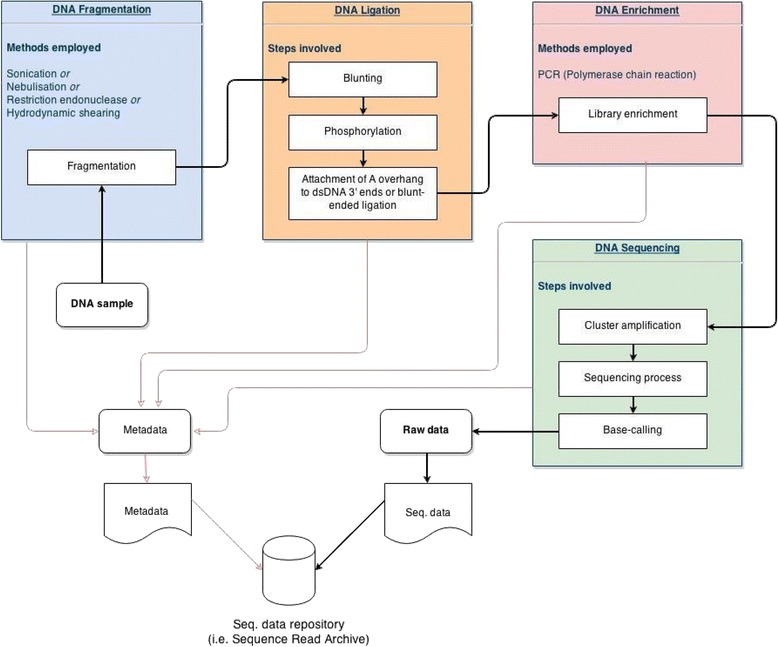
典型的下一代测序工作流程
SRA数据库和NCBI其它的数据库
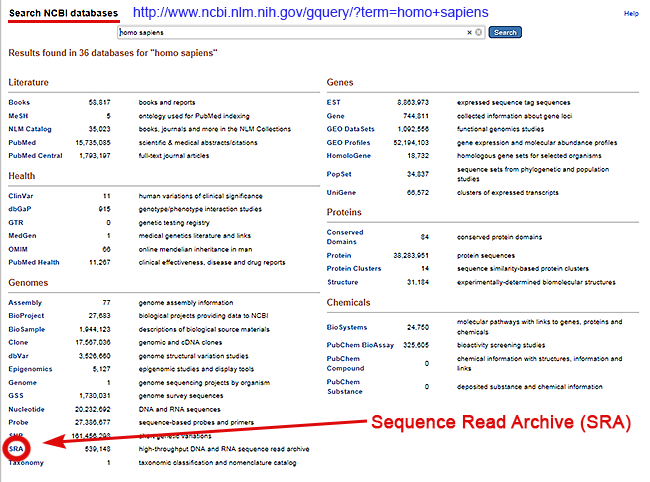
NCBI开发和维护了35个以上生物数据类别数据库,包括科学文献、健康、基因组、基因、蛋白质和化学品六个大类。
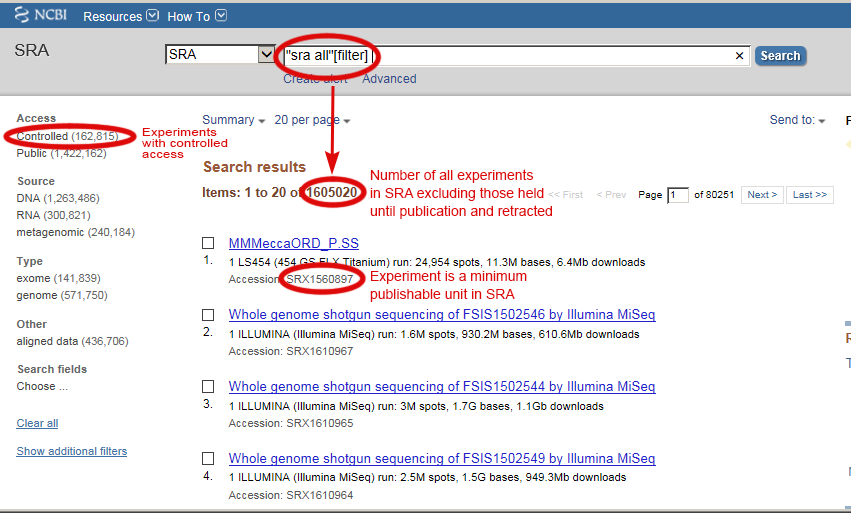
每个数据库都有自己的最小可发布单元。 例如,PubMed的最小可发布单位是一篇文章,而在SRA中,最小可发布单位是一个实验(以SRX#的形式登录)。 SRA实验包括序列数据和有关生物样品如何测序的(元数据)。
SRA数据库与其它数据库的交互
所有NCBI数据库都是互连的。 这种相互关联可以实现强大的搜索功能。 例如:
查找PubMed中引用SRA研究的文章: “pubmed sra”[Filter]
查找发表在PubMed的SRA实验:“sra pubmed”[Filter]
同样,您可以找到与其他NCBI数据库的SRA连接,反之亦然。
SRA数据
SRA接受来自各种测序项目的数据,包括涉及人类受试者或其基因组的临床重要研究,其可能含有人类序列。 这些数据通常通过dbGaP(基因型和表型数据库)进行受控访问。
SRA数据下载
1.SRA Toolkit工具下载
2.下载数据
首先去NCBI里面搜索并找到你想要的数据的SRA地址,然后写脚本批量下载。
$ while read line ; do wget $line; done<sra_ftp.txt
然后解压*.sra文件
$ for i in $(ls *.sra) ;do echo $i ; fastq-dump –split-3 $i ; done
查看fastq文件
【参考文献】
以上是关于NCBI SRA数据库的主要内容,如果未能解决你的问题,请参考以下文章