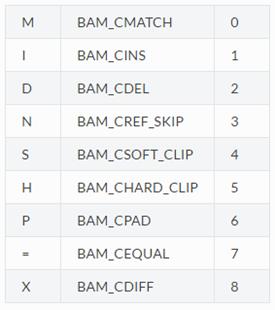
sam文件转换为bam文件——SAMtools
Posted
tags:
篇首语:本文由小常识网(cha138.com)小编为大家整理,主要介绍了sam文件转换为bam文件——SAMtools相关的知识,希望对你有一定的参考价值。
参考技术A 在转录组数据与参考基因组进行比对后,得到sam文件,后续分析需要将sam转换为bam,这里用到的工具是SAMtools。序列比对 —— Hisat2 - (jianshu.com)
SAMtools的主要功能是读取、写出、编辑、查看SAM/BAM/CRAM格式的数据文件。
SAMtools官方网站:
http://www.htslib.org/
链接:
http://www.htslib.org/download/
安装:
这里看到基础命令主要分为六个模块,分别为索引、编辑、文件操作、统计、视图以及其他。这里想要SAM格式转为BAM格式,主要用到的是Viewing模块中的view。
说明书:
http://www.htslib.org/workflow/fastq.html
在老版本1.9的samtools中,需要用-s 指定sam文件,1.14中不需要指定。
samtools view命令完成sam转为bam。
-@8:8个线程
-b:输出格式bam文件 >:输出文件名
pysam读取bam files
pysam - 多种格式基因组数据(sam/bam/vcf/bcf/cram/…)读写与处理模块(python)
在开发基因组相关流程或工具时,经常需要读取、处理和创建bam、vcf、bcf 文件。目前已经有一些主流的处理此类格式文件的工具,如samtools、picard、vcftools、bcftools,但此类工具集成的大多是标 准功能,在编程时如果直接调用的话往往显得不够灵活。
本文介绍的是一个处理基因组数据的python模块,它打包了htslib-1.3、samtools-1.3 和 bcftools-1.3的核心功能,能在编程时非常灵活的处理bam和bcf文件。
以下主要介绍pysam的安装和使用方法:
1. 安装
如果Linux上安装了pip,可以一键安装,在集群上的话,需要登录安装节点进行安装。
import pysam
2.读取bam文件(pysam.AlignmentFile)
bam是sam的二进制文件,因其占用空间少,所以都会使用bam进行存储和操作。
要读取bam文件,必须先创建一个AlignmentFile对象.
for line in samfile:
print(line)
break
但顺序读取还不够灵活,我们有时需要随机读取(提示:sam不能随机读取),pysam的fetch方法提供了随机读取功能.
直接使用fetch会报错
samtools index corrected.bam
fetch返回的是一个迭代器(iterator),可以迭代读取内容.
fetch(self, reference=None, start=None, end=None, region=None, tid=None, until_eof=False, multiple_iterators=False)
3.读取vcf/bcf文件(pysam.VariantFile)
读取方法同上,只是使用的是VariantFile方法:
bgzip -c MHC.g.vcf > MHC.g.vcf.gz
然后用bcftools建立索引
for rec in vcf_in.fetch(\'chr6\', 28577796, 28577896):
... print(rec)
... break
3.随机读取fasta文件(faidx建立索引)
读取方法略有不同,fetch返回的本身就是一个字符串。
fasta_file = pysam.FastaFile(path)
fasta_file.fetch("m160727_060737_42266_c101014182550000001823222610211695_s1_p0/110008/22268_22731")
4.创建并写入到新的bam或vcf文件
pysam的核心功能是可以随心所欲的读取数据,处理之后,写入到一个新建的bam或bcf文件里.
我们可以完全自定义一些内容,然后写入到一个新的bam文件里,如下:
同理,我们也可以读取一个已有的bam文件,逐个修改以上的属性,然后存储到一个新的bam文件里.这里不再举例.
上面设置header可能有点麻烦,容易出错,但我们可以复制一个已有bam文件的header到一个新的bam文件里.
outf = pysam.AlignmentFile(path_out, "wb", template=samfile)
以上template参数指定了模板bam文件.
5. 关闭文件
outf.close()
总结:
pysam模块非常实用,有了pysam模块,我们就可以非常灵活的操纵bam/bcf文件,而不必依赖于samtools或bcftools. pysam可以随机读取bam/bcf文件,也可以将处理后的内容自定义输出到bam/bcf文件.
以上只介绍了pysam最常见的功能,更多pysam功能请参照:http://pysam.readthedocs.io/en/latest/index.html
以上是关于sam文件转换为bam文件——SAMtools的主要内容,如果未能解决你的问题,请参考以下文章