DL笔记|使用卷积神经网络构建转录因子结合位点预测模型(deepbind)
Posted 组学时代
tags:
篇首语:本文由小常识网(cha138.com)小编为大家整理,主要介绍了DL笔记|使用卷积神经网络构建转录因子结合位点预测模型(deepbind)相关的知识,希望对你有一定的参考价值。
卷积神经网络(convolutional neural networks,CNN)是一类包含卷积计算且具有深度结构的前馈神经网络,是深度学习领域重要的算法之一,目前主要用于计算机视觉领域。2015年,多伦多大学首次使用卷积神经网络构建了转录因子结合位点的预测模型-deepbind,自此深度学习开始迈入基因组学领域。
由于deepbind是使用深度学习(CNN)用于基因组学最早的模型,因此了解deepbind的建模过程十分有必要。本文将尽可能详尽的描绘deepbind如何建模以及模型如何优化,可供各位同仁今后参考。
一、构建前馈神经网络
与常见的CNN模型相似,deepbind模型也包含最基本的四个步骤:卷积、非线性转化、池化、全连接网络。对于构建好的模型而言,输入一条包含ATCG的序列,会返回一个标量值,值越高,说明序列是该种转录因子结合序列的可能性越高。
1.卷积
给定一条DNA序列s=(s(1),...,s(n))和一个长度为m(模型中使用length=101bp)的卷积filterM,第一步是将DNA序列按下列公式进行转换,转换成一个维度(n+2m-2)×d的数组S。
假设序列s=ATGG,卷积fileter长度为3,则S为

设定卷积filter的个数为d,每个filter的维度为m×4。因此对于一条DNA序列而言,使用d个filter,卷积求内积后得到维度(n+m-1)×d的数组X。
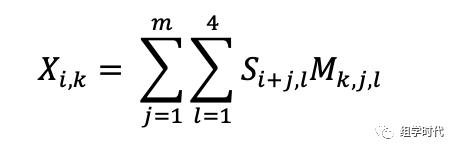
其中,1≤ 以上是关于DL笔记|使用卷积神经网络构建转录因子结合位点预测模型(deepbind)的主要内容,如果未能解决你的问题,请参考以下文章 易基因:全基因组ChIP-seq分析揭示细菌转录因子PhoB的基因内结合位点|mBio