易基因:全基因组ChIP-seq分析揭示细菌转录因子PhoB的基因内结合位点|mBio
Posted E-GENE
tags:
篇首语:本文由小常识网(cha138.com)小编为大家整理,主要介绍了易基因:全基因组ChIP-seq分析揭示细菌转录因子PhoB的基因内结合位点|mBio相关的知识,希望对你有一定的参考价值。
大家好,这里是专注表观组学十余年,领跑多组学科研服务的易基因。
细菌编码许多转录因子(transcription factor,TF),这些转录因子通过与启动子周围的DNA结合并调控RNA聚合酶(RNAP)全酶以结合启动子DNA或异构化为主动转录构象的能力来调节转录起始。目前对TF功能的研究几乎集中在调节基因上游基因间区域的TF结合位点。然而,基因组规模分析鉴定出大量的TF结合位点位于基因内(within genes),基因内TF结合位点的比例在不同TF之间具有显著差异。尽管基因内存在大量TF结合位点,但对其功能知之甚少。
大肠杆菌(Escherichia coli)TF PhoB是一种保守的转录因子,可调控参与磷酸盐稳态的基因转录。大部分PhoB结合位点位于基因内,这些基因内结合位点与可检测的转录调控无关,且在进化上不保守。许多基因内PhoB位点位于H-NS结合区域,可能是由于PhoB和H-NS的共同序列偏好。
2023年04月17日,《mBio》杂志发表了题为“Genome-Wide Mapping of the Escherichia coli PhoB Regulon Reveals Many Transcriptionally Inert, Intragenic Binding Sites”的研究论文,该研究通过对细菌转录因子PhoB结合位点的ChIP-seq、RNA-seq整合分析,揭示了大肠杆菌PhoB的许多基因内转录因子结合位点和转录调控机制。

标题:Genome-Wide Mapping of the Escherichia coli PhoB Regulon Reveals Many Transcriptionally Inert, Intragenic Binding Sites.(大肠杆菌PhoB调控元件的全基因组定位揭示了许多转录惰性的基因内结合位点)
时间:2023-04-17
期刊:mBio
影响因子:IF 7.786
技术平台:ChIP-seq、RNA-seq、RT-qPCR等
研究摘要:
基因组规模分析揭示许多转录因子结合位点在基因内部,引发了关于这些结合位点功能的问题。最近研究揭示细菌基因内的大量转录因子结合位点,但绝大多数结合位点的功能尚未研究。本研究绘制了转录因子PhoB在大肠杆菌基因组中的结合,揭示了大多数PhoB结合位点都位于基因内。分析结果表明,基因内PhoB结合位点与重叠基因调控无关。数据揭示了细菌可以耐受大量非调控性基因内转录因子结合位点的存在,且这些结合位点不受选择性压力影响。
研究使用染色质免疫沉淀测序(ChIP-seq)和转录组测序(RNA-seq)对pho调控元件进行高分辨率的全基因组分析。研究对一组已知的pho调控基因进行分析和扩展,并鉴定出许多基因内PhoB结合位点。分析结果显示,绝大多数基因内PhoB结合位点不保守,且与可检测的调控功能无关。本研究数据表明,单个基因内PhoB位点无功能,转录因子可以结合许多基因内位点,对局部转录(local transcription)几乎没有影响。
实验结果:
(1)磷酸盐限制条件下PhoB的全基因组结合
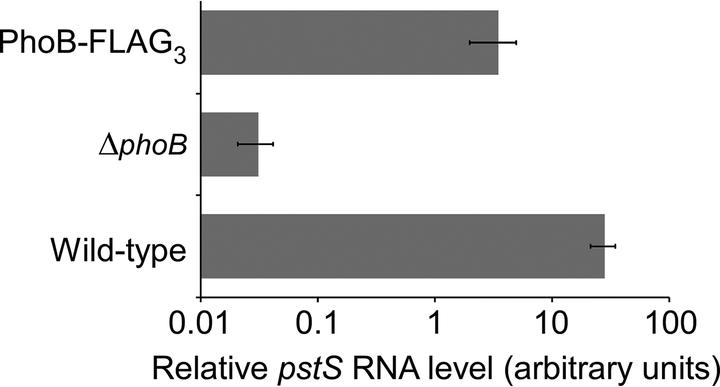
图1:C末端带有FLAG3标签的PhoB活性部分降低。qRT-PCR检测在低磷酸盐条件下生长的细胞中,野生型MG1655/pBAD24(wild type)、MG1655 ΔphoB(CDS091)/pBAD24(ΔphoB)和MG1655 phoB-FLAG3(DMF34)/pBAD24(phoB-FLAG3)中相对minD RNA对照的pstS RNA水平。值是三个生物学重复平均值;误差线表示±1个标准差。

图2:ChIP-seq鉴定PhoB结合位点。
- ChIP-seq数据:(i)低磷酸盐条件下的无标签对照,(ii)低磷酸盐条件下的PhoB-FLAG3标签,(iii)高磷酸盐条件下的PhoB-FLAG3标签。三个基因组区域,其中一个数据来自两个生物学重复。x轴表示基因组位点。y轴表示归一化序列reads覆盖范围,正值表示序列reads比对到正义链,负值表示序列reads比对到反义链。
- 每个ChIP-seq peaks周围100 bp区域显著富集的DNA序列motif。
- PhoB结合位点相对于ChIP-seq peaks中心区域位点分析。
- ChIP-seq鉴定的PhoB结合位点的基因组背景饼图。“基因内和上游”位点是基因内但注释基因起始上游<200bp位点。

表1:ChIP-seq鉴定的PhoB结合区域列表

图3:ChIP-seq和ChIP-ChIP数据集比较分析
- 本研究ChIP-seq数据和已发表的ChIP-ChIP数据集之间的共有区域中,每个ChIP-seq peaks周围100 bp区域显著富集的DNA序列motif。
- 本研究ChIP-seq数据的特异性区域,每个ChIP-seq peaks周围100 bp区域显著富集的DNA序列motif。
(2)pho调控元件的再评估

图4:大肠杆菌野生型和ΔphoB菌株的RNA-seq分析。散点图显示野生型(MG1655/pBAD24)或ΔphoB(CDS091/pBAD24)细胞中所有基因的相对RNA水平。每个数据点对应一个基因,三角形数据点代表先前报道的pho调控元件中的基因,红色三角形表示该转录本ChIP-seq鉴定的上游PhoB位点,灰色三角形表示没有上游位点。红色圆圈表示以前没有报道过的基因位于pho调控元件中,但在ChIP-seq中鉴定出上游PhoB位点。蓝色圆圈表示ChIP-seq鉴定出的内部PhoB位点基因。灰色圆圈表示所有其他基因。
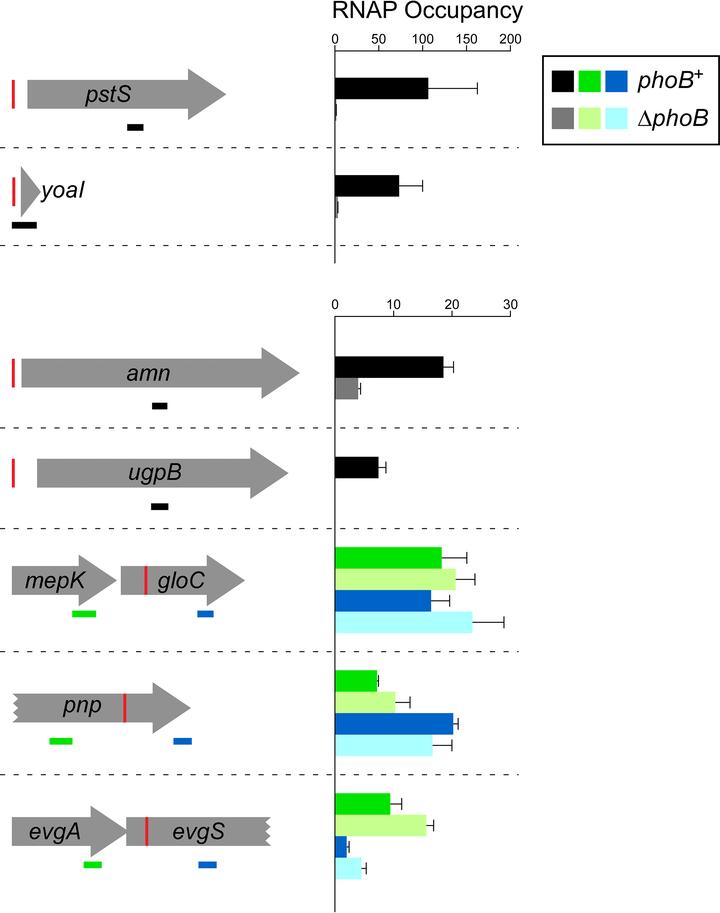
图5:pho调控元件潜在成员基因中RNAP(β亚基)占有率差异。
野生型MG1655(深色条)和MG1655 ΔphoB(CDS091;浅色条)中,ChIP-qPCR检测的RNAP(β亚基)占有率是pho调控元件潜在成员的基因内区域。左侧示意图显示上游或内部PhoB位点的基因(红色垂直线)。水平条表示ChIP-qPCR中PCR扩增子位点,黑色条表示PhoB位点上游基因内的扩增子,绿色条表示基因内PhoB位点上游的扩增子,蓝色条表示基因内PhoB位点下游的扩增子。
(3)起始RNAP的PhoB依赖性招募

图6:野生型和ΔPhoB细胞PhoB结合位点周围σ70占有率差异。
散点图显示ChIP-seq鉴定的phoB结合位点周围400bp区域在野生型MG1655和MG1655 ΔphoB(DMF84)中的归一化σ70占有率。每个数据点代表一个PhoB结合位点。基因间结合位点由红色数据点表示,基因内结合位点由蓝色数据点表示。与PhoB结合位点相关的基因在野生型和ΔPhoB细胞的σ70占有率相差>2倍。
(4)H-NS与许多基因内PhoB位点相关联,但不阻断RNAP招募

图7:H-NS抑制许多启动子的转录
- 通过ChIP-seq鉴定的野生型MG1655和MG1655 Δhns(AMD565a)PhoB结合位点周围400 bp区域的归一化σ70占有率散点图。
- 通过ChIP-seq检测MG1655Δhns(AMD565a)细胞中所有σ70结合位点,鉴定出野生型MG1655和MG1655 Δhns(AMD565a)的归一化化σ70占有率散点图。

图8:H-NS不抑制PhoB依赖性起始RNA聚合酶招募效应
(5)PhoB结合位点的序列保守性
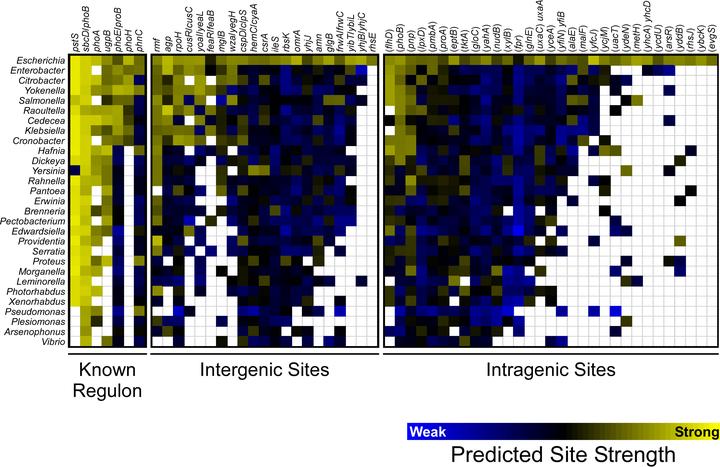
图9:PhoB结合位点在γ -变形菌属(Gammaproteobacteria)物种中的保守性
关于易基因染色质免疫共沉淀测序 (ChIP-seq)
染色质免疫共沉淀(Chromatin Immunoprecipitation,ChIP),是研究体内蛋白质与DNA相互作用的经典方法。将ChIP与高通量测序技术相结合的ChIP-Seq技术,可在全基因组范围对特定蛋白的DNA结合位点进行高效而准确的筛选与鉴定,为研究的深入开展打下基础。
DNA与蛋白质的相互作用与基因的转录、染色质的空间构型和构象密切相关。运用组蛋白特定修饰的特异性抗体或DNA结合蛋白或转录因子特异性抗体富集与其结合的DNA片段,并进行纯化和文库构建,然后进行高通量测序,通过将获得的数据与参考基因组精确比对,研究人员可获得全基因组范围内某种修饰类型的特定组蛋白或转录因子与基因组DNA序列之间的关系,也可对多个样品进行差异比较。
应用方向:
ChIP 用来在空间上和时间上不同蛋白沿基因或基因组定位
- 转录因子和辅因子结合作用
- 复制因子和 DNA 修复蛋白
- 组蛋白修饰和变异组蛋白
技术优势:
- 物种范围广:细胞、动物组织、植物组织、细菌微生物多物种富集经验;
- 微量建库:只需5ng以上免疫沉淀后的DNA,即可展开测序分析;
- 方案灵活:根据不同的项目需求,选择不同的组蛋白修饰特异性抗体。
技术路线:

易基因提供全面的DNA与蛋白互作测序方案。

参考文献:
Fitzgerald DM, Stringer AM, Smith C, Lapierre P, Wade JT. Genome-Wide Mapping of the Escherichia coli PhoB Regulon Reveals Many Transcriptionally Inert, Intragenic Binding Sites. mBio. 2023 Apr 17:e0253522.
相关阅读:
干货系列:染色质免疫共沉淀测序(ChIP-seq)的数据挖掘思路
干货系列:高通量测序后的下游实验验证方法——ChIP-seq篇
转录调控实战 | 一文解决转录调控问题 | chIP-seq | ATAC-seq
做生物想发文章怎么办?转录调控来解析(huyou)!
最简单的情形:
1. 我在研究一个非常重要的基因A,功能已经做得差不多了,现在想深挖,第一步就是想知道哪个转录因子调控这个基因A;
2. 我发现了一个新颖的转录因子B,非常想知道这个B到底再调控哪个基因。
研究方法不过几种:
1. 基于大量的ChIP-seq公共数据挖掘;
2. motif分析预测;
3. 做实验验证,DNase/ATAC-seq;
待续~
参考:嘉因
以上是关于易基因:全基因组ChIP-seq分析揭示细菌转录因子PhoB的基因内结合位点|mBio的主要内容,如果未能解决你的问题,请参考以下文章