07 线性分类器(Linear Classifiers)
Posted
tags:
篇首语:本文由小常识网(cha138.com)小编为大家整理,主要介绍了07 线性分类器(Linear Classifiers)相关的知识,希望对你有一定的参考价值。
机器学习中监督学习模型的任务重点在于,根据已有经验知识对未知样本的目标/标记进行预测。
根据目标预测变量的类型不同,把监督学习任务大体分为分类学习与回归预测两类。
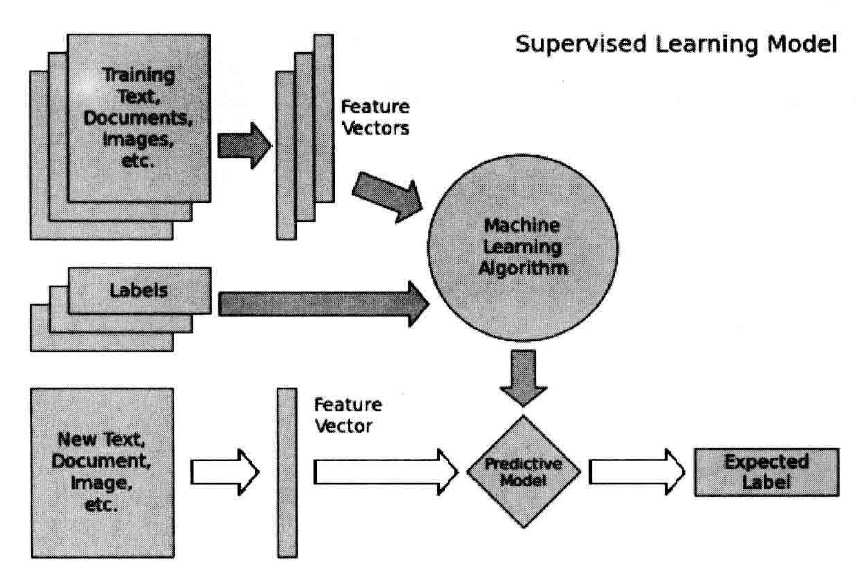
监督学习任务的基本架构和流程
- 首先准备训练数据,可以是文本、图像、音频等;
- 然后抽取所需要的特征,形成特征向量(Feature Vectors);
- 接着,把这些特征向量连同对应的标记/目标(Labels)一并送入学习算法(Machine Learning Algorithm)中,训练处一个预测模型(Predictive Model);
- 然后,采用同样的特征抽取方法作用于新测试数据,得到用于测试的特征向量;
- 最后,使用预测模型对这些待测试的特征向量进行预测并得到结果(Expected Label)。
分类学习
最基础的是二分类(Binary Classification)问题,及判断是非,从两个类别中选择一个作为预测结果;除此之外还有多分类(Multiclass Classification)的问题,即在多于两个类别中选择一个;甚至还有多标签分类(Multi-label Classification)问题,与上述二分类及多分类问题不同,多标签分类问题判断一个样本是否同时属于多个不同类别。
线性分类器
#coding=UTF-8
#良/恶性乳腺癌肿瘤数据预处理
#导入pandas与numpy工具包
import pandas as pd
import numpy as np
#创建特征列表
column_names=[‘Sample code number‘,‘Clump Thickness‘,‘Uniformity of Cell Size‘,‘Uniformity of Cell Shape‘,‘Marginal Adhesion‘,‘Single Epithelial Cell Size‘,‘Bare Nuclei‘,‘Bland Chromatin‘,‘Normal Nucleoli‘,‘Mitoses‘,‘Class‘]
#使用pandas.read_csv函数从互联网读取指定数据
data=pd.read_csv(‘http://archive.ics.uci.edu/ml/machine-learning-databases/breast-cancer-wisconsin/breast-cancer-wisconsin.data‘,names=column_names)
#将?替换为标准缺失值表示
data=data.replace(to_replace=‘?‘,value=np.nan)
#丢弃带有缺失值的数据(只要有一个维度有缺失)
data=data.dropna(how=‘any‘)
#输出data的数据量和维度
data.shape
#[out]:(683, 11)
#准备良/恶性乳腺癌肿瘤训练、测试数据
#使用sklearn.cross_validation里的train_test_split模块用于分割数据
from sklearn.cross_validation import train_test_split
#随机采样25%的数据用于测试,剩下的75%用于构建训练集合
X_train,X_test,y_train,y_test=train_test_split(data[column_names[1:10]],data[column_names[10]],test_size=0.25,random_state=33)
#查验训练样本的数量和类别分布
y_train.value_counts()
#[out]:
# 2 344
# 4 168
# Name: Class, dtype: int64
#查验测试样本的数量和类别分布
y_test.value_counts()
#[out]:
# 2 100
# 4 71
# Name: Class, dtype: int64
#使用线性分类模型从事良/恶性肿瘤预测任务
#从sklearn.preprocessing里导入StandardScaler
from sklearn.preprocessing import StandardScaler
#从sklearn.linear_model里导入LogisticRegression与SGDClassifier
from sklearn.linear_model import LogisticRegression
from sklearn.linear_model import SGDClassifier
#标准化数据,保证每个维度的特征数据方差为1,均值为0。使得预测结果不会被某些维度过大的特征值而住到
ss=StandardScaler()
X_train=ss.fit_transform(X_train)
X_test=ss.transform(X_test)
#初始化LogisticRegression与SGDClassifier
lr=LogisticRegression()
sgdc=SGDClassifier()
#调用LogisticRegression中的fit函数/模块用来训练模型参数
lr.fit(X_train,y_train)
#使用训练好的模型lr对X_test进行预测,结果存储在变量lr_y_predict中
lr_y_predict=lr.predict(X_test)
#调用SGDClassifier中的fit函数/函数用来训练模型参数
sgdc.fit(X_train,y_train)
#使用训练好的模型sgdc对X_test进行预测,结果储存在变量sgdc_y_predict中
sgdc_y_predict=sgdc.predict(X_test)
#从sklearn.metrics里导入classification_report模块
from sklearn.metrics import classification_report
#使用逻辑斯蒂回归模型自带的评分函数score获得模型在测试集上的准确性结果
print ‘Accuracy of LR Classifier:‘,lr.score(X_test,y_test)
#[out]: Accuracy of LR Classifier: 0.988304093567
#利用classification_report模块获得LogisticRegression其他三个指标的结果
print classification_report(y_test,lr_y_predict,target_names=[‘Begin‘,‘Malignant‘])
# precision recall f1-score support
# Begin 0.99 0.99 0.99 100
# Malignant 0.99 0.99 0.99 71
# avg / total 0.99 0.99 0.99 171
#使用随机梯度下降模型自带的评分函数score获得模型在测试集上的准确性结果
print ‘Accuracy of SGD Classifier:‘,sgdc.score(X_test,y_test)
#[out]: Accuracy of SGD Classifier: 0.988304093567
#利用classification_report模块获得SGDClassifier其他三个指标的结果
print classification_report(y_test,sgdc_y_predict,target_names=[‘Begin‘,‘Malignant‘])
# precision recall f1-score support
# Begin 1.00 0.98 0.99 100
# Malignant 0.97 1.00 0.99 71
# avg / total 0.99 0.99 0.99 171
1 #coding=UTF-8
2
3 #良/恶性乳腺癌肿瘤数据预处理
4 #导入pandas与numpy工具包
5 import pandas as pd
6 import numpy as np
7
8 #创建特征列表
9 column_names=[‘Sample code number‘,‘Clump Thickness‘,‘Uniformity of Cell Size‘,‘Uniformity of Cell Shape‘,‘Marginal Adhesion‘,‘Single Epithelial Cell Size‘,‘Bare Nuclei‘,‘Bland Chromatin‘,‘Normal Nucleoli‘,‘Mitoses‘,‘Class‘]
10
11 #使用pandas.read_csv函数从互联网读取指定数据
12 data=pd.read_csv(‘http://archive.ics.uci.edu/ml/machine-learning-databases/breast-cancer-wisconsin/breast-cancer-wisconsin.data‘,names=column_names)
13
14 #将?替换为标准缺失值表示
15 data=data.replace(to_replace=‘?‘,value=np.nan)
16 #丢弃带有缺失值的数据(只要有一个维度有缺失)
17 data=data.dropna(how=‘any‘)
18 #输出data的数据量和维度
19 data.shape
20 #[out]:(683, 11)
21
22
23 #准备良/恶性乳腺癌肿瘤训练、测试数据
24 #使用sklearn.cross_validation里的train_test_split模块用于分割数据
25 from sklearn.cross_validation import train_test_split
26 #随机采样25%的数据用于测试,剩下的75%用于构建训练集合
27 X_train,X_test,y_train,y_test=train_test_split(data[column_names[1:10]],data[column_names[10]],test_size=0.25,random_state=33)
28
29 #查验训练样本的数量和类别分布
30 y_train.value_counts()
31 #[out]:
32 # 2 344
33 # 4 168
34 # Name: Class, dtype: int64
35
36 #查验测试样本的数量和类别分布
37 y_test.value_counts()
38 #[out]:
39 # 2 100
40 # 4 71
41 # Name: Class, dtype: int64
42
43
44 #使用线性分类模型从事良/恶性肿瘤预测任务
45 #从sklearn.preprocessing里导入StandardScaler
46 from sklearn.preprocessing import StandardScaler
47 #从sklearn.linear_model里导入LogisticRegression与SGDClassifier
48 from sklearn.linear_model import LogisticRegression
49 from sklearn.linear_model import SGDClassifier
50
51 #标准化数据,保证每个维度的特征数据方差为1,均值为0。使得预测结果不会被某些维度过大的特征值而住到
52 ss=StandardScaler()
53 X_train=ss.fit_transform(X_train)
54 X_test=ss.transform(X_test)
55
56 #初始化LogisticRegression与SGDClassifier
57 lr=LogisticRegression()
58 sgdc=SGDClassifier()
59 #调用LogisticRegression中的fit函数/模块用来训练模型参数
60 lr.fit(X_train,y_train)
61 #使用训练好的模型lr对X_test进行预测,结果存储在变量lr_y_predict中
62 lr_y_predict=lr.predict(X_test)
63 #调用SGDClassifier中的fit函数/函数用来训练模型参数
64 sgdc.fit(X_train,y_train)
65 #使用训练好的模型sgdc对X_test进行预测,结果储存在变量sgdc_y_predict中
66 sgdc_y_predict=sgdc.predict(X_test)
67
68
69 #从sklearn.metrics里导入classification_report模块
70 from sklearn.metrics import classification_report
71
72 #使用逻辑斯蒂回归模型自带的评分函数score获得模型在测试集上的准确性结果
73 print ‘Accuracy of LR Classifier:‘,lr.score(X_test,y_test)
74 #[out]: Accuracy of LR Classifier: 0.988304093567
75 #利用classification_report模块获得LogisticRegression其他三个指标的结果
76 print classification_report(y_test,lr_y_predict,target_names=[‘Begin‘,‘Malignant‘])
77 # precision recall f1-score support
78
79 # Begin 0.99 0.99 0.99 100
80 # Malignant 0.99 0.99 0.99 71
81
82 # avg / total 0.99 0.99 0.99 171
83
84 #使用随机梯度下降模型自带的评分函数score获得模型在测试集上的准确性结果
85 print ‘Accuracy of SGD Classifier:‘,sgdc.score(X_test,y_test)
86 #[out]: Accuracy of SGD Classifier: 0.988304093567
87 #利用classification_report模块获得SGDClassifier其他三个指标的结果
88 print classification_report(y_test,sgdc_y_predict,target_names=[‘Begin‘,‘Malignant‘])
89 # precision recall f1-score support
90
91 # Begin 1.00 0.98 0.99 100
92 # Malignant 0.97 1.00 0.99 71
93
94 # avg / total 0.99 0.99 0.99 171
以上是关于07 线性分类器(Linear Classifiers)的主要内容,如果未能解决你的问题,请参考以下文章
如果我们在 SGD 分类器上使用校准后的 cv 作为线性核,如何获得特征权重
机器学习理论基础学习3--- Linear classification 线性分类之线性判别分析
降维算法二:LDA(Linear Discriminant Analysis)
机器学习理论基础学习3.4--- Linear classification 线性分类之Gaussian Discriminant Analysis高斯判别模型