KEGG Pathway Anonatation
Posted BioMarkerInfo
tags:
篇首语:本文由小常识网(cha138.com)小编为大家整理,主要介绍了KEGG Pathway Anonatation相关的知识,希望对你有一定的参考价值。
转载于
KEGG 数据库中,把功能相似的蛋白质归为同一组,然后标上 KO 号。通过相似性比对,可以为未知功能的蛋白序列注释上 KO 号。通过KEGG数据库的注释极大的方便我们进行生物学通路的研究,可以直接查看物种某条生物学通路上基因的存在情况。
最简单的方法是看公司给的KEGG注释或者直接下载本物种每个基因的注释结果(比如,植物Phytozome;动植物Ensemble),然后对应到自己的差异基因集里面。
当然如果自己的物种没有KEGG注释结果,那只能自己动手了!
截止到 2015 年 6 月 12 日,KEGG 数据库中共收录了 3,904 个完整的基因组。其中 304 个为真核生物,3,600 个为原核生物。在真核生物中,共有 299 个物种(一个物种可能不止一个基因组),分为 172 科,227 属;在原核生物中,共有 1,858 个物种,分为 809 属。
KEGG 对这些物种的基因序列构成了一个非冗余的 KEGG GENES 数据库;通过 BlastKOALA 和 GhostKOALA, 可对用户提交的蛋白质序列,与 KEGG GENES 数据库分别进行 BLAST 或 GHOSTX 相似性比对,为蛋白质序列注释上 K number,即 KO 号。其中,GHOSTX 比对和 BLAST 比对类似,能够检测到分歧度较大的同源序列(remote homologues),在速度上比 BLAST 大约快 100 倍,两者的区别是:
-
BlastKOALA:用于注释高质量基因组,只能提交 5,000 - 10,000 条蛋白质序列。
-
GhostKOALA:用于注释宏基因组,文件大小为 300 M 以内。
有了 KO 号,就可以重构 KEGG 数据库中的 KEGG pathways 及其他分子网络,然后进行其它分析。
这里以 BlastKOALA 为例,对蛋白质序列进行 KO 注释。
分析步骤如下:
-
到这个网页:http://www.kegg.jp/blastkoala/
-
上传 fasta 格式的蛋白质序列
选择物种所属的分类单元,如这里选择植物“plant”
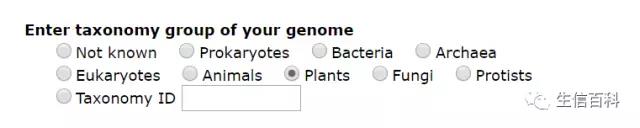
选择一个数据库进行比对。这些数据库由 KEGG GENES 分别在种、属、科水平去冗余后生成。这里选“属”水平的真核生物,如下图右表所示,上传的蛋白质序列限制为 7,500 条序列。

填写自己的邮箱地址,并提交任务,开始分析
回跳转到这个界面,耐心等待即可

分析完成后,会收到邮件通知。

点击链接,返回如下结果。需要注意的是分析结果会在 7 天后删除,所以尽快将结果下载到本地。
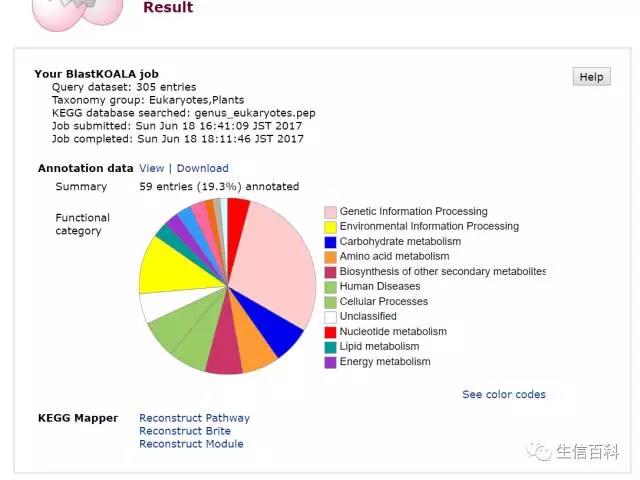
在这个结果界面,可以查看 pathway 等信息。
Reference: Kanehisa, M., Sato, Y., and Morishima, K. (2016) BlastKOALA and GhostKOALA: KEGG tools for functional characterization of genome and metagenome sequences. J. Mol. Biol. 428, 726-731.
以上是关于KEGG Pathway Anonatation的主要内容,如果未能解决你的问题,请参考以下文章