预测氨基酸替换的致病性及分子机制:MutPred工具的使用
Posted
tags:
篇首语:本文由小常识网(cha138.com)小编为大家整理,主要介绍了预测氨基酸替换的致病性及分子机制:MutPred工具的使用相关的知识,希望对你有一定的参考价值。
MutPred的功能是预测氨基酸替换后的致病性及其分子机制,旧版本见链接:http://mutpred1.mutdb.org/
新版本更新为MutPred2,见网站链接:http://mutpred2.mutdb.org/index.html,如下图所示,点击predict here
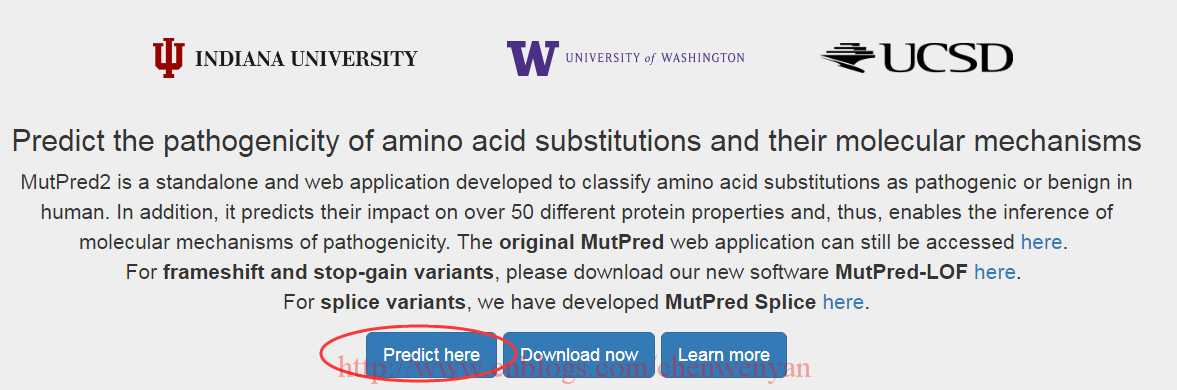
操作步骤见下图:
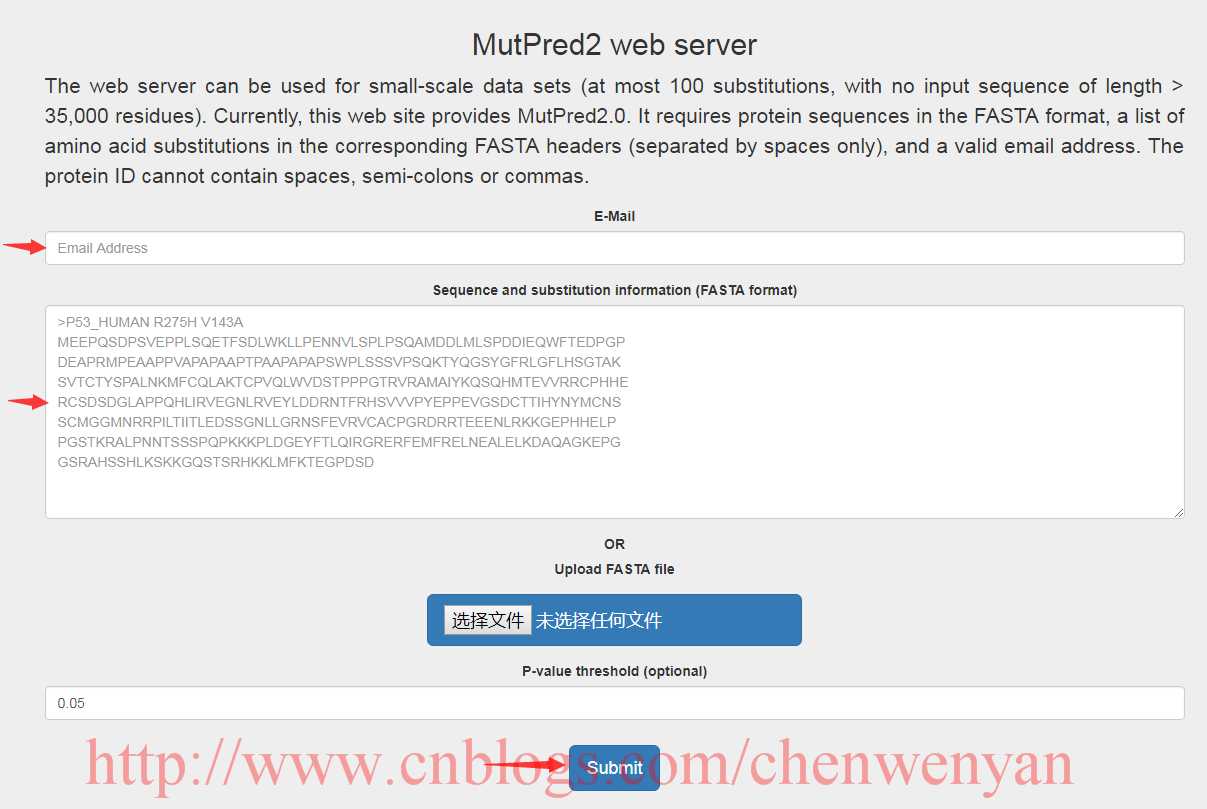
输入邮件地址、输入想要查询的蛋白质序列、最后submit等结果。
重点讲中间输入的蛋白质序列怎么来的:
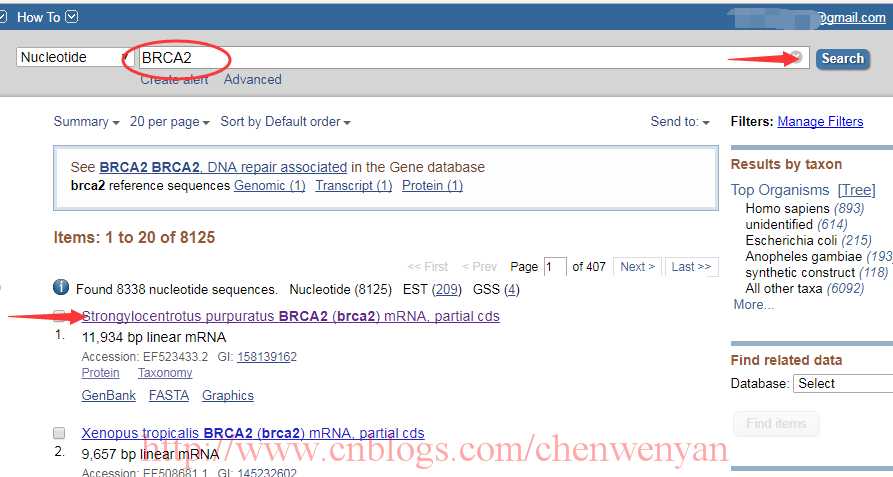
首先,进入网站https://www.ncbi.nlm.nih.gov/nuccore/
假定寻找BRCA2 蛋白质氨基酸序列改变的致病性和分子机制,则在网页中输入BRCA2 ,点击搜索,在结果展示页点击第一个,如下图所示:
进入后,找到CDS,查看translation,即该蛋白质对应的野生型氨基酸,见下图:

回到MutPred2的页面,箭头修改为:
>BRCA2_HUMAN 替换氨基酸及位置
框框里为野生型的氨基酸序列,即上个步骤找到的translation。见下图:

提交后,邮箱会收到工作确认邮件,大约几分钟后就会收到结果,示例结果如下:

(图片来源于:http://mutpred2.mutdb.org/help.html#interpret)
以上是关于预测氨基酸替换的致病性及分子机制:MutPred工具的使用的主要内容,如果未能解决你的问题,请参考以下文章
密码子演化假说|凝固事件假说|立体化学假说|共进化假说|代谢途径相关性假说|四重兼并|假四重兼并|最小损伤原则|AU-rich|GC-rich|逐步进化假说|分子机制进化假说