每日学习记录20230306_培养基
Posted Molesular Blog
tags:
篇首语:本文由小常识网(cha138.com)小编为大家整理,主要介绍了每日学习记录20230306_培养基相关的知识,希望对你有一定的参考价值。
20230306:培养基
-
细菌培养基
M9培养基和lb培养基. -
细菌对数生长期(logarithmic growth phase)

测试原理是:不同的细菌个数对光的反射和折射不同,导致吸光系数不同,进而测量细菌不同生长状态下的OD值.
-
Molecular structural diversity of mitochondrial cardiolipins文献解读
1. 峰值集成和数据分析:
- 使用MZmine2提取mzML
- 裁剪每个数据集的相关区域(原始数据方法/过滤/作物过滤器:1.8-12.5 min,1000-1600 m/z)和基线修正数据(原始数据方法/过滤/基线校正:TIC,0.2 m/z箱,非对称基线校正器:100,000,000平滑,0.5不对称因子)。可以创建具有这些参数的批处理分析管道以进行自动化。
- 通过使用精选的心磷脂峰列表(原始数据方法/峰检测/目标峰检测),识别和整合每个样本中的所有心磷脂峰。
- 该峰值列表包含多达135个心磷脂和35个单心磷脂的m/z和保留时间信息,它们的第一个同位素峰值已在所有测量样本中确定。
- 为了补偿保留时间和m/z位移,使用各自样品中的ISTD(CL(14:0)4)和CL(18:4)4峰测量的m/z信息对模板峰列表进行校准。通过线性回归分析和使用R环境(http://www.r-project.org/)来执行这一步
- 为目标峰值检测设定以下参数:100%强度公差、0.15 m/z或50 ppm m/z公差和0.2 min保留时间公差。根据在400到800之间的个体样本噪声水平调整噪声水平。通过目视检查检查正确集成,必要时手动修正或调整集成参数。将示例特定峰值列表导出为CSV文件以供文档。
- 将MZmine2中所有特性的合成综合峰面积数据导出为CSV结果文件。
2. 从MS1数据中获得心磷脂的定量:
- 总结了单同位素质量的峰面积数据及其各自的第一同位素峰面积。
- 使用心磷脂标准品的稀释系列(步骤19D),使用具有对数转换的线性回归模型将峰面积转换为浓度值。
- 当适用时,使用每个单独样品的内标准测量值来调整潜在的稀释误差。
- 将获得的浓度数据归一化为样品中相应的蛋白质含量,并进行了平行测量(见步骤13)
3. MS2数据中心磷脂结构信息的数学建模:
- 执行如补充R-Script 1中所述的分析步骤。
- 这个分析基于MS2光谱包含mzML文件(步骤22),量化心磷脂MS1结果(CSV文件来自步骤23),质量公差信息描述质量精度和使用的质谱仪的分辨率,以及碳链长度和双键计数的边界预期PG片段。
- 使用R包
mzR导入mzML数据 - 根据相应的MS1心磷脂峰窗对记录的MS2片段谱进行注释,鉴定亲本心磷脂种类,并过滤相关的MS2谱
- 在双键范围[0,15]和碳侧链长度范围[20,50]内构建所有理论上可能的PG单体的列表。根据主要碎片路径为每个PG计算三个m/z值。
- 利用所构建的理论片段列表,根据仪器质量公差将碎片分组为唯一质量,构建片段标注矩阵。
- 用一个描述单个片段路径的相对效率的向量形成片段注释矩阵的标量向积\\([_1 , _2 , _3]\\). 允许从给定的PG配置文件中预测片段模式\\(G_ℎo=[_1,x_2,_3..._]\\),因此是MS2光谱自动分析所需的一组关键的约束条件(见方程(1))。
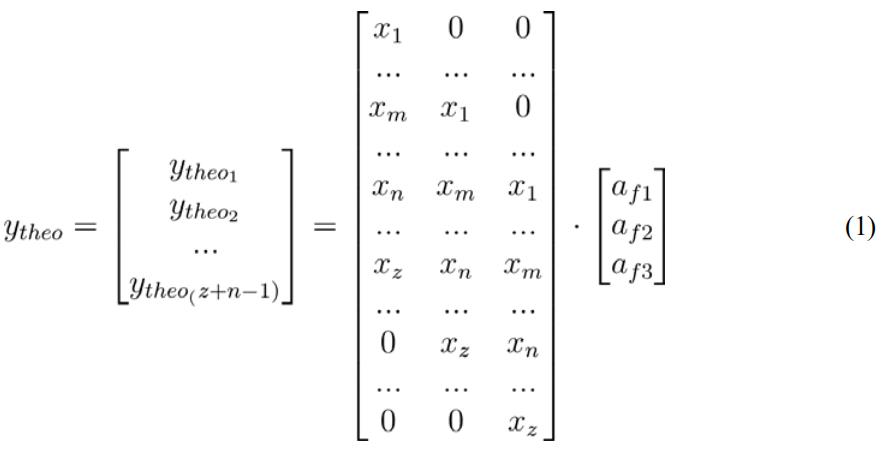
- 将单个的MS2光谱转换为一个向量\\(_obs\\),遵循与\\(_ℎo\\)相同的逻辑格式,以质量公差依赖的方式注释归一化峰值强度。如果需要,请使用内标CL(14:0)中记录的碎片对MS2光谱进行先前的质量校准。
- 将最终的最小化函数构造为理论破碎化模型\\(_ℎo\\)与重新格式化的MS2谱\\(_obs\\)之间的欧氏距离(式2)
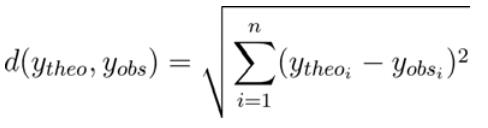
- 应用通用优化函数optim,使参数上的等式(2)最小化[1,2,3....]。]和\\([_1 _2 _3]\\)。作为\\([_1,_2,_3..._]\\)的初始值。]使用\\(_obs\\)的范围[1,z]和10:1:3的ratio来提高碎片路径的效率。
- 使用L-BFGS-B方法运行optim函数,该方法利用了BFGS拟牛顿算法(10)的有限内存修改。将方框约束设置为[0,1],以考虑待优化参数的轮廓性质。
- 对于每个MS2光谱,结果得到了一个模型的PG轮廓。这包括对各自亲本心磷脂种类的注释,以及总MS2强度值。
- 为了获得每个心磷脂物种的PG谱,所有求解的谱都根据它们的MS2强度进行加权并进行总结。如果需要进行说明,则将合成的缩合PG曲线投影到它们各自的双键和侧链碳组分上。
4. FA配置文件建模:
- 脂肪酰基侧链的分布按照如上所述的类似方法进行模拟(步骤25)
- 构建一个描述可用脂肪酸酰基空间\\(A_ℎo=[A_1,A_2,A_3,...,A_n]\\)基于碳链长度[10,28]和双键数[0,8]的边界。
- 从这个脂肪酸列表中计算所有独特的双组合,以获得一个PG谱\\(G=[G_1,G_2,...,G_]\\),描述理论上可用的PG空间。
- 生成一个函数计算\\(G_ℎo\\),其中每个向量元素由所有根据:\\(G_=A_⋅A_\\)的相关相对脂肪酸丰度构造
- 为了生成\\(G_bs\\),用结果配置文件注释\\(G_ℎo\\)的结构\\([_1,_2,_3...x_n]\\)。从步骤25开始
- 在类比步骤25中,使用具有[0,1]盒约束的optim函数和LBFGS-B方法,最小化\\(G_bs\\)and\\(G_ℎo\\)在参数\\(A_ℎo\\)上的欧氏距离(方程2)。根据公式(3)估计来自\\(G_bs\\)的\\(FA_ℎo\\) 分布的初始值,得到具有偶数碳数和最大双键对称性的最近的脂肪酸对。
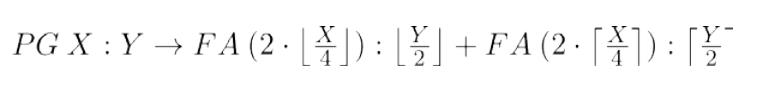
- 脂肪酰基谱既可以从描述样品中PG组成的累积PG谱中解决,也可以是针对单一心磷脂物种的PG组成。
TestLink学习——每日例会记录
2017.11.16例会内容
1、操作视频制作小组制作出了TestLink完整操作流程的图文文档,并在群里分享
2、各小组下载操作文档,在个人电脑上进行操作
3、撰写工具使用手册小组通过操作流程,记录下工具的使用说明,并通过具体例子来进一步说明TestLink软件的作用,完成工具使用手册的撰写
4、各小组成员在课后进行第二次例会,并在例会中,一同协助视频拍摄小组进行录屏拍摄的协作工作
5、最后完成工具实用手册和软件操作视频
6、博客记录小组完成最后的例会内容记录,并生成word文档,最终整合所有作业资料形成zip文件,并在SPOC平台上提交
以上是关于每日学习记录20230306_培养基的主要内容,如果未能解决你的问题,请参考以下文章