第一课第三周1-2节-了解医学图像分割以及探索 MRI 数据格式以及作业解读
Posted Tina姐
tags:
篇首语:本文由小常识网(cha138.com)小编为大家整理,主要介绍了第一课第三周1-2节-了解医学图像分割以及探索 MRI 数据格式以及作业解读相关的知识,希望对你有一定的参考价值。
本周将学习图像分割,图像分割在许多医学影像应用中起着至关重要的作用,例如组织大小的量化、疾病的定位和治疗计划。
我们将重温您在过去两周学到的一些相同的想法,看看它们是如何扩展到图像分割的。
在本课中,我们将学习 MRI 数据和肿瘤分割。了解MRI数据,对于指导我们如何在下一课中为构建分割模型和思考如何表示数据非常重要。
正如你很快就会看到的,使用3D医疗数据提供了一些重大挑战,你将学习一些创造性的想法来应对这些挑战。
MRI 数据
我们将首先讨论如何表示 MRI 数据。我们希望能够以一种可以输入到分割模型中的形式来表示 MRI 数据。
如你所见,我们的核磁共振图像不仅仅是一个像X光片那样的二维图像,而是一个三维立体图像。此外,一个MRI例子将由多个序列组成,这将包括多个3D体积。
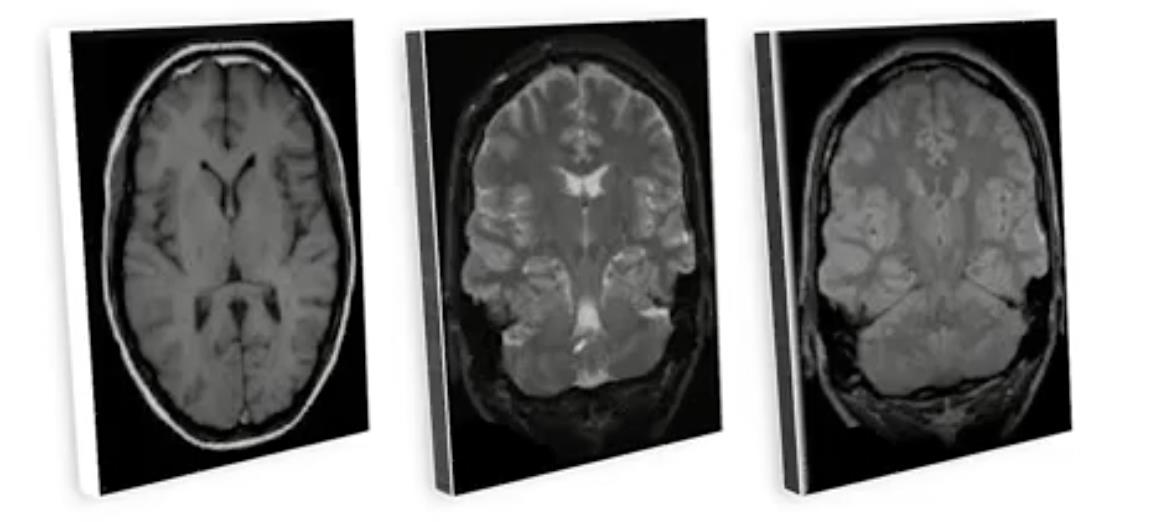
我们将研究如何将这些多个3D体积合并为一个3D体积。
为此,让我们从大脑中挑一层。

我们将用于组合来自不同序列的信息的关键思想是将它们视为不同的通道。
我们可以说其中一个通道是红色的,一个是绿色的,还有一个是蓝色通道,就像我们有三个通道一样。
对代表RGB通道的三个通道的类比对于我们将如何组合这些通道非常有用。当有四个或五个序列时,这些序列可以用四个或五个通道来表示。
一旦每个序列用不同的通道表示,我们现在要做的就是将这些序列组合在一起生成一个图像,即所有序列的组合。
对我们来说,这是一个RGB图像。对于机器来说,这些是以深度维度堆叠的通道。
组合这些序列的一个挑战是它们可能彼此不一致。例如,如果患者在获取这些序列中的每一个序列之间移动,那么他们的头部可能会在一个序列中倾斜。
如果我们合并图像时,图像没有相互对齐,那么红色通道中某个位置的大脑区域与绿色通道或蓝色通道中的相同位置不对应。
那么,我们如何解决这个对齐问题呢?一种通常用来解决这个问题的预处理方法称为图像配准(image registration)。

图像配准的基本思想是对图像进行变换,使它们彼此对齐或配准。我们将不再详细讨论图像配准。在作业中,你将使用已经配准的图像。
但是,在尝试合并三维体积时,注意图像配准是一种有用的工具
作业解读
想要更多的了解 MRI 数据,查看第一次作业
作业文件:
吴恩达-医学图像AI 专项课程-作业/第一课/week3/AI4M_C1_W3_lecture_ex_01.ipynb
使用的MRI数据来自 Medical Segmentation Decathlon

这是一个极其丰富的数据集,提供 3D 表示中每个点(体素)相关联的标签。最后,在本周的作业中,您将训练一个神经网络,对常见的脑部疾病进行三维空间分割预测。
在本次作业中你将学习如下内容
1 加载 nii.gz 数据
使用 nibabel 库加载数据
import nibabel as nib
image_path = "BraTS-Data/imagesTr/BRATS_001.nii.gz"
image_obj = nib.load(image_path)
2 提取数据为numpy array
image_data = image_obj.get_fdata()
height, width, depth, channels = image_data.shape
print(f"The image object has the following dimensions: height: {height}, width:{width}, depth:{depth}, channels:{channels}")
The image object has the following dimensions: height: 240, width:240, depth:155, channels:4
我们可以看到,这个 MRI 数据有4个通道,每个通道是一个240 * 240 * 155的三维体
3 交互查看数据
由于维度过大,用 plt 无法展示三维体积,但我们可以通过交互的方式分别展示每一层
def explore_3dimage(layer):
plt.figure(figsize=(10, 5))
channel = 3
plt.imshow(image_data[:, :, layer, channel], cmap='gray');
plt.title('Explore Layers of Brain MRI', fontsize=20)
plt.axis('off')
return layer
# Run the ipywidgets interact() function to explore the data
interact(explore_3dimage, layer=(0, image_data.shape[2] - 1));

如果经常处理三维图像,建议学习这个交互式方法
4 可视化 label

标签数据数组的尺寸高度:240,宽度:240,深度:155
标签里一共有4种值:[0. 1. 2. 3.],对应以下标签类别:
- 0:正常
- 1:水肿
- 2:非强化肿瘤
- 3:强化肿瘤
如果你不训练图像,我在作业文件中已经给出了3个数据示例,可以尝试查看一下图像信息。
图像地址:
吴恩达-医学图像AI 专项课程-作业/第一课/第一课大作业/week3seg/BraTS-Data
以上是关于第一课第三周1-2节-了解医学图像分割以及探索 MRI 数据格式以及作业解读的主要内容,如果未能解决你的问题,请参考以下文章
第一课第三周3-4节--2D和3D分割的优缺点以及如何构建UNet模型
吴恩达-医学图像人工智能专项课程-第一课第一周6-10节总结+作业解读
吴恩达-医学图像人工智能专项课程-第一课第一周16-18节-如何确保数据集病人不重叠+作业解说