第一课第三周5-6节-数据增强以及dice损失函数
Posted Tina姐
tags:
篇首语:本文由小常识网(cha138.com)小编为大家整理,主要介绍了第一课第三周5-6节-数据增强以及dice损失函数相关的知识,希望对你有一定的参考价值。
既然我们已经讨论了分割体系结构,那么让我们来讨论一种可以应用于这种模型训练的技术,即数据扩充(数据增强 data augumentation)。
我们在前面学过,我们可以对输入胸部X光片的变换,这样每个例子的分类标签都是一样的。

现在让我们看看如何将相同的原则应用于分割,但有几个关键的区别。
分割过程中数据扩充的一个关键区别是我们把图像旋转了,同时标签也得跟着转,保证对齐。

所以当我们将输入图像旋转90度以产生变换后的输入。我们还需要将标签旋转90度以获得变换后的标签。
第二个区别是,我们现在有了三维体积,而不是二维图像。所以这些变换必须应用于整个三维体。
有了这个,我们几乎拥有了训练我们的脑肿瘤分割模型所需的所有部件。
最后我们要看的是损失函数。
让我们举一个非常简单的例子。实际上,我们会有一个更高分辨率的图像,我们会看到一个三维的体积。
但是我们这里简单的二维例子可以让我们快速的理解。

这里P表示分割模型在9个像素上的输出。在每个位置,我们都有肿瘤的预测概率。
G指定每个像素位置上ground truth(gt)。9个像素中有3个是肿瘤,表示为1,其余6个是正常脑组织,表示为0。
表格的每一行是一个单元位置,以及它们对应的预测值和gt。
例如,这里的i4指定概率输出为0.8,gt为0。在这个表中表示p和g将使我们更清楚地理解损失函数。
我们将使用 dice损失 来优化分割模型。dice损失是分割模型中常用的损失函数。
它优点是在不平衡数据的存在下工作良好。在我们的脑肿瘤分割任务中,这一点尤其重要,因为大脑中很小的一部分会成为肿瘤区域。
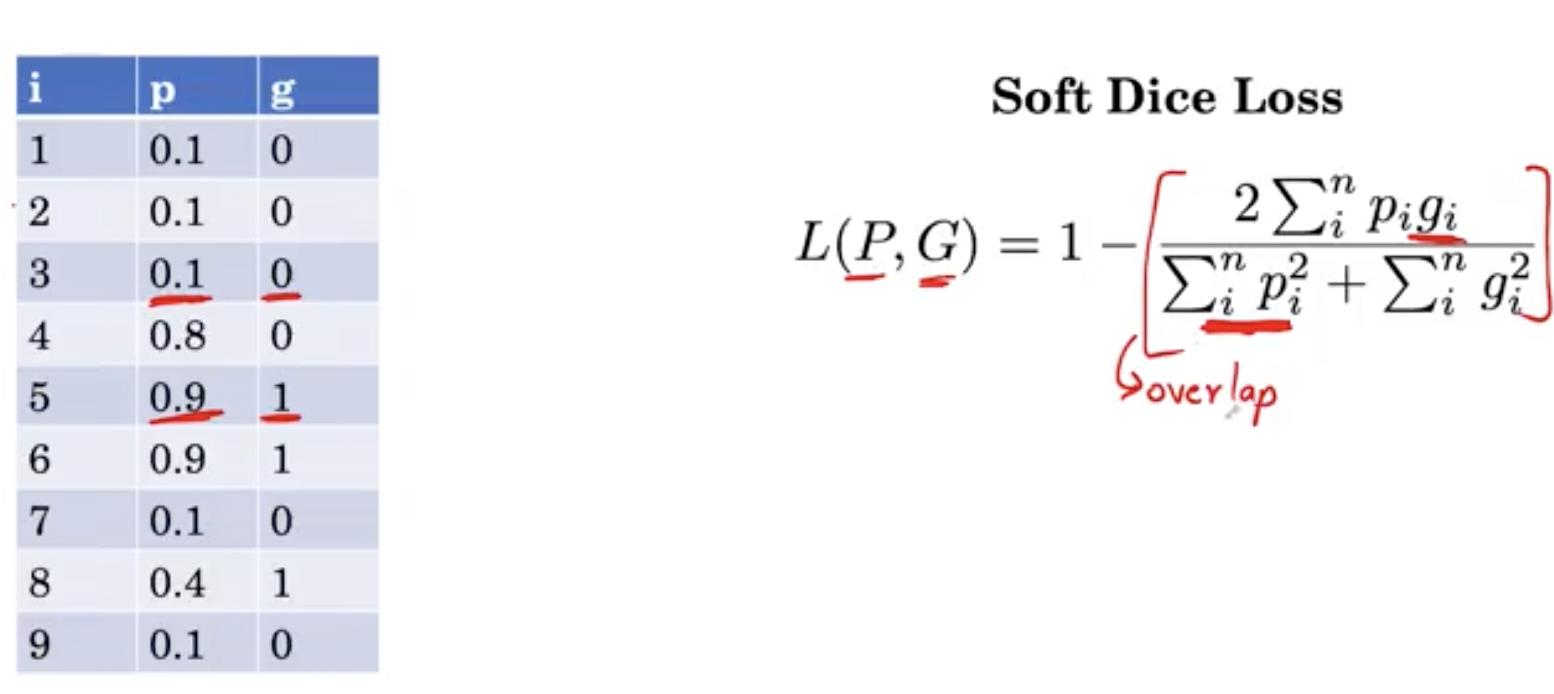
dice损失 将测量P和G之间的误差。我们希望分子很大,当gi等于1时,我们希望Pi接近1。
我们也希望分母较小, 所以当gi等于0时,我们希望Pi接近0,否则这个项会很大,分母也会很大。
现在,我们取1减去这个分数,这样重叠区域越小,损失越大,相反,重叠区域越大,损失越小。
为了计算这个例子中的损失,我们将P和G元素相乘得到pigi。例如,0.9乘以1等于0.9。
为了计算分母,我们需要求pi平方和 和 gi的平方和。类似地,我们可以通过将p列平方得到pi的平方和g列得到gi的平方来计算这些。

我们可以将这些值代入这个特殊例子的dice损失中,结果大约是0.2。
下一步我们将研究分割模型的评估。
文章持续更新,可以关注微信公众号【医学图像人工智能实战营】获取最新动态,一个关注于医学图像处理领域前沿科技的公众号。坚持已实践为主,手把手带你做项目,打比赛,写论文。凡原创文章皆提供理论讲解,实验代码,实验数据。只有实践才能成长的更快,关注我们,一起学习进步~
我是Tina, 我们下篇博客见~
白天工作晚上写文,呕心沥血
觉得写的不错的话最后,求点赞,评论,收藏。或者一键三连

以上是关于第一课第三周5-6节-数据增强以及dice损失函数的主要内容,如果未能解决你的问题,请参考以下文章