文献太多看不过来?教你用R语言快速挖掘pubmed文献信息数据
Posted 医学方
tags:
篇首语:本文由小常识网(cha138.com)小编为大家整理,主要介绍了文献太多看不过来?教你用R语言快速挖掘pubmed文献信息数据相关的知识,希望对你有一定的参考价值。

今天给大家分享两个用来挖掘PubMed的R包,有了现成的工具,省去了不少自己写爬虫的功夫,可以实现:快速分析研究方向的发文趋势,通过关键词找到合适自己的投稿期刊,看看自己领域内的大牛人物;这么多好玩的功能,赶紧行动起来吧,玩转pubmed吧!
RISmed:适合快速分析pubmed文献,统计文章的机构作者信息,期刊信息发表的年份等发文趋势情况,这个包自带网络爬取功能,可以即时下载期刊信息。
pumed.mineR:比较适合用来做pubmed摘要文本的数据挖掘,有摘要英文文本分词、词频统计的功能,摘要内文本基因名的频率统计的功能。
没有安装RISmed可以从CRAN上安装,先载入这个包。
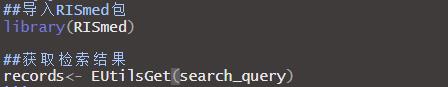
按照Pubmed的检索式写一个字符串,并将他赋值给search_topic,这里我选择了表观遗传和突触可塑性两个关键词作为检索式,中间用AND代表逻辑值同时包含两个字段的文献,还可以通过添加[author]搜索作者,[Affiliation]搜索机构,各种pubmed支持的检索词都可以直接添加;接下来调用EUtilsSummary函数,第一个参数传入检索式的字符串search_topic,db用于选择NCBI的数据库类型,可以选择NCBI的其他基因蛋白数据库,不仅限于Pubmed;retmax用于设置最大获取量,这里为了演示只获取了100条;mindate和maxdate分别设定检索的开始时间和结束时间。这一步其实只返回了pubmedID,和pubmed检索式,没有真正进行文献信息爬取。最后使用summary查看检索结果。我们可以看到它返回了pubmed实际的检索式,年份以及网络状态码。
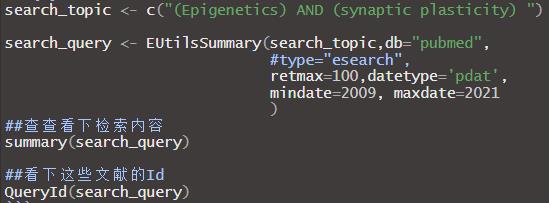
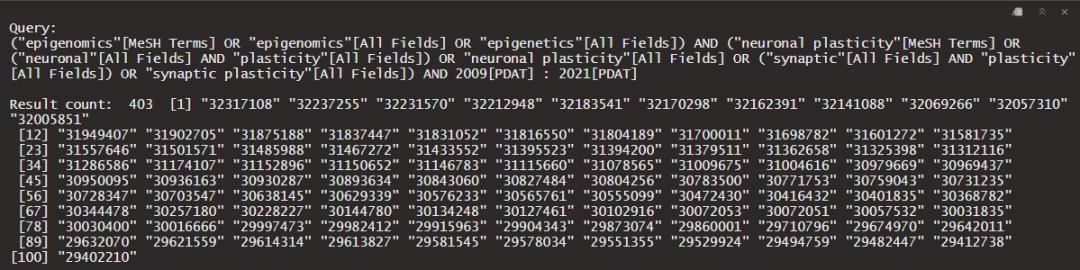
接下来EUtilsGet这个函数将会依据之前的pubmedID进行文献信息爬取,并返回一个Medline对象,这一步需要一定网络速度,特别是连接pubmed的速度,国内有的地方速度还是很慢的,就不一定能下载下来哦,可以多执行几次试试。
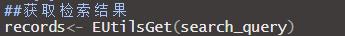
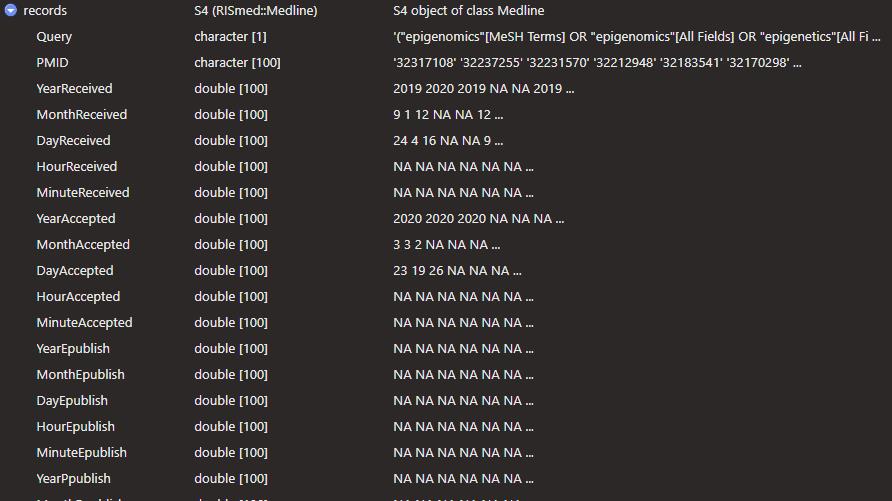
这个Medline是一个S4的对象,里面包含了文章的接收、发表日期,全部的作者和作者机构,文章的摘要。
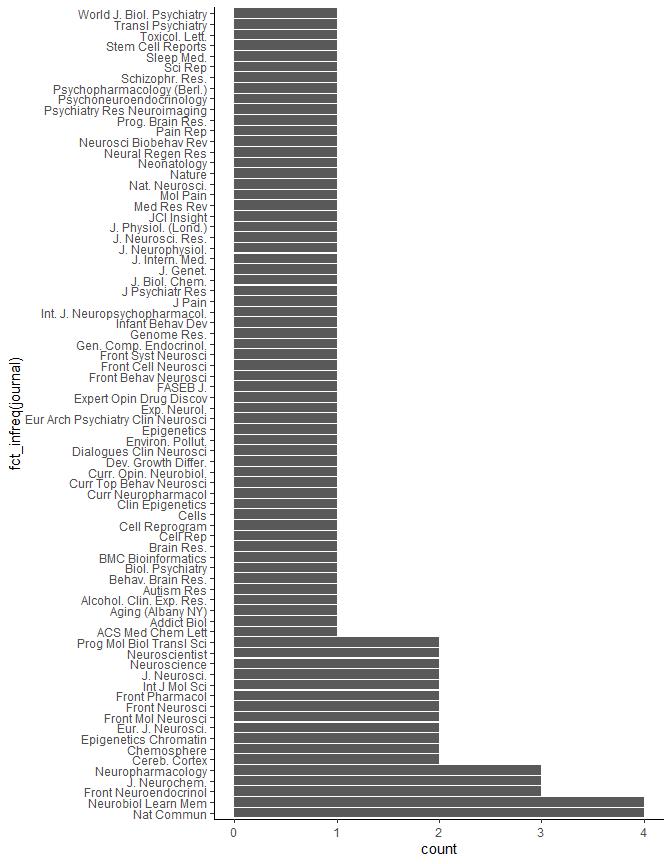
有了文章的摘要,其实可以利用文本分析工具进行英文分词,计算词频,但是这个工作用后面介绍的包pumed.mineR进行效率更高。接下来可以可视化一下,分析这个领域内发论文最多的杂志,还怕找不到合适的期刊投稿吗?各种数据探索性数据分析的方法可以搭配使用,满满的潜力!我们可以看到表观遗传和突触可塑性的文章,当然这里样本量有点少,等我网速好,可以增加点样本量。

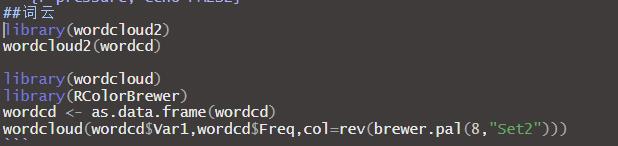
下面绘制词云,可视化结果,载入wordcloud2包绘制可交互的词云,这包传入两列数据,第一列是词,第二列是频率,一键自动化图;也可以用wordcloud和RColorBrewer绘制,这个可以到处PDF矢量图。

这个包并不能即时的通过包内函数爬取pubmed数据,需要自行从pubmed上下载,摘要信息,然后导入包内的函数。
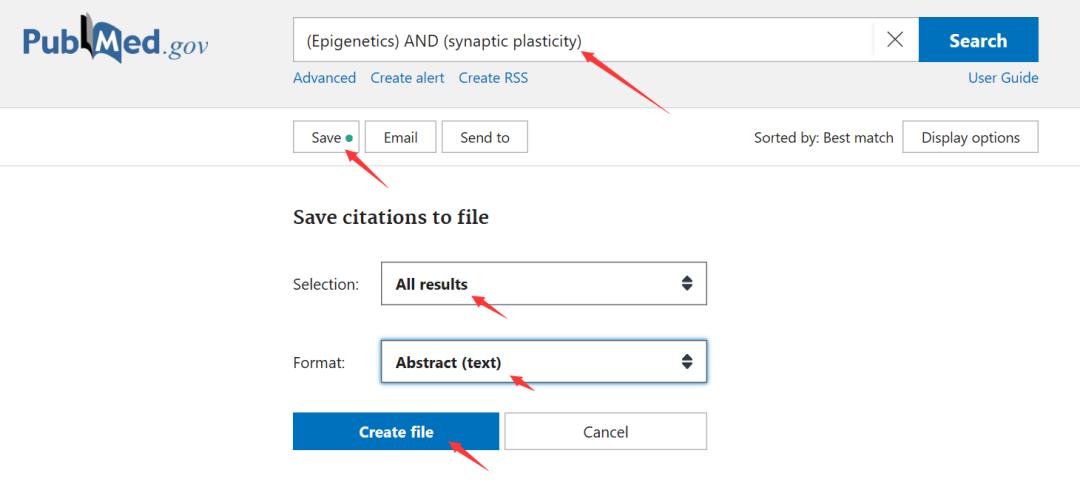
我们首先先从pubmed上根据自己的需要检索文献,如图点击SAVE,选择ALL RESULTS,数据类型选择ABSTRACT,即可下载所有的文章摘要等信息数据,下载好的数据是一个txt文本文件。
如果需要特定的几篇文章,可以选中文章前面的勾,不打勾默认是下载全部文章信息,这里下载了1059篇文章的信息。导入摘要数据成一个S4对象。

重点来了,废了我好大的功夫才找到solution,之前在文本分词的时候出现错误,后来发现由于分词时遇到特殊字符造成,通过Sys.setlocale设置系统参数解决。接下来分词,计算词频。

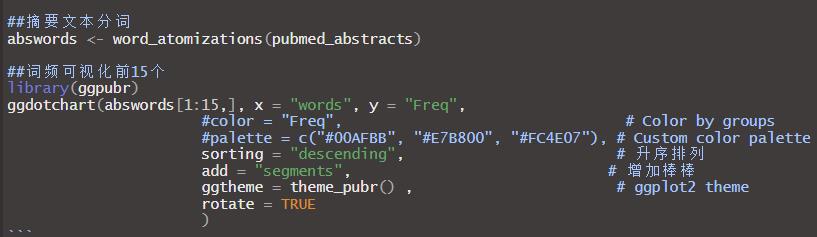
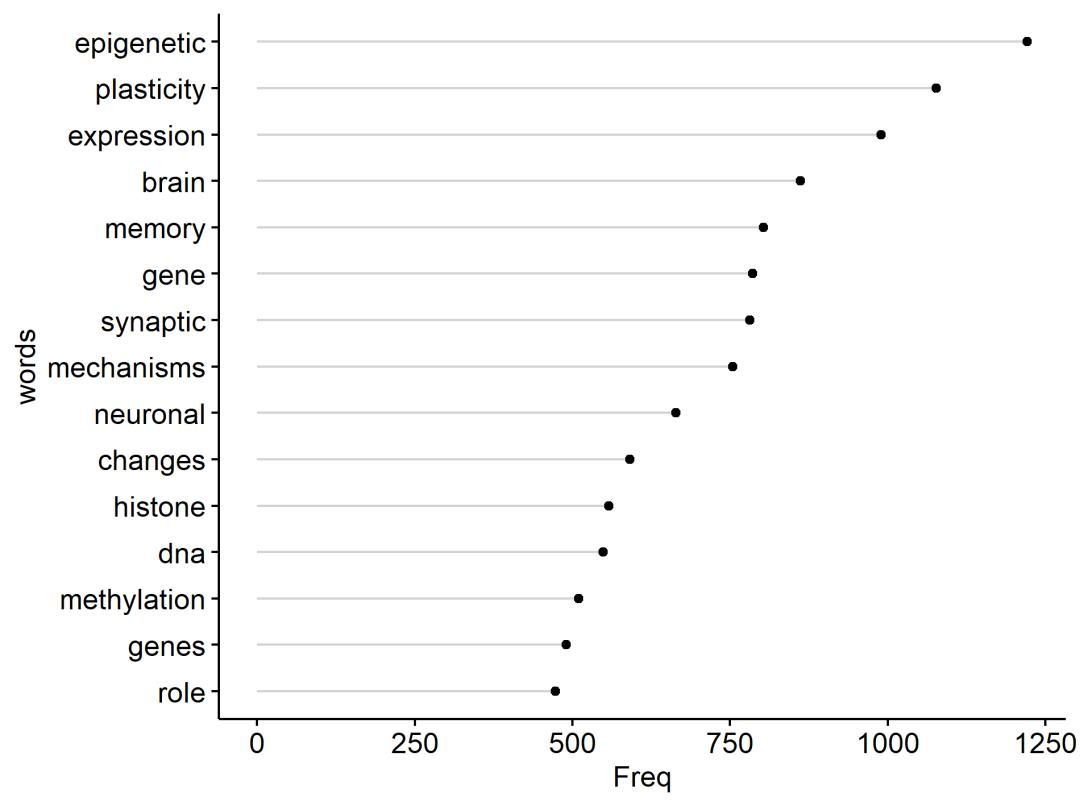
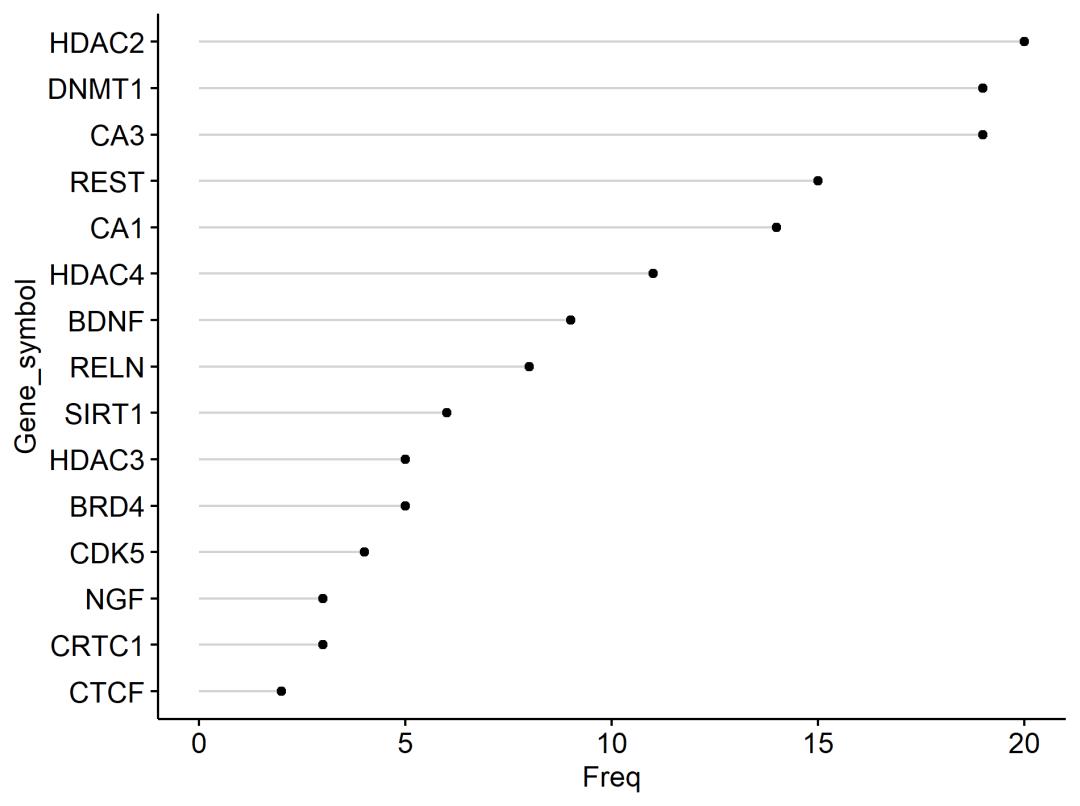
选取前15个出现频率最高的词可视化一下。
gene_atomization函数原理大概是把文献的摘要进行分词,比对摘要中出现的基因名并进行统计,我们从图中可以看到表观遗传和突触可塑性的研究中,出现最多的基因有HDAC2、DNMT1。
这里的基因名必须是HGNC认证的基因名才能被函数识别,而且有可能出现假阳性的情况,例如这里出现的CA3、CA1基因,查看原文可以发现CA3、CA1指的是海马CA3、CA1区,不过也不影响使用。
通过比较RISmed 、pumed.mineR这两个R包的函数,RISmed可以用于查询某领域内文献的逐年发文趋势,发文机构,发文作者等信息,方便对整个研究建立全局的了解。pumed.mineR则适合用来挖掘摘要文本,它内置文本处理函数,短时间内了解上千篇文献的研究不是梦。
有了这两个R包神器,还怕文献读不过来吗?赶紧分析一下自己研究方向的文献,看看自己研究领域的发文趋势吧!

「医学方」现正式向粉丝们公开征稿!内容须原创首发,与科研相关,一经采用,会奉上丰厚稿酬(300-2000元),。
“医学方”始终致力于服务“医学人”,将最前沿、最有价值的临床、科研原创文章推送给各位临床医师、科研人员。
腾讯课堂:https://medfun.ke.qq.com
网易云课堂:http://study.163.com/u/ykt1467466791112
客服微信:yixuefang1234
温馨提示:医学方还设有专门的讨论群哦~各位明星导师都在群中,可以解答各位的遇到的问题,如有兴趣,可以加客服微信后加入群聊
以上是关于文献太多看不过来?教你用R语言快速挖掘pubmed文献信息数据的主要内容,如果未能解决你的问题,请参考以下文章