R语言 | 生存分析之R包survival的单变量和多变量Cox回归
Posted 生物空间站
tags:
篇首语:本文由小常识网(cha138.com)小编为大家整理,主要介绍了R语言 | 生存分析之R包survival的单变量和多变量Cox回归相关的知识,希望对你有一定的参考价值。

在前文初步简介了生存分析的概念,以及展示了一种生存分析模型的使用。Kaplan-Meier是一种非参数的单变量分析方法,通过估计生存率以及中位生存时间,以生存曲线方式展示生存特征。通常,中位生存时间越长,或者曲线变化幅度平缓则代表着预后较好。Kaplan-Meier分析和对数秩检验(Log-rank test)的结合使用可用于比较两组或多组的生存曲线是否存在显著差异,评估关键的预后因素。
另一种方法是Cox回归模型(或称Cox比例风险回归模型,Cox proportional hazards regression model),它既适用于定量预测变量也适用于类别变量。此外,Cox回归模型扩展了生存分析方法:尽管Kaplan-Meier分析非常流行,但其一种局限性是只能用于单变量分析,相比之下,Cox回归模型可分析多种风险因素对生存的影响。
本篇继续简介Cox回归模型,及其在R语言中的计算方法。
Cox回归模型简介
Cox回归是医学研究中常用的统计回归模型,其原理是将生存时间与协变量联系起来(Cox, 1972)。例如在医学领域,期望寻求找出哪个(或哪些)协变量对患者的生存时间具有最重要的影响,Cox回归模型将建立患者的生存率和几个解释变量之间的关系,评估这些因素对生存时间的效应。
Cox回归模型无需生存时间有任何特定的分布,其假设不同变量对生存的影响随时间变化是恒定的,并且在特定范围内是累加的。
Cox回归执行式
Cox回归的执行式如下:
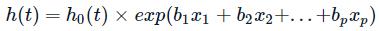
式中:t表示生存时间;h(t)是由一组p个协变量(x1,x2,…,xp)确定的风险函数(hazard function);系数(b1,b2,…,bp)量化了协变量的影响(例如,效应的大小);h0称为基线风险(baseline hazard),如果所有xi=0(即exp(xi)=1),则它对应于风险值;h(t)中的“t”提醒我们,风险可能随时间而变化。
将上述公式进行对数转换后,可以变换为形似线性方程的结构:
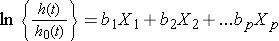
因此,Cox回归模型可以视为风险与变量xi的对数的多元线性回归,其中基线风险是一个随时间变化的“截距”项。
偏回归系数与风险的关系
Cox回归模型涉及检查每个自变量的系数,如b1、b2等,称之为偏回归系数。正回归系数意味着该变量值较高的患者存在高风险,即预后较差;负回归系数意味着该变量值较高的患者预后较好。
将偏回归系数作为自然对数的幂,即eb,写作exp(b),称为风险比(hazard ratio,HR)。因此,当HR>1(即b>0),意味着该变量值较高的患者存在高风险,即预后较差;当HR<1(即b<0),意味着该变量值较高的患者预后较好。
在临床上,也常将HR>1的自变量称之为坏的预后因子,将HR<1的自变量称之为好的预后因子。
R包survival的Cox回归模型
类似前文,R包survival中也提供了执行Cox回归模型的方法,本篇继续以survival包的方法为例展示。
示例数据
survival包的内置数据集lung,为NCCTG(North Central Cancer Treatment Group)的肺癌患者临床资料数据。
library(survival)
#示例数据,详情 ?lung
data(lung)
head(lung)
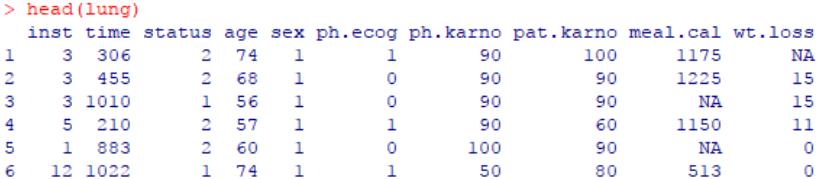
该数据集中记录了228例晚期肺癌患者的生存时间(time,单位为天)、生存状态(status,1为在世,2为去世)以及其它状态特征,例如年龄(age)、性别(sex,1为男性,2为女性)等,详情?lung查看帮助文档中的简介即可。
单变量Cox回归(对于分类变量)
首先期望比较男性与女性肺癌患者的生存率是否具有显著不同,此时可通过单变量Cox回归来解决。(单变量生存分析中的另一种常用方法Kaplan-Meier分析,可)
指定指定数据集中的生存时间(time)、患者生存状态(status)以及分组(sex,性别)等执行单变量Cox回归。
注:尽管分组是分类变量,但仍需要以1、2等连续的离散数值表示。
#单变量 Cox 回归,详情 ?coxph
#生存时间与性别(分类变量)的关系
cox_sex <- coxph(Surv(time, status) ~ sex, data = lung)
cox_sex
summary(cox_sex)
#结果提取,例如
#names(summary(cox_sex))
summary(cox_sex)$coefficients #变量的回归系数、p 值等
summary(cox_sex)$logtest #Likelihood ratio test 的统计量、p 值等
summary(cox_sex)$waldtest #Wald test 的统计量、p 值等
summary(cox_sex)$sctest #Score (logrank) test 的统计量、p 值等

结果的下方,给出了三种类型的检验p值(Likelihood ratio test、Wald test、Score (logrank) test),它们所示的是整个回归方程的显著性,均显示p=0.001,结果非常显著,表明两组(男性和女性)患者之间的生存率具有显著差异。
评估变量的效应,查看上方的自变量的偏回归系数等指标。在单变量Cox回归中,单个自变量的显著性p值和整个回归方程p值是一致的。coef为偏回归系数,exp(coef)即代表了HR,这里HR<1,代表了随数值增大而风险降低。由于1代表了男性,2代表了女性,所以男性患者(组1)比女性患者(组2)的生存期更短,有理由认为男性肺癌患者比女性肺癌患者具有更差的预后,猜测原因可能与男性更多存在吸烟史有关(数据集中没体现)。
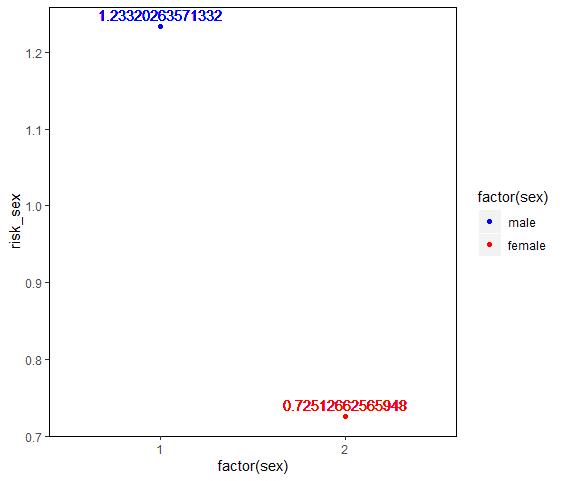
构建好的Cox回归模型可进一步用于预测患者风险。
如下示例,显示男性患者的风险明显大于女性患者。
#通过构建好的 Cox 回归模型执行风险预测
lung$risk_sex <- predict(cox_sex, type = 'risk')
head(lung$risk_sex)
library(ggplot2)
ggplot(lung, aes(factor(sex), risk_sex, color = factor(sex), label = risk_sex)) +
geom_point() +
scale_color_manual(values = c('blue', 'red'), limits = c('1', '2'), labels = c('male', 'female')) +
theme(panel.background = element_rect(fill = 'transparent', color = 'black'),
panel.grid = element_blank()) +
geom_text(vjust = -0.5, show.legend = FALSE)
对于生存曲线,可通过survminer包绘制,比较男性和女性患者的生存率。
注:以下过程实际上执行了一个,借助Kaplan-Meier的结果绘制生存曲线。
library(survminer)
#生存曲线和累积风险曲线
KM <- survfit(Surv(time, status) ~ sex, data = lung) #先执行一个 Kaplan-Meier 分析
ggsurvplot(KM, conf.int = TRUE, palette = c('blue', 'red'), risk.table = TRUE, pval = TRUE)
ggsurvplot(KM, conf.int = TRUE, palette = c('blue', 'red'), fun = 'cumhaz')
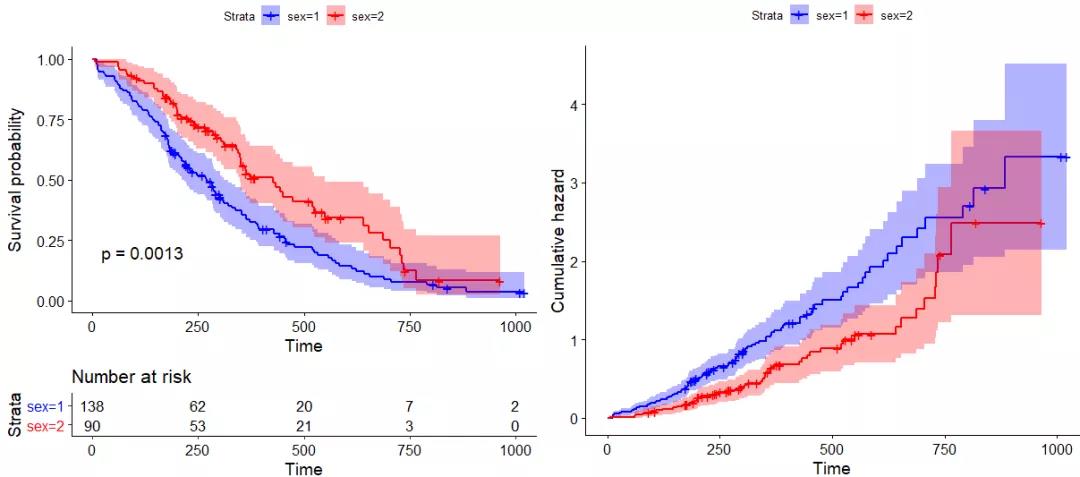
曲线中同时显示了置信区间以及分位数时间段的生存患者数量等,便于我们比较。但需要注意的是,这里显示的p值实际上是Kaplan-Meier分析的p值,同样是非常显著的。
通过生存曲线以及风险曲线,可以直观看出男性肺癌患者比女性肺癌患者具有更差的预后,体现在生存曲线下降更快以及风险曲线上升更快。
单变量Cox回归(对于连续变量)
由于通常认为癌症与年龄密切相关,即老年人口存在更高的风险,因此年龄因素也是不可忽略的。接下来试图通过单变量Cox回归来探索患者生存时间与年龄的关系。
指定指定数据集中的生存时间(time)、患者生存状态(status)以及多个自变量(sex,性别以及age,年龄)等执行多变量Cox回归。
#生存时间与年龄(连续变量)的关系
cox_age <- coxph(Surv(time, status) ~ age, data = lung)
cox_age
summary(cox_age)
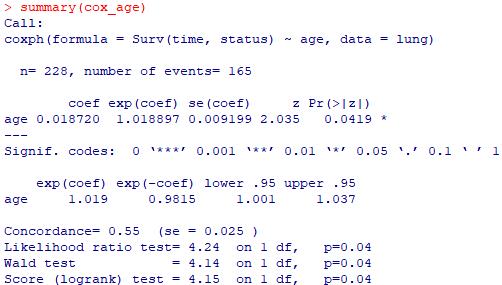
结果显示p<0.05,表明患者的生存时间确实与年龄存在关联。并由于exp(coef)>1,即代表了HR>1,表明风险随患者年龄增加而增加,高龄人群对应了更差的预后。
因为这种连续变量的情况下难以绘制生存曲线,我们不妨绘制散点图查看年龄和生存时间的关系。(以下表示方式可能有些不妥,但只是用于探索趋势,所以散点图+简单的线性回归线似乎仍是可行的)
#生存时间与年龄的散点图
ggplot(lung, aes(age, time, color = factor(status), shape = factor(status))) +
geom_point() +
scale_shape_manual(values = c(1, 16), limits = c('1', '2')) +
theme(panel.background = element_rect(fill = 'transparent', color = 'black'),
panel.grid = element_blank()) +
geom_smooth(method = lm, se = FALSE, show.legend = FALSE)
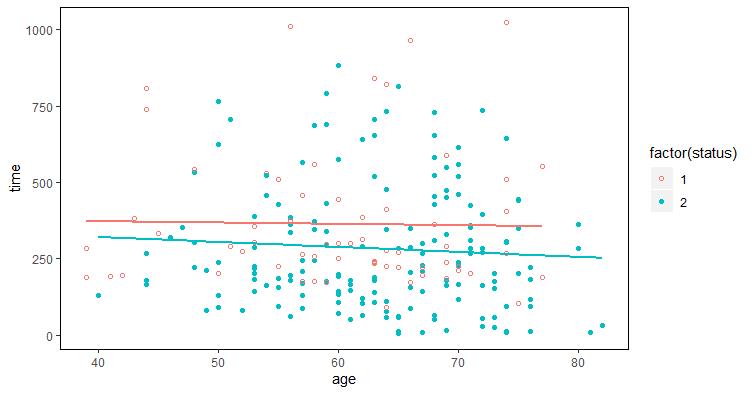
通过观察,患者生存时间表现出随年龄增长而下降的趋势(关注status=2的趋势,这些是已经去世的患者;status=1是仍在世的患者,暂且忽略),同样透露着高龄人群存在更高风险。
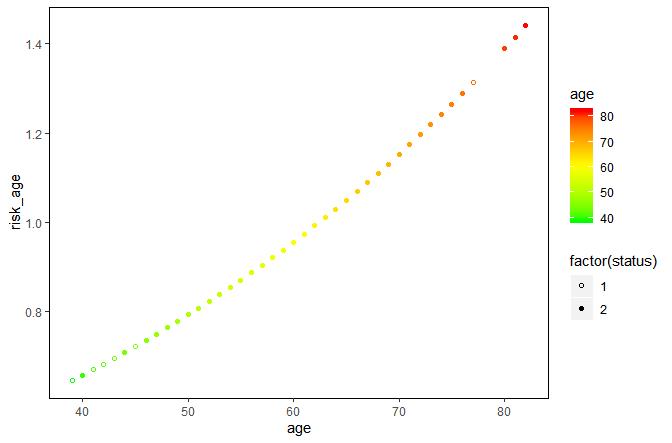
类似地,通过已构建好的Cox回归模型执行风险预测,同样可以清楚地看到,患者风险随年龄增加而增加。
#通过构建好的 Cox 回归模型执行风险预测
lung$risk_age <- predict(cox_age, type = 'risk')
head(lung$risk_age)
ggplot(lung, aes(age, risk_age, color = age, shape = factor(status))) +
geom_point() +
scale_color_gradientn(colors = c('green', 'yellow', 'red')) +
scale_shape_manual(values = c(1, 16), limits = c('1', '2')) +
theme(panel.background = element_rect(fill = 'transparent', color = 'black'),
panel.grid = element_blank())
多变量Cox回归
再来看一种考虑多组变量的情况。现在我们同时考虑性别和年龄与患者生存时间的关系,通过多变量Cox回归来解决。
指定指定数据集中的生存时间(time)、患者生存状态(status)以及将多个自变量(sex,性别以及age,年龄)等同时作为协变量执行多变量Cox回归。
#多变量 Cox 回归,详情 ?coxph
#多变量时,直接在执行式中以“+”相连即可
cox2 <- coxph(Surv(time, status) ~ sex+age, data = lung)
cox2
summary(cox2)
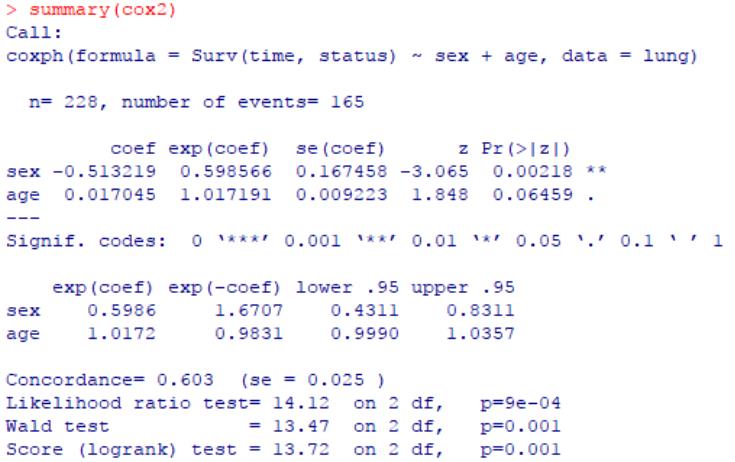
类似的,结果中给出了三种类型的检验p值(Likelihood ratio test、Wald test、Score (logrank) test),它们所示的是整个回归方程的显著性。根据p≤0.001可认为回归方程是统计学显著的,说明在多个自变量中包含了对生存时间具有影响的因素。
在多变量情况下,若评估各个自变量的相对效应,同样可关注上方偏回归系数那一栏,多变量情况下,比较各个自变量的回归p值。性别(sex)更为显著的,表明性别是患者之间生存期不同的主要因素,患者年龄次之,但作为协变量的情况下表现不明显(p>0.05)。
随后再审查偏回归系数等指标,关注exp(coef)信息,和上文中分别使用性别(sex)和年龄(age)的单变量Cox回归具有一致的趋势。性别(sex)的exp(coef)<1(即HR<1),年龄的exp(coef)>1(即HR>1),表明男性肺癌患者比女性肺癌患者具有更差的预后,高龄人群可能存在更高的风险。
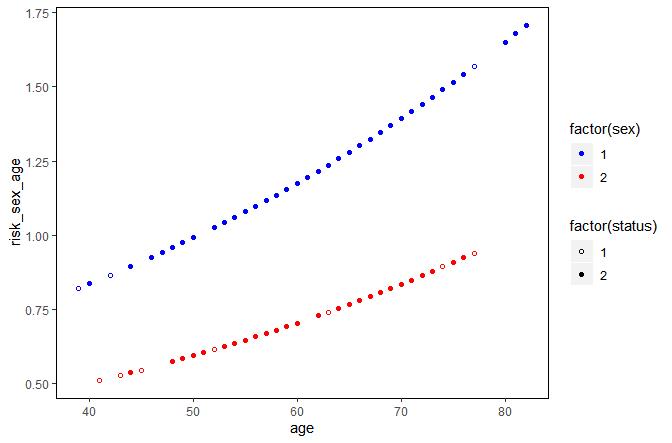
通过已构建好的Cox回归模型执行风险预测,观察两种自变量协同作用对患者风险的效应。可以看到,男性患者(sex=1)比女性患者(sex=2)具有更高的风险,并且均表现出随年龄增加而增加的趋势。
#通过构建好的 Cox 回归模型执行风险预测
lung$risk_sex_age <- predict(cox2, type = 'risk')
head(lung$risk_sex_age)
ggplot(lung, aes(age, risk_sex_age, color = factor(sex), shape = factor(status))) +
geom_point() +
scale_color_manual(values = c('blue', 'red'), limits = c('1', '2')) +
scale_shape_manual(values = c(1, 16), limits = c('1', '2')) +
theme(panel.background = element_rect(fill = 'transparent', color = 'black'),
panel.grid = element_blank())
此外,还可通过survminer包绘制森林图,比较多变量的效应。
如下示例,对本次多变量Cox回归中的性别(sex)和年龄(age)的HR值以及p值等进行可视化。
#森林图,详情 ?ggforest
ggforest(cox2, main = 'hazard ratio', refLabel = 'reference', noDigits = 2)
关于多变量Cox回归中自变量的选择
对于多变量的情况,通常会考虑这样一个问题,就是选择怎样的变量组合是最合适的?以下提供几点参考。
首先可以根据各个自变量的p值进行评估,如果p值特别地大(非常不显著的情况),则表明这个变量作为协变量时效应不明显,可以考虑剔除它。
对比多个Cox模型的AIC值(Akaike信息准则)等信息。AIC会在给定模型的复杂性与其拟合优度之间进行权衡,可以将AIC值视为对应了模型的准确性,AIC值越小的模型表明越有可能准确地预测新数据。
#AIC 值获取,以上述结果为例
extractAIC(cox2)
简约性原则也是必需考虑的,也就是避免使用太多的自变量,尽可能使模型简约更利于解释。
参考资料
以上是关于R语言 | 生存分析之R包survival的单变量和多变量Cox回归的主要内容,如果未能解决你的问题,请参考以下文章
R语言构建生存分析(survival analysis)模型示例
R语言生存分析模型简介及survival包实现实战:基于survival包lung数据集
R语言 | 生存分析及R包survival的Kaplan-Meier
R语言生存分析预测中位生存时间生存曲线可视化log-rank检验HR值分析:基于survival包lung数据集
R语言生存分析详解:KM曲线COX比例风险模型HR值解读模型比较残差分析是否比例风险验证:基于survival包lung数据集