如何利用perl快速筛选差异基因?
Posted SCI狂人
tags:
篇首语:本文由小常识网(cha138.com)小编为大家整理,主要介绍了如何利用perl快速筛选差异基因?相关的知识,希望对你有一定的参考价值。
今天给大家分享一下如何利用perl快速筛选差异基因。例如我们在用GEO2R,或者其他方法得到这样的数据:我们就可以快速筛选差异基因和上下调的差异基因,当然用EXCEL也是可以的,但是没有Perl的速度快。
我们只要将我们的input.txt文件和perl脚本放到同一个文件下,然后双击相应的脚本就可以得到我们的结果文件。
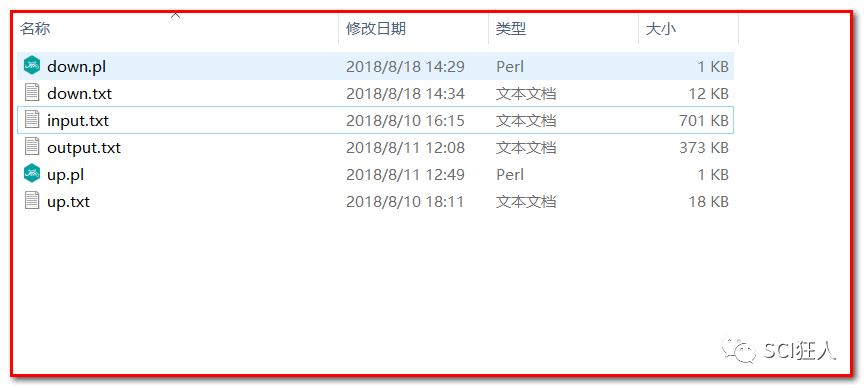
下面我们就将代码放在这里:
筛选上调差异基因的代码:
#!/usr/bin/perl -w
open IN,"<input.txt";
open OUT,">up.txt";
while (<IN>) {
chomp;
my @gene=split /s+/,$_;
if ($gene[1] <=0.05 && $gene[3] >=2) {
print OUT "$_ ";
}
else {
next;
}
}
close IN;
close OUT;
筛选下调差异基因的代码:
#!/usr/bin/perl -w
open IN,"<input.txt";
open OUT,">up.txt";
while (<IN>) {
chomp;
my @gene=split /s+/,$_;
if ($gene[1] <=0.05 && $gene[3] <=-2) {
print OUT "$_ ";
}
else {
next;
}
}
close IN;
close OUT;
►
►
►
►
►
►
►
►
►
►
►
►
►
►
►
►
►
SCI狂人团队
长按二维码
关注SCI狂人
以上是关于如何利用perl快速筛选差异基因?的主要内容,如果未能解决你的问题,请参考以下文章