KEGG如何使用KEGG数据库查询pathway?
Posted 海普洛斯HaploX
tags:
篇首语:本文由小常识网(cha138.com)小编为大家整理,主要介绍了KEGG如何使用KEGG数据库查询pathway?相关的知识,希望对你有一定的参考价值。
怎么看KEGG中的代谢通路图?
图1
我们昨天说到,点开KO的一个ID后,我们会跳往该酶的具体通路信息。如图1所示,在pathway栏目中,有8个与人类丝氨酸甲基转移酶相关的pathway,我们随意点开第一个hsa00260通路,跳往如下页面(图2):
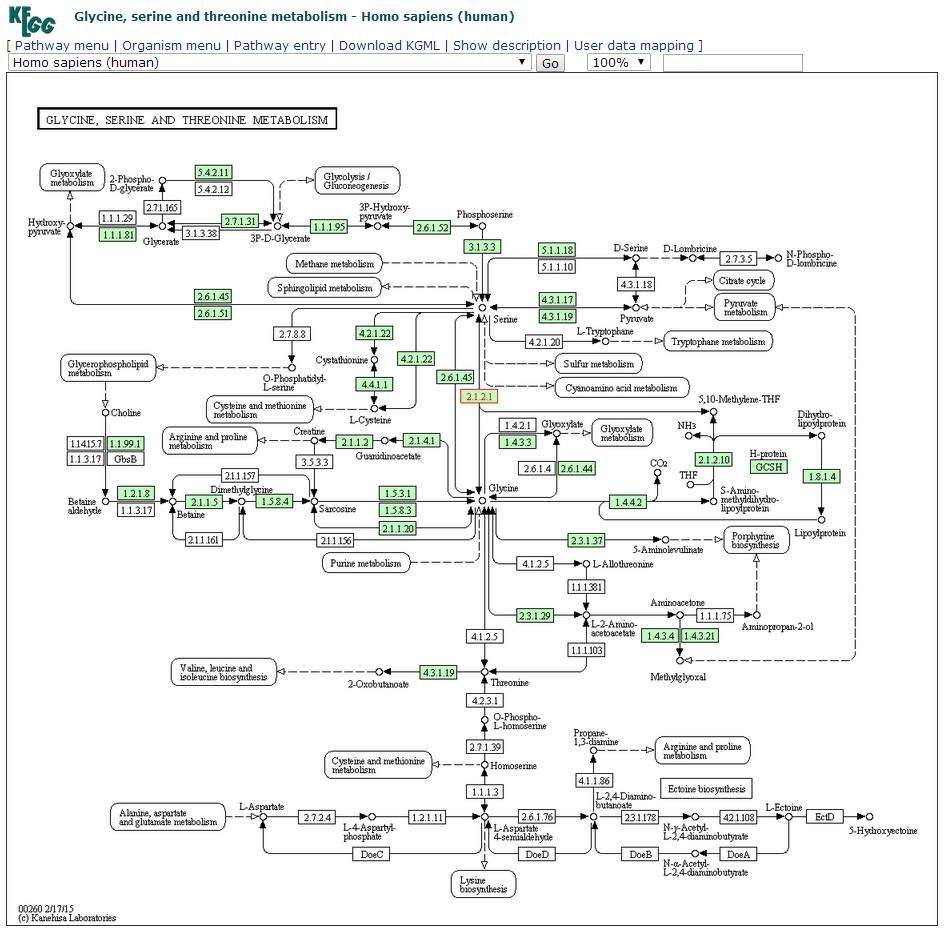
图2
方框一般就是酶,方框里面的2.1.2.1不是IP 而是EC编号;加黄边的方框代表查询的酶所在的位置,如图3所示,2.1.2.1就代表我们查询的丝氨酸甲基转移酶6470和6472。
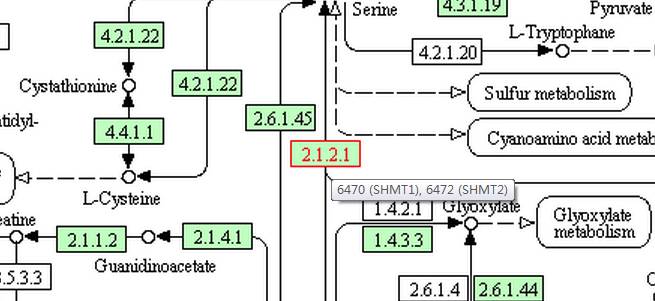
图3
小圆圈代表代谢物,把鼠标放上去,会出现C00065(L-Serine)以及该代谢物即L-Serine的分子式(图4),非常直观。C代表compound,00065是这种化合物在KEGG中的编号,一般在KEGG中数据条目都是这样的,前面一个标志,后面一个五位数编号;大的圆方块,就表示是另一个代谢图了。
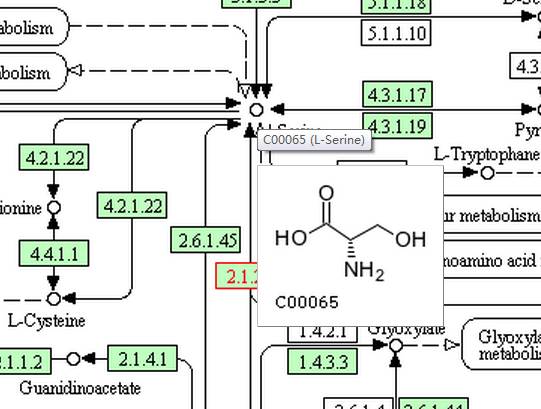
图4
代谢通路图的分类
图2、3、4中的方框涂成绿色也是有讲究的。因为这是一张特定物种(Homo sapiens ,人类)的代谢图,绿色的框框表示专属于这个物种。
在KEGG中有两种代谢图,一种是参考代谢通路图reference pathway,是根据已有的知识绘制的概括的、详尽的具有一般参考意义的代谢图,这种图上就不会有绿色的小框,而都是无色的,所有的框都可以点击查看更详细的信息;另一种就是像上面这样的属于特定物种的代谢图species-specific pathway,会用绿色来标出这个物种特有的基因或酶,只有这些绿色的框点击以后才会给出更详细的信息,白色的方框属于其他物种,点击则没有链接。
这两种图很好区分,reference pathway在KEGG中的名字是以map 开头的,比如map00260,就是甘氨酸、丝氨酸和苏氨酸的代谢途径的参考图,而特定物种的代谢通路图开头三个字符不是map而是种属英文单词的缩写(一个属的首字母+2个种的首字母)比如我们上面举的栗子:人类的甘氨酸、丝氨酸和苏氨酸的代谢通路图hsa00260属于species-specific pathway,hsa是Homo sapiens(人属智人种)的缩写。
那么:怎么找这两种图呢?
(1)有下拉列表的时候,在列表选择reference 或者是特定物种即可。
(2)在pathway检索的页面http://www.genome.jp/kegg/pathway.html,如图5:
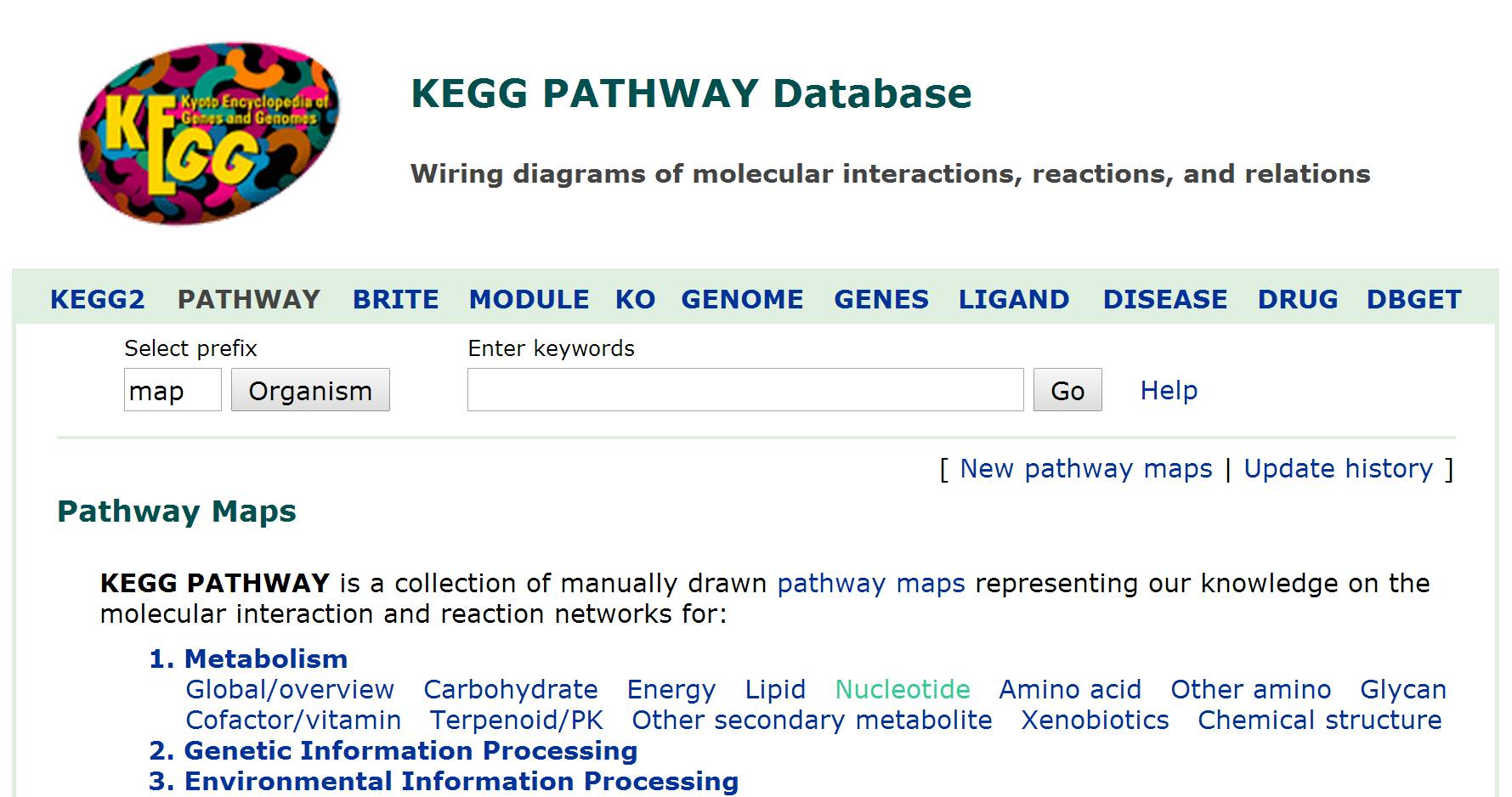
图5
默认的就是map,参考图,你想要什么物种的代谢图写上它的名称就好了(种属缩写),如果不知道是哪3个字母,点击organism 选择即可(不过你点进去也是一片空白,你要提示两个字母才会给出下拉条目)。例如,我在keyword搜索框中写入hsa,跳往人类的所有代谢通路图,第一个是芳香族化合物的降解通路图(图6):
图6
到处点点都可以找到的!
我们回头看下图5,在导航栏上有brite、module、KO、genome、gene、ligand、disease、drug等选项,学会了以上找pathway的方法,以此类推,我们同样可以找到相关的gene、drug等信息。
纠错:昨天的KEGG第一讲中,点击Glutamate metabolism就进入其代谢通路图,应该改为点击Glycine,serine and threonine metabolism就进入其代谢通路图。
参考资料
[1] http://blog.sciencenet.cn/blog-364884-779116.html
[2] http://blog.sciencenet.cn/blog-364884-779438.html
以上是关于KEGG如何使用KEGG数据库查询pathway?的主要内容,如果未能解决你的问题,请参考以下文章