读取mhdraw图像并切片归一化保存
Posted friedrichor
tags:
篇首语:本文由小常识网(cha138.com)小编为大家整理,主要介绍了读取mhdraw图像并切片归一化保存相关的知识,希望对你有一定的参考价值。
前言
raw 存储的是一个3D的CT影像,mhd 文件存储的是相应 raw 文件的信息
我的数据集train/low dose文件夹下内容如下,mhd 和 raw同在一个文件夹下:
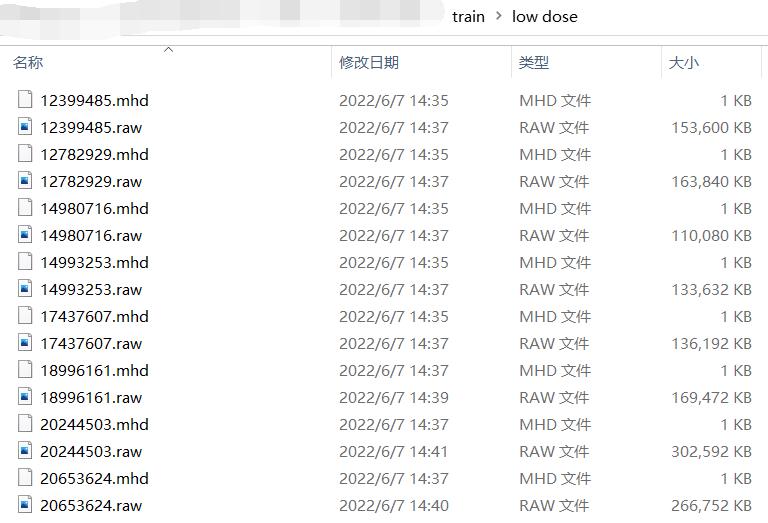
读取mhd、raw并切片
使用 SimpleITK 库进行mhd和raw文件的读取
import os
from tqdm import tqdm
import SimpleITK as sitk
import matplotlib.pyplot as plt
def slice(ori_path: str, pro_path: str):
for path in os.listdir(ori_path):
if path.find('mhd') >= 0:
data_mhd = sitk.ReadImage(os.path.join(ori_path, path))
spacing = data_mhd.GetSpacing()
scan = sitk.GetArrayFromImage(data_mhd)
for i in tqdm(range(len(scan))):
plt.figure()
plt.imshow(scan[i], cmap=plt.cm.bone)
plt.show()
break # 这里只是演示切片后效果,所以用了break,正常遍历的话直接删掉break就行
调用:
low_dose_path = 'train/low dose' # 原数据集路径
low_dose_slice_path = 'train_slice/low_dose_slice' # 保存路径
slice(low_dose_path, low_dose_slice_path)
图片演示示例:

切片、归一化、保存
def slice(ori_path: str, pro_path: str):
id = 0
for path in os.listdir(ori_path):
if path.find('mhd') >= 0:
id += 1
save_content = os.path.join(pro_path, str(id))
if os.path.exists(save_content):
shutil.rmtree(save_content)
os.makedirs(save_content)
data_mhd = sitk.ReadImage(os.path.join(ori_path, path))
spacing = data_mhd.GetSpacing()
scan = sitk.GetArrayFromImage(data_mhd)
for i in tqdm(range(len(scan))):
img = cv2.normalize(scan[i], None, 0, 255, cv2.NORM_MINMAX, cv2.CV_8U)
img = Image.fromarray(img)
save_path = os.path.join(save_content, f'id_i.png')
img.save(save_path)
说明:
我的数据集中3D影像的像素值是 -3000 到 3000 多,而我们正常的图像都是0-255的,因此最好的办法就是使用cv2.normalize归一化到0-255之间,然后再保存。
如果是自己写一个归一化的代码,或者使用from sklearn.preprocessing import minmax_scale 的这个minmax_scale函数,会出现一些奇怪的问题,我猜测可能是这样归一化时的损失很大,所以会出现一些问题。
调用同上:
low_dose_path = 'train/low dose' # 原数据集路径
low_dose_slice_path = 'train_slice/low_dose_slice' # 保存路径
slice(low_dose_path, low_dose_slice_path)
保存效果:


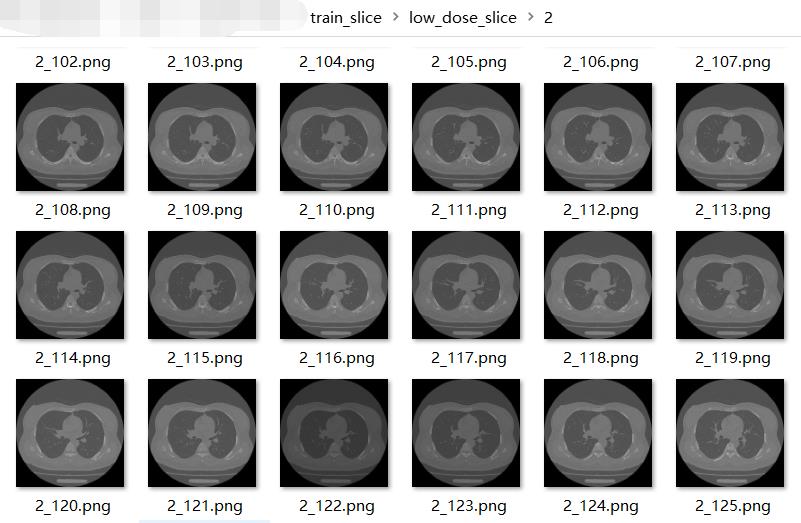
以上是关于读取mhdraw图像并切片归一化保存的主要内容,如果未能解决你的问题,请参考以下文章
Python遥感图像处理应用篇(十五):GDAL 读取多光谱数据做归一化处理
数据预处理中归一化(Normalization)与损失函数中正则化(Regularization)解惑
Python遥感图像处理应用篇(十六):GDAL 将归一化处理csv数据转化为遥感影像
Python遥感图像处理应用篇(十六):GDAL 将归一化处理csv数据转化为遥感影像