基因家族分析(2) ggplot2绘制motif分析图
Posted
tags:
篇首语:本文由小常识网(cha138.com)小编为大家整理,主要介绍了基因家族分析(2) ggplot2绘制motif分析图相关的知识,希望对你有一定的参考价值。
参考技术A meme官网( https://meme-suite.org/meme/tools/meme )上传我们的数据进行分析即可;由于我习惯使用本地版 meme ,如果您也喜欢本地版,可以运行以下命令进行分析:(此处使用的为氨基酸序列)快来看看如何使用R语言绘制一张漂亮的火山图
火山图(Volcano Plot)是差异基因可视化非常好的工具,本文将向大家展示如何用R语言来实现火山图的绘制
在对基因芯片数据、测序数据进行差异表达分析之后,我们通常会选择绘制火山图(Volcano Plot)来对各基因的表达情况进行可视化,正如下图所示:
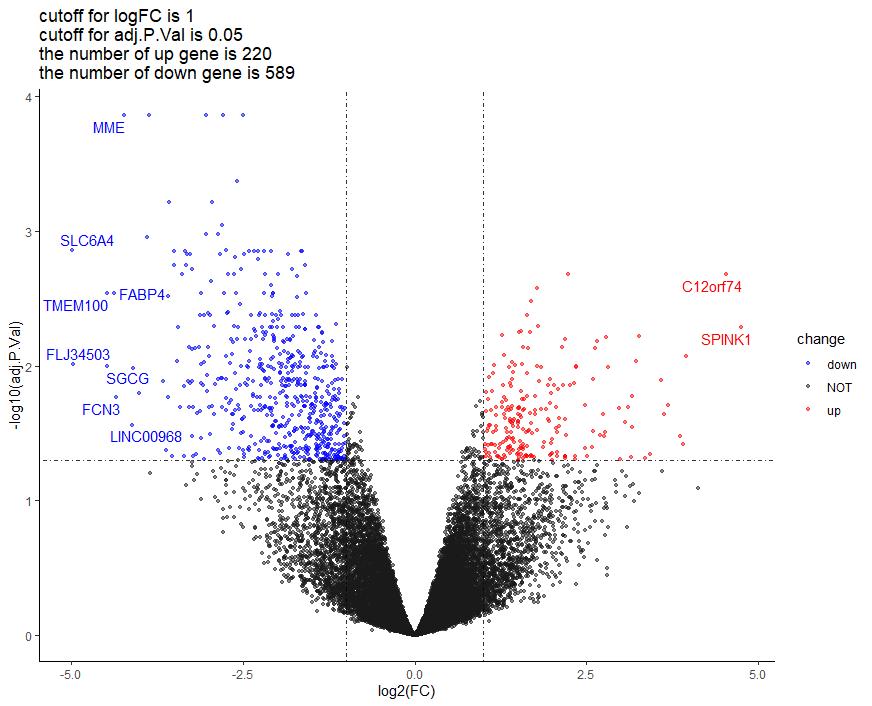
 。
。
本篇推文,我就来教大家如何使用R语言绘制如上图所示的火山图,同时为了发扬偷懒的精神,方便日后使用,我最后还把绘图过程封装成了一个函数,这样再次使用的时候只用输入数据、简单调整参数,就可以很方便地得到一张好看的火山图啦 !
!

好,废话不多说,我们这就开始吧

 !
!

首先准备绘图所需数据,我们需要一个数据框,里面包含了每个基因的表达变化情况,如下图所示:
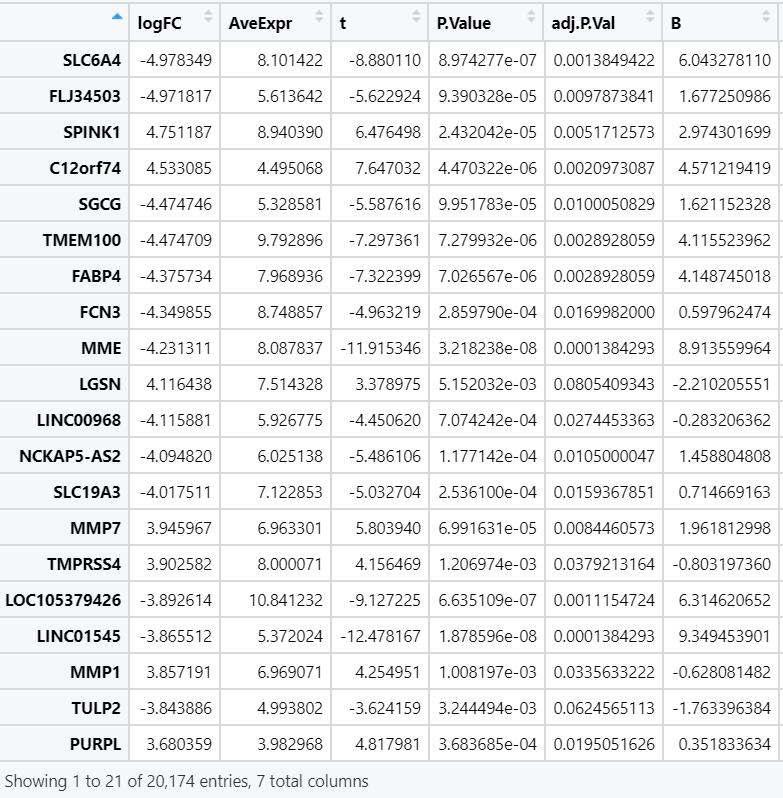
(此为数据集GSE118370经limma包分析的结果:Tumor vs. Normal,命名为DEG)
我们还需要定义一下上下调基因:以logFC>1且adj.P.Val<0.05为显著上调基因,logFC<-1且adj.P.Val<0.05为显著下调基因,其余为表达变化不显著的基因,并在DEG中新增一列,命名为Change(logFC代表对变化倍数进行log2转换,adj.P.Val为校正后的P值,筛选标准更为严格)。
##给DEG添加一列,标注各基因上下调情况
DEG$Change <- as.factor(ifelse(DEG$adj.P.Val < 0.05 & abs(DEG$logFC) > 1,
ifelse(DEG$logFC > 1, 'Up', 'Down'),'NOT'))加上Change这一列后,就可以开始绘图了。
library(ggplot2)
ggplot(data = DEG, aes(x = logFC, y = -log10(adj.P.Val),colour = Change))+
geom_point(alpha = 0.5)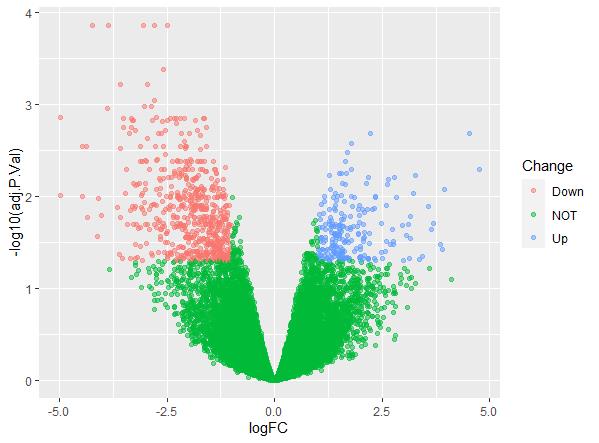
怎么样,最简单版的火山图就绘制好了 ,接下来就是对图片元素进行修饰,例如加上基因的名字、添加水平竖直分割线、调整背景等,具体的修饰过程参见下述代码:
,接下来就是对图片元素进行修饰,例如加上基因的名字、添加水平竖直分割线、调整背景等,具体的修饰过程参见下述代码:
##ggrepel包用来给散点添加标签
library(ggrepel)
##为了给散点添加标签,我们还需要给DEG再加上一列,为基因的名称(其实就是每行的名字)
##把DEG按logFC绝对值从大到小排序
DEG <- DEG[order(abs(DEG$logFC),decreasing = T),]
##新增加一列,为基因的名字
DEG$anno_name <- rownames(DEG)
##只取表达变化前10的基因进行标注,所以其余的都设置为NA
DEG$anno_name[11:nrow(DEG)] <- NA
##设置一个标题
title <- paste0('cutoff for logFC is ',1,
'
cutoff for adj.P.Val is ',0.05,
'
the number of up gene is ',nrow(DEG[DEG$Change=='Up',]),
'
the number of down gene is ',nrow(DEG[DEG$Change=='Down',]))
##现在开始绘图
ggplot(data = DEG, aes(x = logFC, y = -log10(adj.P.Val),colour = Change))+
geom_point(alpha = 0.5)+
scale_colour_manual(values = c('blue','grey10','red'))+ #自定义定义散点颜色
geom_text_repel(aes(label=anno_name),show.legend = F,segment.colour = 'black')+ #给散点添加标签
xlab('log2(FC)')+ylab('-log10(adj.P.Val)')+ggtitle(title)+ #设置坐标轴及主标题
geom_hline(yintercept = -log10(0.05),lty=4,lwd=0.6,alpha=0.8)+ #设置水平分割线
geom_vline(xintercept = c(1,-1),lty=4,lwd=0.6,alpha=0.8)+ #设置竖直分割线
theme_bw()+theme(panel.border = element_blank(), #设置背景
panel.grid.major = element_blank(),
panel.grid.minor = element_blank(),
axis.line = element_line(colour = "black"))这样,最开始那张好看的火山图就绘制完成了 ,大家可以根据自己的喜好对图中元素进行修改
,大家可以根据自己的喜好对图中元素进行修改 。最后放一个大招
。最后放一个大招 :把整个绘图过程定义成一个函数,这样我们运行函数之后就只用短短一句话就可以生成火山图啦
:把整个绘图过程定义成一个函数,这样我们运行函数之后就只用短短一句话就可以生成火山图啦 !
!
plot_vocalno <- function(DEG,logfccutoff=1,pcutoff=0.05,show.number=10,p=c('adj.p','p')){
##加载绘图所需包
library(ggrepel);library(ggplot2)
##根据输入选择P.Value或者adj.P.Val
if(p[1]=='p'){p <- 'P.Value'} else {
if(p[1]=='adj.p'){p <- 'adj.P.Val'} else {
print('请输入正确的p!');break}}
#Change列要根据设置的cutoff阈值而定
DEG$Change = as.factor(ifelse(DEG[, colnames(DEG) %in% p[1]] < pcutoff & abs(DEG$logFC) > logfccutoff,
ifelse(DEG$logFC > logfccutoff, 'Up', 'Down'),'NOT'))
##还要规定一下show.number值
show.number <- floor(show.number)
##设置后续画图的标签列,以下代码是为了避免那些logFC符合但p值不符合的
if(show.number >= 1){
DEG$anno_name <- rownames(DEG) #把行名设为打标签的一列
DEG <- DEG[order(abs(DEG$logFC),decreasing = T),] #按logFC绝对值从大到小进行排序
k <- 0
for(i in 1:nrow(DEG)){
if(DEG$Change[i]=='NOT') {DEG$anno_name[i] <- NA} else {k <- k+1}
if(k==show.number) break
}
DEG$anno_name[{i+1} : nrow(DEG)] <- NA #只要前面的的show.number个,剩下的都为NA
} else {
DEG$anno_name <- NA #当show.number为0时,整列都设为NA
}
##设置标题
title <- paste0('cutoff for logFC is ',logfccutoff,
'
cutoff for ',p,' is ',pcutoff,
'
the number of up gene is ',nrow(DEG[DEG$Change=='Up',]),
'
the number of down gene is ',nrow(DEG[DEG$Change=='Down',]))
##自定义颜色(因为有时候没有Up或者Down,所以要选择一下)
manualcolor <- c('blue','grey10','red')
if(sum(DEG$Change %in% 'Up')==0) manualcolor <- c('blue','grey10')
if(sum(DEG$Change %in% 'Down')==0) manualcolor <- c('grey10','red')
if(sum(DEG$Change %in% 'Up')==0 & sum(DEG$Change %in% 'Down')==0) manualcolor <- c('grey10')
##根据p的选择,要选择一下纵坐标轴的映射列
if(p=='P.Value'){
plot_tmp <- ggplot(data = DEG,aes(x=logFC,y=-log10(P.Value),colour=Change))
} else {
plot_tmp <- ggplot(data = DEG,aes(x=logFC,y=-log10(adj.P.Val),colour=Change))
}
##开始绘图
plot_tmp+
geom_point(alpha=.5,size=1)+ #画散点
scale_colour_manual(values = manualcolor)+ #自定义定义散点颜色
geom_text_repel(aes(label=anno_name),show.legend = F,segment.colour = 'black')+ #给散点添加标签
xlab('log2(FC)')+ylab(paste0('-log10(',p,')'))+ggtitle(title)+ #设置坐标轴及主标题
geom_hline(yintercept = -log10(pcutoff),lty=4,lwd=0.6,alpha=0.8)+ #设置水平分割线
geom_vline(xintercept = c(logfccutoff,-logfccutoff),lty=4,lwd=0.6,alpha=0.8)+ #设置竖直分割线
theme_bw()+theme(panel.border = element_blank(), #设置背景
panel.grid.major = element_blank(),
panel.grid.minor = element_blank(),
axis.line = element_line(colour = "black"))
}参数解释:
DEG:差异表达分析结果,格式如前图;
logfccutoff:即为logFC的筛选阈值,默认为1;
pcutoff:即为P值的筛选阈值,默认为0.05;
show.number:设置散点标签的个数;
p:设置选择adj.P.Val还是P.Value,默认选择adj.P.Val。
(如发现代码有错误之处或不足,欢迎指正!)
—END—
以上是关于基因家族分析(2) ggplot2绘制motif分析图的主要内容,如果未能解决你的问题,请参考以下文章