一、nt和nr数据库
nt库和nr库大家都比较熟悉,一个核酸库,一个蛋白库,两者既可以通过NCBI进行在线BLAST,也可以在ftp://ftp.ncbi.nih.gov/blast/db地址中将如下文件下载后,进行


本地BLAST。在此还是简单说明一下在线比对方法:
打开https://blast.ncbi.nlm.nih.gov/Blast.cgi,根据下表选择合适的程序(图表来自网络)

然后可以直接进行序列与nt和nr库的比对,如果还有疑问,可以查看帮助文档:
ftp://ftp.ncbi.nlm.nih.gov/pub/factsheets/HowTo_BLASTGuide.pdf。
对于这两个数据库不再赘言,如果是大批量比对,我们欧易可以提供优质的服务。
二、Swissprot数据库
Swissprot(http://www.uniprot.org/uniprot/?query=*&fil=reviewed%3Ayes),最新更新版本包含554515条蛋白序列,正如uniptort所说:“It is a high quality annotated and non-redundant protein sequence database, which brings together experimental results, computed features and scientific conclusions.”这些序列都是经过验证和注释的蛋白序列,可信度较高。
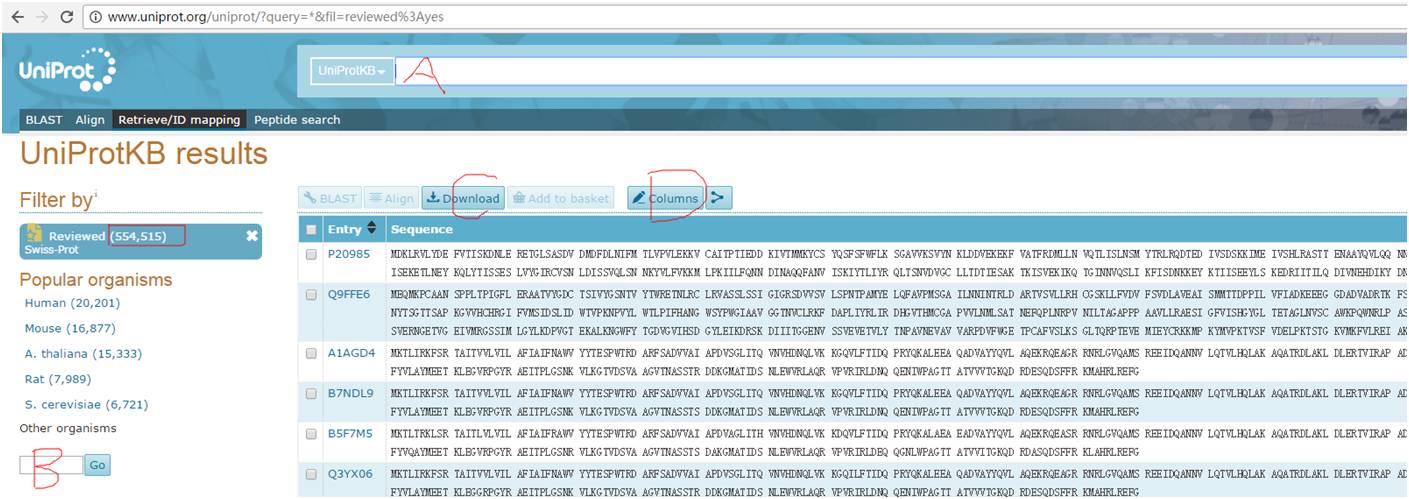
可以看到,在这个网址的左侧列出了常见物种,如果你要寻找某一特定物种的蛋白序列信息,可以在B处输入直接查找,也可以在A中输入查找。在工作中经常遇到用uniprot蛋白作为蛋白库来进行分析的itraq项目(对于转录组来说,这属于拓展知识),对于某些关注的蛋白,如果想要了解该蛋白的具体注释信息,那么可以通过如下方法来实现:
登录http://www.uniprot.org/uploadlists/或者点击网站首页
会跳转到如下网页
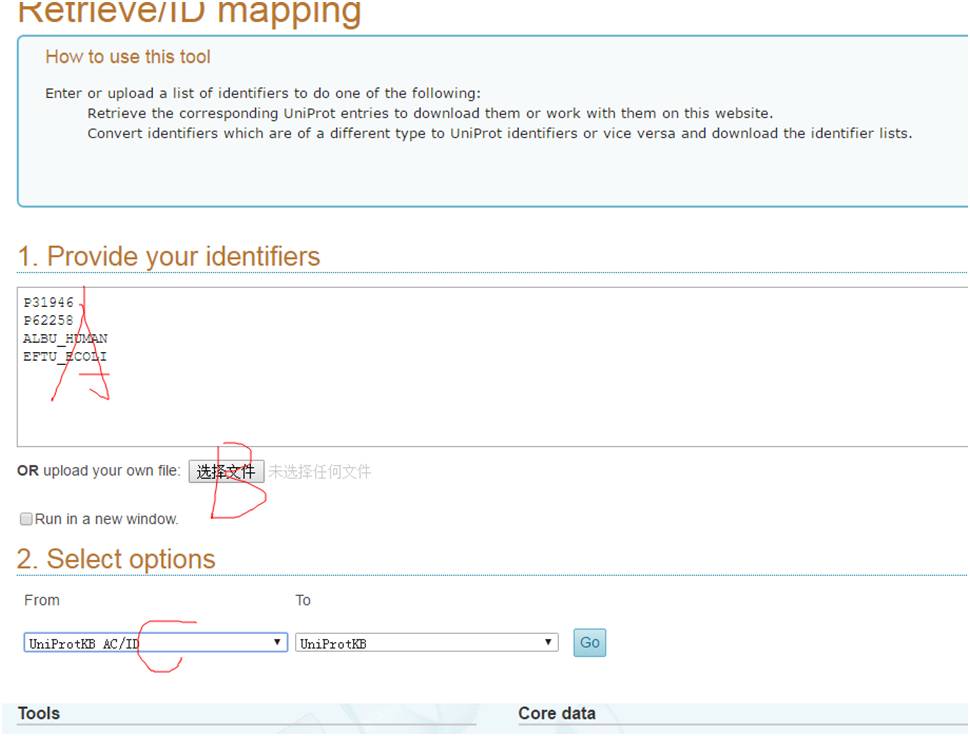
在A处输入UniprotKB AC或者ID号,或者直接在B处导入列表(每行一个),按照默认即可进行下一步。当然有的老师会说什么是Uniprot KB或者ID号,一个完整的swissprot ID是这样的
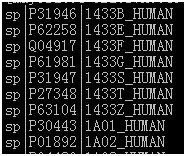
一般来说,sp代表该蛋白来自swissprot,||中间部分为ID号,也成为Entry号,|后面的一般格式是gene name缩写加上“_”跟物种名。那么使用ID号或者|后面部分都可以搜索到对应信息的。顺便一提,点开
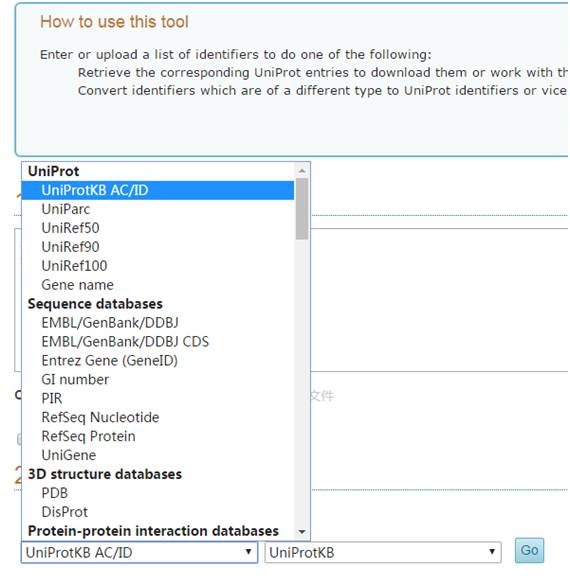
可以看到,它支持很多输入格式,对于最常用的Gene name也是可以的,不过ID号是唯一的,可以精准搜索蛋白信息。
当然点击右边
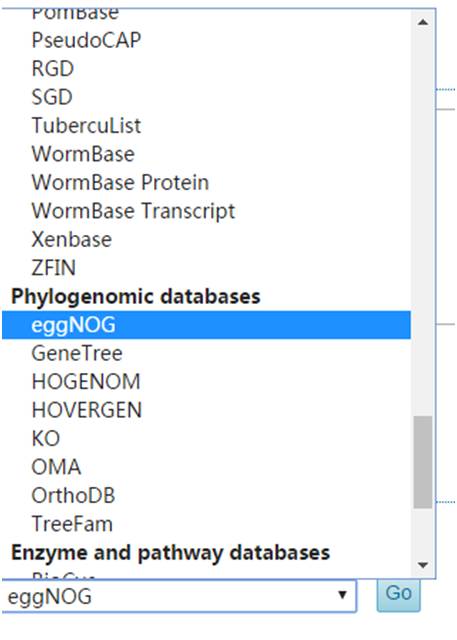
也可以指定比对到的数据库,可以根据需要查询相关注释信息。
点击Go之后会弹出如下界面:
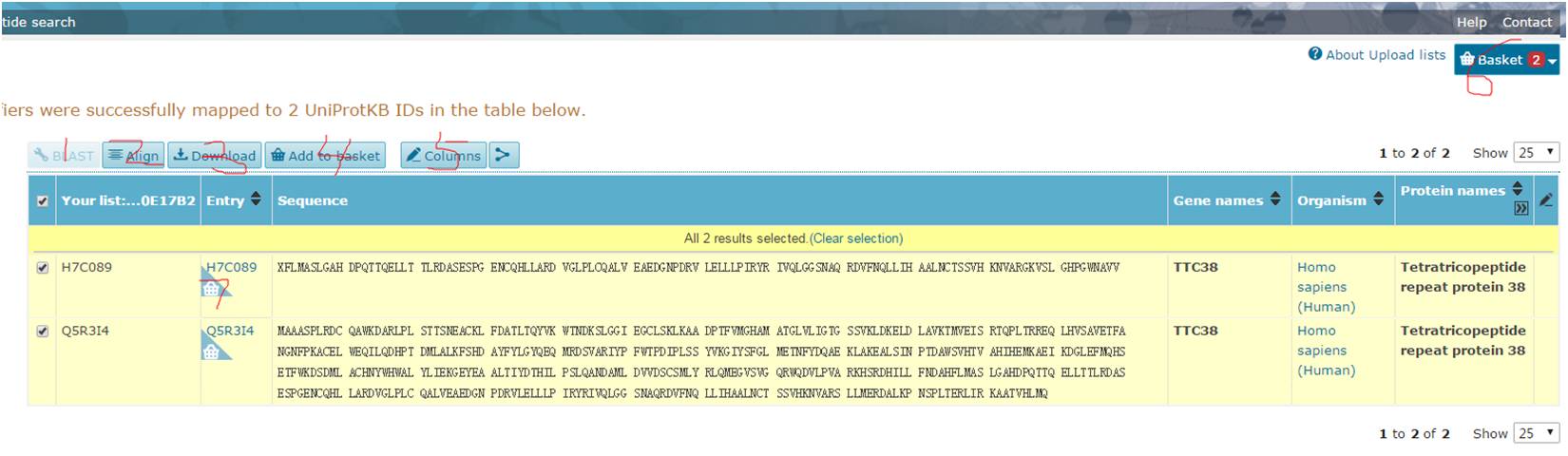
当你勾选一条序列的时候,1的BLAST会点亮,你可以将勾选的序列和uniprot的序列进行BLAST比对。当你勾选多条序列的时候,2的Align会点亮,你可以进行勾选序列之间的alignment,3是下载所有信息或者勾选信息,4是将勾选的蛋白(会相应的在左下角加上篮子标志如7处显示)加入basket(篮子),加入的蛋白会在右上角6处显示,点击6可以进行针对收藏的序列进行一些指定分析,在此不再赘述。
需要着重说的是5Columns选项,点击它,你便发现了新天地。
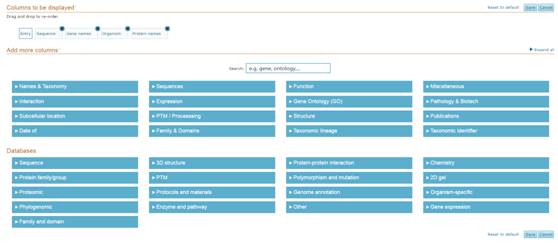
理论上说,你能想到的常用注释数据库信息,都可以在其中找到,包括GO,kegg,序列信息,蛋白名,基因名,亚细胞定位,pfam信息等。因为这里包含的信息极为庞杂,所以没有一一列举,只要勾选其中的选项,然后点击右上角的save,便可以将这些信息收入囊中。
在一般转录组分析中,swissprot的注释率是仅低于NR的,依靠swissprot的注释,其实可以扩展得到很多其他相关注释,例如GO,KEGG,pfam等。
三、KEGG数据库
KEGG数据库应该也是大家比较熟悉的,在此也不做过多赘述,可以参考
http://muchong.com/html/201009/2325769.html 来更加深入的了解KEGG数据库,在这里,仅针对老师一些序列的kegg注释为老师提供一个在线提交的方法:
1. 打开网址 http://www.genome.jp/kaas-bin/kaas_main 进行如下操作:
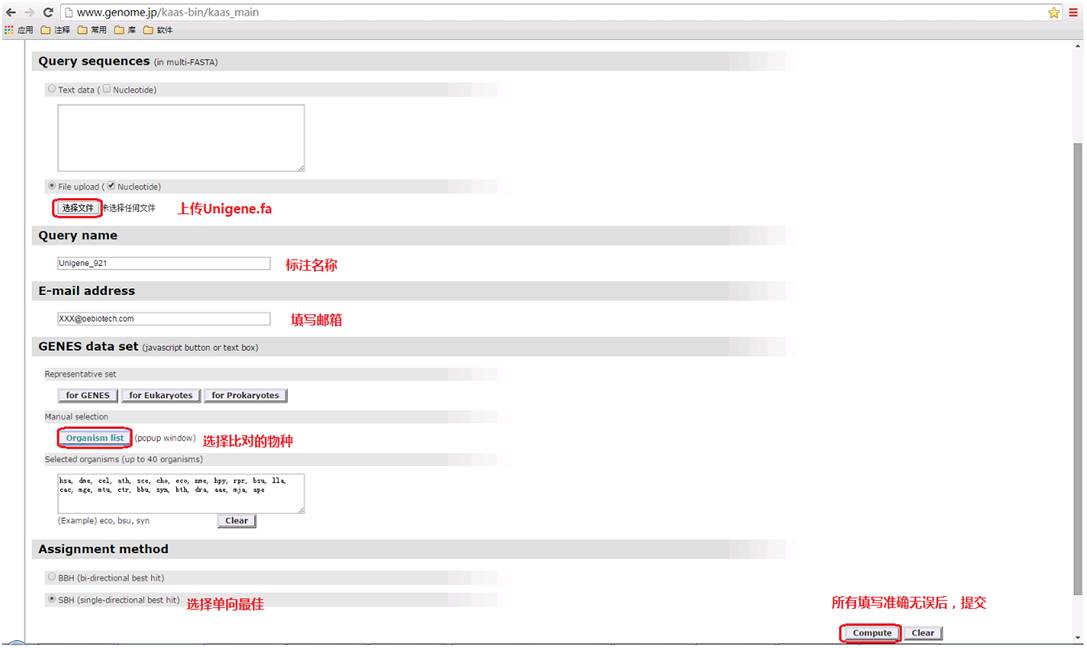
2. 待任务上传完成,邮箱会收到一份邮件告知任务被受理:
3. 按照邮件提示操作即可,完成后会收到一封邮件。
4. 打开邮件中链接,点击对应任务的 html:
5.选择 brite hierarchies:
6. 选择 KEGG Orthology(KO)
7. 选择Download htext将文件下载到本地,文件名保持默认(q00001.keg)
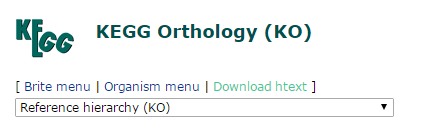
这样就会得到序列的KEGG注释结果了,一般来说,对于1万条左右序列,1-6小时即可完成。
四、KOG数据库
“KOG”是Clusters of orthologous groups for eukaryotic complete genomes(真核生物蛋白相邻类的聚簇)的缩写。构成每个KOG的蛋白都是被假定为来自于一个祖先蛋白,并且因此或者是orthologs或者是paralogs。Orthologs是指来自于不同物种的由垂直家系(物种形成)进化而来的蛋白,并且典型的保留与原始蛋白有相同的功能。Paralogs是那些在一定物种中的来源于基因复制的蛋白,可能会进化出新的与原来有关的功能。数据库链接:ftp://ftp.ncbi.nih.gov/pub/COG/KOG/kyva。
该数据库,目前没有在线提交的注释方法,但是如果你仔细看上文的话,会发现其实可以通过swissprot来获得KOG注释信息,没有发现的话,请回头再仔细看看。
五、string数据库
STRING数据库(https://string-db.org/)是一个搜寻已知蛋白质之间和预测蛋白质之间相互作用关系的系统。这种作用关系既可以是蛋白质之间的物理作用,也可以是间接的功能相关性。它基于染色体临近、系统进化谱、基因融合和基因芯片数据等生物学信息来计算基因或者蛋白的共表达。
最新的string数据库为10.5版本,包含2031个物种9‘643‘763种蛋白1‘380‘838‘440种相互作用关系。您可以通过下载收录物种蛋白序列的方法进行本地blast比对,点击download
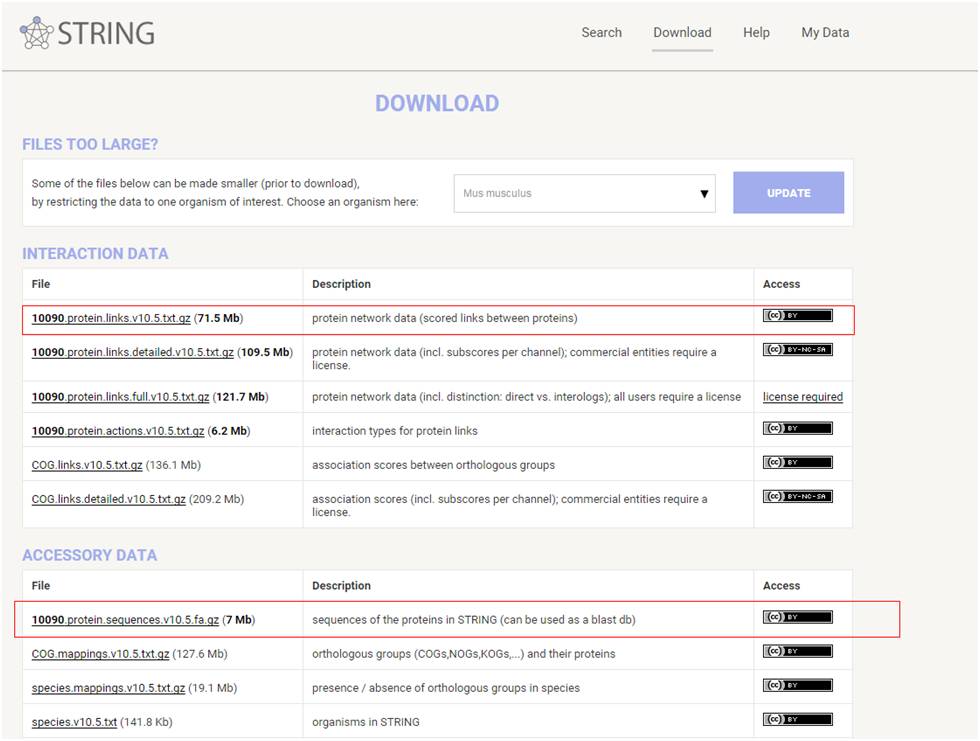
选定物种后下载作用关系文件*.protein.links.v10.5.txt.gz 和*.protein.sequences.v10.5.fa.gz
文件即可。
对于在线比对,string使用起来还是非常方便的,如下图
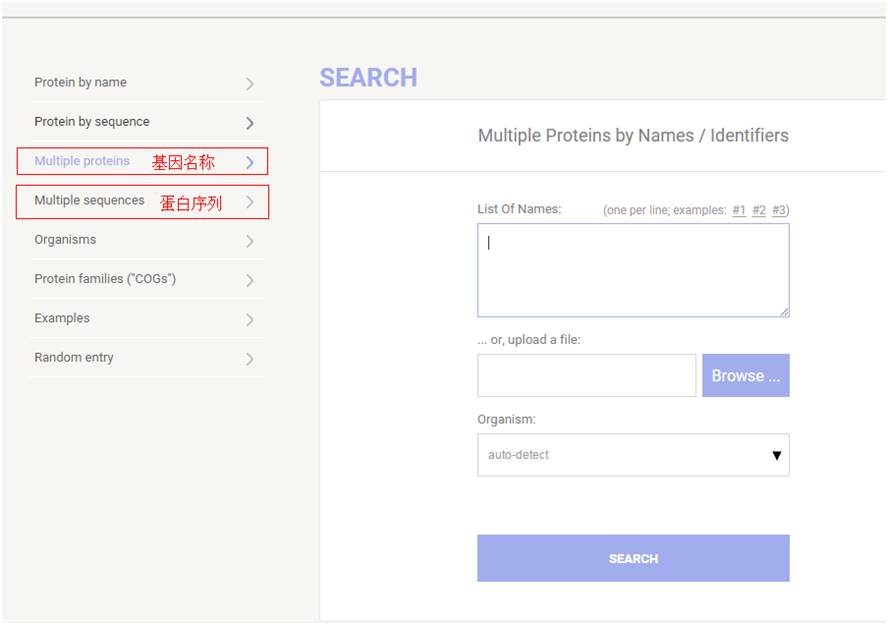
可以使用基因名或者蛋白序列进行查询,蛋白序列查询方法我们在此不多做说明,基因名查询的方法对我们来更为常用,以人为例,输入gene symbol(一行一个),如下图,点击search
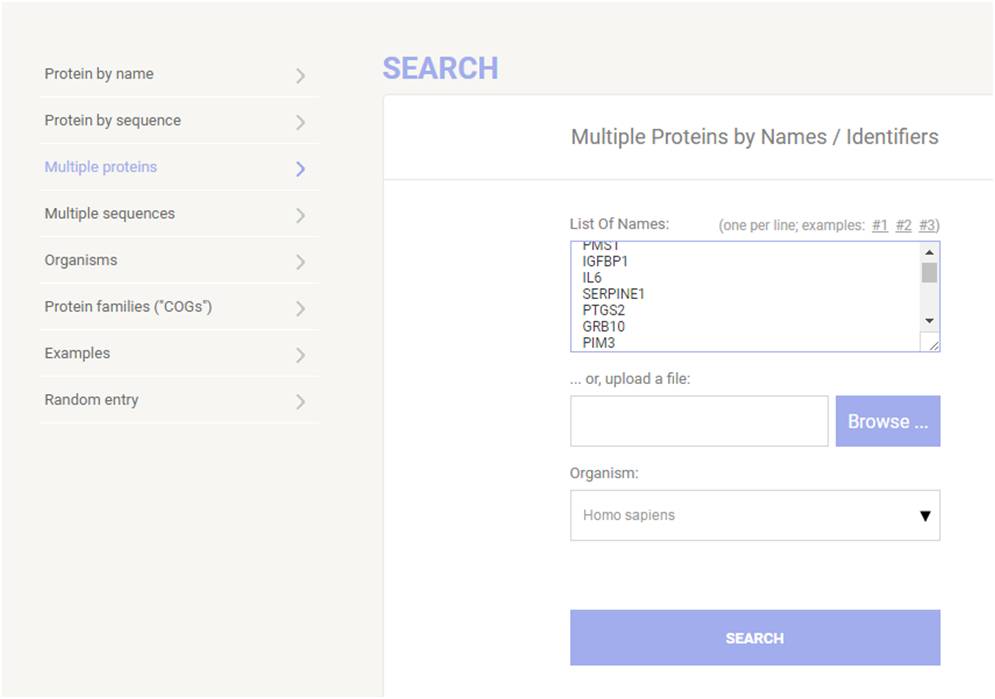
然后会弹出如下网页
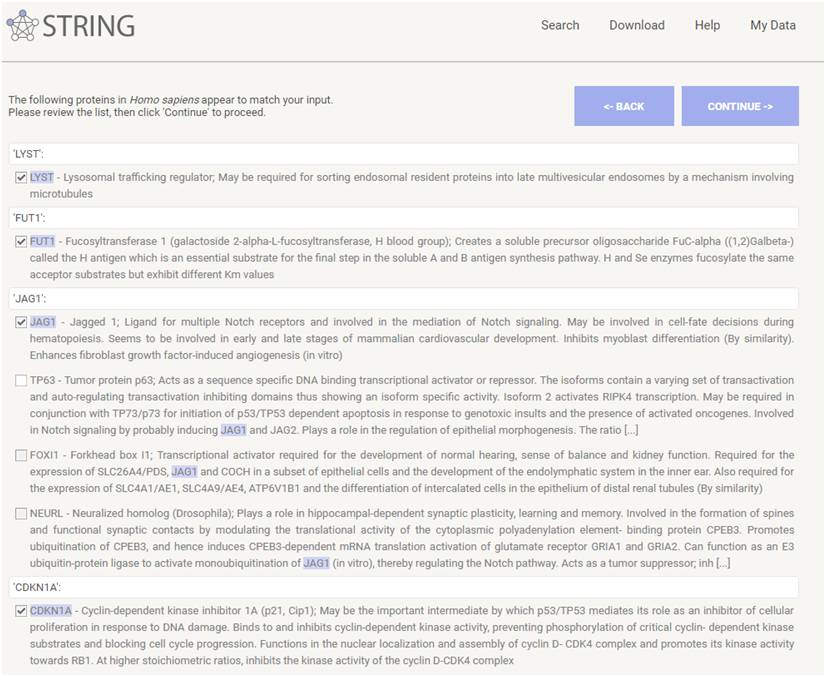
String网站会将输入的gene name与数据库中该物种的基因进行匹配,输出匹配最好的一条画钩,一般来说,在这里可以检查一下是否匹配正确,绝大多数情况下,是没有问题的,有些时候输入的基因名和收录的基因名可能稍有出入,实际输出的时候,是以sring网站为准。确认无误后,点击CONTINUE(输入越多,这一步越慢,后面的图和表也越复杂,因此不建议输入过多基因)。
在弹出的如下网页中,上半部分是蛋白互作图,中间是一些设置参数等,如下图:
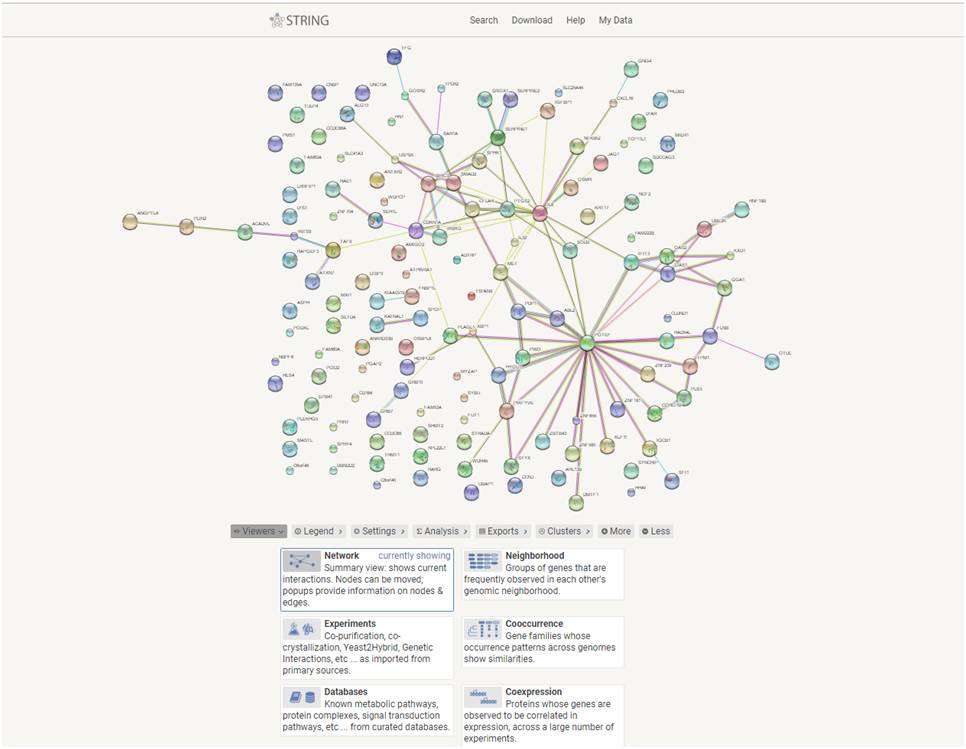
对于上图的互作关系图片,可以直接使用,也可以导出数据之后用Cytoscape自己绘图。
是以不同的方法去查看结果,一般默认是Network,对于其他感兴趣的可以点击查看一下。
是一些说明,包括图标说明,线条说明以及输入说明。
是进行一些设置,比如用互作关系或者可信度展示线条,图片保存格式(png或者svg矢量图),以及最低可信度等,如图所示使用0.4会显示所有打分值在0.4之上的nodes,最高可以设置为0.9以减少低可信度nodes,也可以是图片使加简单美观。
可以对这些输入基因进行GO和KEGG注释及富集,并输出结果。
可以将结果文件输出,包括图片和表格信息,如前面所说如果需要自己用 Cytoscape绘图的话可以点击TSV输出格式,得到互作用excel表格。
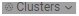
是对输入进行聚类,其实如果用Cytoscape也是可以进行聚类的,使用的是MCODE,可以查看小编上一篇文章详细了解。如下图所示,是对TSV输出结果进行绘图并聚类的结果,不同的cluster用不同的颜色显示。
关于string还有一个文件想再说明一下,就是download中的species.v10.5.txt 文件,该文件是以taxon_id进行排序的,大家可以在其中查找相应物种进行检索,到这里,关于string的介绍就结束了。
六、AnimalTFDB
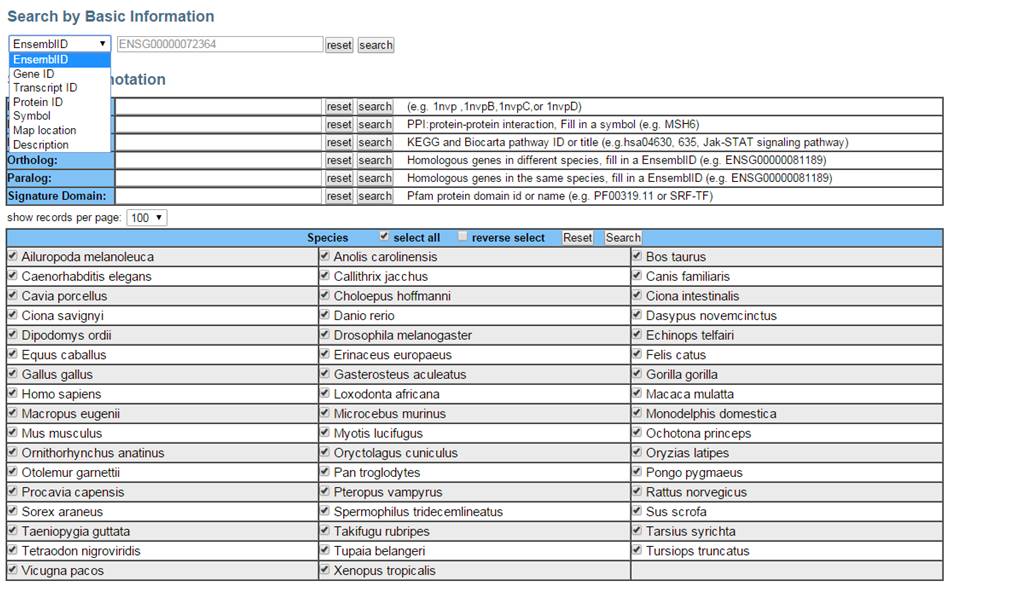
AnimalTFDB(http://www.bioguo.org/AnimalTFDB/)是一个动物转录因子数据库,该数据库收录了大部分动物模式物种,包括人,猪,蟾蜍、果蝇等50个物种的71个转录因子家族,他们的注释信息是基于ensemble 6.0 (ftp://ftp.ensembl.org/pub/release-60/gtf/),所以也是可以下载数据进行本地blast比对的。该数据库支持多种输入格式的数据检索,如下图
七、PlnTFDB
PlnTFDB(http://plntfdb.bio.uni-potsdam.de/v3.0/)是一个植物转录因子数据库,该数据库收录了大部分植物模式物种,包括拟南芥,水稻等20个物种的84个转录因子家族,包含28193 protein models, 26184 distinct protein sequences。它支持在线blast进行比对,也可以将数据下载后进行本地blast。
八、PRGDB
PRGDB(http://prgdb.crg.eu/wiki/Resistance_genes)是一个植物抗性基因数据库,它也支持本地和在线BLAST比对,关于它能介绍的不多,在此简单提一下。