FusionCancer-人类癌症相关的融合基因的数据库
Posted 庐州月光
tags:
篇首语:本文由小常识网(cha138.com)小编为大家整理,主要介绍了FusionCancer-人类癌症相关的融合基因的数据库相关的知识,希望对你有一定的参考价值。
RNA-seq 测序可以用于融合基因的发现,在过去的十几年里,RNA-seq 测序数据不断增加,发现的融合基因的数据也不断增加;
FusionCancer 是一个人类癌症相关的融合基因的数据库,利用NCBI SRA数据库中的RNA-seq 数据,采用tophat-fusion, soap-fusion, fusionmap, chimerascan 4款预测融合基因的软件进行预测;
网址如下:
http://donglab.ecnu.edu.cn/databases/FusionCancer/
点击 Borwse DB 按钮,首先浏览下数据库中的记录
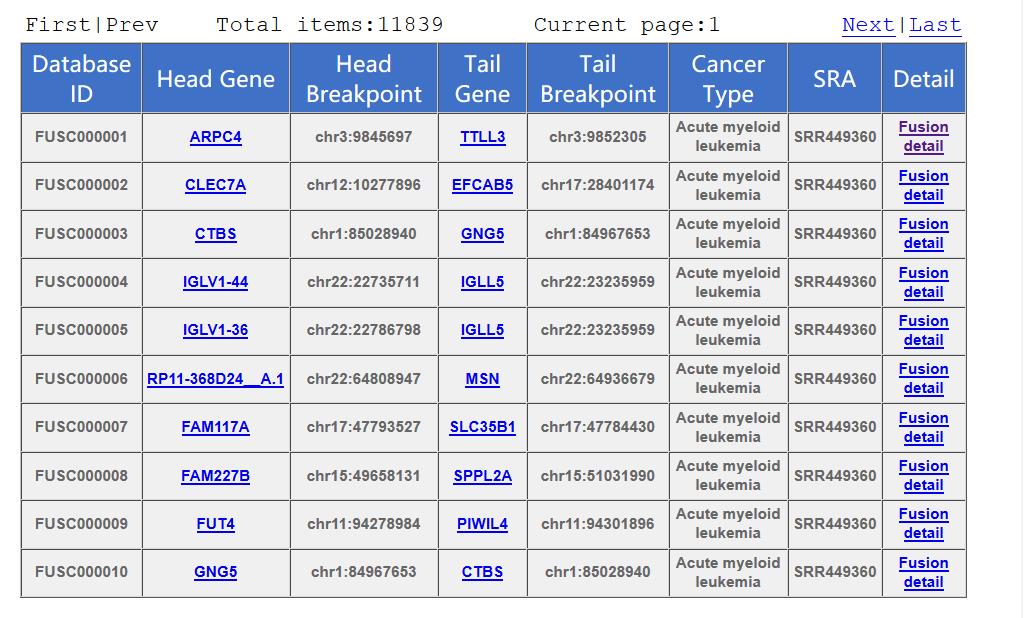
DataBase ID : 对于每条融合基因的记录,都会给出一个唯一的ID,以FUSC开头,后面加数字;
Head Gene : 生成融合基因的第一个的基因
Head Breakpoint : 融合基因的连接点在第一个基因上的位置
Tail Gene : 生成融合基因的第二个基因
Tail Breakpoint : 融合基因的连接点在第二个基因上的位置
CanCer Type: 对应的癌症类型
SRA : 原始数据在SRA 数据库中的编号
Detail : 融合基因的详细信息;
以第一条为例,点击Fusion detail,详细信息如下
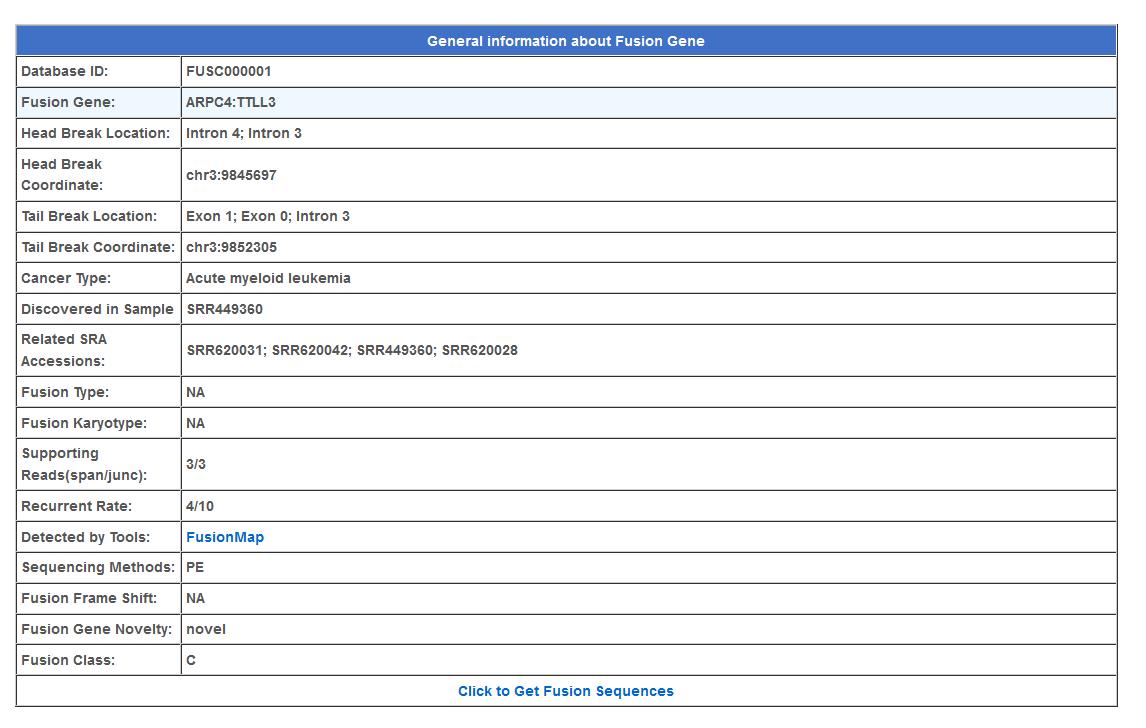
在详细信息界面中,还会给出breakpoint 在基因的内含子区还是外显子区,预测到该融合基因的 软件;支持的Reads 数,
点击最下方的链接,还可以得到融合基因的序列。对于上述的融合基因,其转录本序列如下:
对于融合基因而言,最直接的结果只是检测到了两个基因的连接处,breakpoint , 其实是不能够直接确定融合转录本的全长序列的,这个是二代测序相比三代测序的一个劣势;
这里给出的序列其实是两个基因所有转录本的组合,可以看到断点都是一样的



总结:
对于融合基因的检测而言,二代测序存在以下缺点:
1)只能给出有无该融合基因,而且检测到的融合基因还存在假阳性;
2)没有具体的融合转录本的序列,没有融合基因的直接证据;
三代测序由于其读长的优势,在融合基因的检测上有着先天的优势;
FusionCancer 数据库可以看做二代测序在融合基因检测方面的集大成之作,首先用了4款软件进行预测,通过数据库,可以筛选出4种软件都预测得到的融合基因,可以降低假阳性率;其次给出了可能的融合转录本序列,方面后续的RT-PCR验证;
以上是关于FusionCancer-人类癌症相关的融合基因的数据库的主要内容,如果未能解决你的问题,请参考以下文章