Feature Selection 其一 —— Filter Approach
Posted yuki_lee
tags:
篇首语:本文由小常识网(cha138.com)小编为大家整理,主要介绍了Feature Selection 其一 —— Filter Approach相关的知识,希望对你有一定的参考价值。
这一个部分都将只涉及到选择特征的某个子集的方法,将高纬度的特征空间映射到低维度空间的方法(如PCA)都不会涉及到。
一. 单变量
优点:运算速度快,独立于分类器
缺点:忽略的特征之间的联系,忽略了与分类器的联系(在训练模型的时候不能调参来提高性能)
1. 卡方检验
主要内容参考来自 http://blog.sina.com.cn/s/blog_6622f5c30101datu.html
卡方检验的思想是同过观察实际值与理论值的偏差来确定理论正确与否。原假设H0( null hypothesis) 假设观察值与理论值没有区别。首先假设原假设成立,基于此算出卡方值,它表示观察值与理论值的偏离程度。根据卡方分布及自由度可以确定在H0假设成立的情况下获得当前统计量及更极端情况P。如果P值很小,应当拒绝无效假设。否则就不能拒绝无效假设。
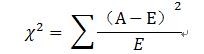
在特征选择中,我们可以假设原假设H0: 第i个特征与类别C 不相关,这样,算出的卡方值越大就说明这个特征与类别C越相关,也就是说这个特征越重要。
|
特征选择 |
属于DNA-binding protein |
不属于DNA-binding protein |
总计 |
|
包含”AA” |
A |
B |
A+B |
|
不包含”AA” |
C |
D |
C+D |
|
总数 |
A+C |
B+D |
N |
A:表示包含AA片段的DNA-binding protein的个数
B:表示包含AA片段的non DNA-binding protein的个数
C:表示不包含AA片段的DNA-binding protein的个数
D:表示不包含AA片段的non DNA-binding protein的个数
原假设:AA片段与DNA-binding protein不相关。
根据原假设,出现在DNA-bindig protein包含的AA的比例应该和所有文档中包含AA的比例相同,所以,理论值应该是:
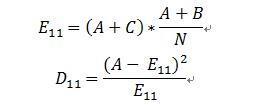
同理可以计算D12,D21,D22.
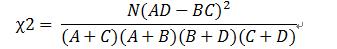
因为我们只需要相对值,所以:
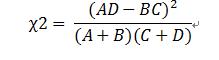
comment: 因为在计算中,并没有考虑到在一条蛋白质中某个片段出现的频率,所以一个片段某类蛋白质所有的样本中出现一次的卡方值会大于,在该类蛋白质99%的样本中出现10次的片段。这就是“低频词缺陷”。
在bioinformatics 中应用卡方检验来检验某种特征对特定类别的作用,我想是可行的。但是有个问题,是不是正样本和负样本都会包含AA这个片段呢?只是频度的区别?如果是这样,那么这种方法就不可行了。因为它并没有考虑到某一个蛋白质序列中某个特征的频度。但是我还是觉得这个方法可以研究的,需要检查一下我们组的特征提取方法,看看是否适用。
以上是关于Feature Selection 其一 —— Filter Approach的主要内容,如果未能解决你的问题,请参考以下文章