如何用深度学习进行CT影像肺结节探测(附
Posted
tags:
篇首语:本文由小常识网(cha138.com)小编为大家整理,主要介绍了如何用深度学习进行CT影像肺结节探测(附相关的知识,希望对你有一定的参考价值。
1.数据预处理首先用SimpleITK把mhd图片读入,对每个切片使用Gaussian filter然后使用阈值-600把肺部图片二值化,然后再分析该切片的面积,去掉面积小于30mm2的区域和离心率大于0.99的区域,找到3D的连通区域。
只保留0.68L到8.2L体积的区域,并且如果大于6000 mm2的区域到切片的中心区域的距离大于62mm也删除该连通区。最后只留下一个最大的连通区域。
左边是原始图,右边是切完肺的。
在实际中预处理中,我们可视化了每个肺的部分切片,存在一些bad case。主要有以下3种,我们也对这3种情况做了优化:
把肺边缘结节切掉。因为阈值导致的,把二值化环境-600改成-150有改善。
切出来全部为黑的(未找到任何肺部区域)。有些ct图是从头部开始扫描的,导致影响了连通区域判断,需要手动查看该mhd文件,看里面的从第个切片到第几个切片是肺部,在做完二值化操作后,人为把前面和后面的切片全部设置为0。
切出来只有一侧肺部情况。
有些患者两个肺的大小差别比较大,需要调整阈值,放宽阈值标注,把大于6000 mm2的区域到切片的中心区域的距离大于62mm也删除该连通区,改为大于1500 mm2的区域到切片的中心区域的距离大于92mm也删除该连通区。并且在最后一步,不只保留最大的连通区,同时保留最大的两个连通区。
2.模型网络结构
我们的网络如图所示,整体上是采用Unet+Resnet的思想。里面每个Resnet Block都是由多个卷积层和bn层和relu层组成的。我们只展示主体结构(整体深度大概150多层):
3.整体优化思路
3.1 数据优化
肺部切割优化:这块其实没有完美的方法能把所有的肺一次性都切好。具体的思路我们已经在第1章数据预处理部分写出来了:我们会先切一遍,然后将切肺中切的不好的,再调参数重新切一次。
10mm 以下结节的训练数据增强。我们在没做数据增强的情况下跑出来的模型,在验证集上漏掉了不少10mm以下的结节,所以对这部分的结节做了增强。
3.2 工业界优化思路:模型架构 > 模型网络
我们的优化思路非常的工业界,用更多的计算资源,和更复杂的模型架构,并不把大量的时间用在调模型网络上面。
3.3 层次化Hard Mining
业界两套网络的做法比较普遍,比如用Unet切割或Faster RCNN检测,用3D CNN分类,如下图所示。
我们用的是如下统一的一套模型架构,即3D Faster RCNN的RPN网络,没有后续的全连接做分类,也并没有
再在后面接一套3D CNN来做降假阳。能减少需要调节的网络参数。
该hard mining的过程,其实就是用上一层的模型作为下一层的输入,每一层的训练数据都选取比上一层更难分的。
这套架构,无需2套网络,只需要选择一套较深的网络。
根据我们的经验,采取层次化模型训练,第二层模型froc能比第一层效果提升0.05,第三层能比第二层提升0.02。
3.4 LOSS 函数的设计
在计算loss函数的时候,我们做了2点优化。
1.在使用hard mining的时候,每个batchsize里面负例的个数会明显多于正例。为了防止算loss的时候被负例主导。我们将loss函数分成3个部分,负例的loss,正例的loss和边框的loss。
2.在上一节提到的层次化hard mining,我们在最后一层训练模型的时候,会修改loss函数的计算,对于分错的负例和正例,做加权。这个思路和focal loss是很像的。
比如:
红框里面的部分,本来是负例,却以很大的概率被分成正例,这部分在算loss的时候权值就大些。红框外面的部分权值就小些。
4.本次比赛的关键点总结:
1) 解决了基于Intel extended Caffe的150多层深度网络的 3D Faster RCNN RPN网络收敛问题。
可以从2个方向来解决(线下Phi卡平台均已验证过)。
a)将 drop out设置为 0.1。缺点是会容易过拟合。
b)先训练一个crop size为32的模型
用这个模型做pre train model,训练crop size 64的模型
依次类推。
直到完成crop size为128的模型训练
由于时间关系,我们并未比较这2种思路的效果。比赛中使用的是第1个思路,收敛的更快些。
2) 提出层次化Hard Mining的训练框架。并没有采用常见的,unet做分割+3D CNN降假阳 或者 2d faster rcnn做检测+3D CNN降假阳的思路。我们只用了一套网络。减少了需要调节的网络参数。
3) 重新设计了loss函数,防止负例主导loss的计算, 并且在降低loss的过程中,更聚焦于分错的训练样本。
5. 经验总结:
我们团队虽然过往深度学习架构经验多,但对医学影像处理的know how属于尚在探索之中。所以,我们的优化思路,是用更多的计算资源,和更复杂的模型架构,来弥补没有专用模型网络积累的短板。在第一轮比赛时通过调用比较充足的计算资源时效果比较显著,但在第二轮限定计算资源的多CPU的框架上,比较受限于计算资源及时间。
在计算资源比较充沛的情况下,选取比较深的Resnet效果会明显。在资源受限的实际场合或者现实的生产环境,我们有两点启发:
学会认同重复造轮子的基础性工作。第一轮比赛我们是pytorch框架,第二轮按要求在caffe上实现,特别是在Intel Extended Caffe对3D支持有限,重写了不少很基础的模块,这种貌似重复造轮子的工作,对我们提出了更高的要求,但也锻炼了我们深入到框架底层的能力,从而对不同框架的性能特点有更深的认识,这种重写甚至还因此帮我们找到我们第一版pytorch代码里detect部分存在的一个bug。
根据资源灵活优化训练策略乃至模型。我们的3D Faster RCNN 初期在Extended Caffe 上过于耗时,但因为在计算资源充足环境下我们的做法比较有效,所以没有去考虑一些更快的检测算法,比如SSD、YOLO等,这点也算是路径依赖的教训了。 参考技术A 要提取,不过是由模型自动从大量数据中提取特征。而不是像传统的Hog,Sift一样设计特征。
医学CT图像特征提取--肺结节CT影像特征提取系统软件设计
肺结节的特征提取在临床中有着重要应用,在上篇文章已经对肺结节的基本特征和CT影像特征提取算法有了介绍,提出了三类肺结节CT影像特征提取算法。本文重点介绍肺结节CT影像特征提取系统的功能介绍及使用,利用肺结节CT影像特征提取系统对一些数据进行特征提取,检验特征提取算法的有效性。
一、肺结节特征提取算法流程

图1 算法流程图
首先,对原始的肺部CT影像数据和放射学家标记的肺结节数据进行预处理,得到标准化的肺部CT影像数据和标记数据;然后,计算得到肺结节区域,用于后续特征提取;接着针对肺结节区域做不同的处理,计算灰度直方图,提取灰度特征。构造灰度共生矩阵,用于提取肺结节的纹理特征。提取肺结节的区域和边缘数据,用于计算得到肺结节的形态特征。
二、系统模块组成
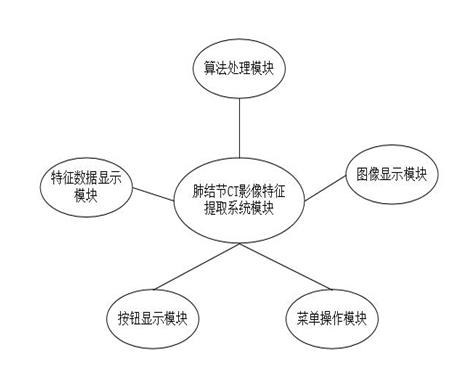
图2 系统模块组成图
三、软件运行原理
(1) 运行环境
操作系统:Windows XP,Windows7,windows 8
硬件要求:奔腾处理器,2G内存,10G硬盘
软件环境:支持matlab R2015a及以后版本
(2) 基本算法
- 对肺部CT影像进行预处理,提取出肺结节区域信息;
- 利用灰度直方图算法、灰度共生矩阵算法、几何参数算法以及Hu不变矩算法对肺结节区域进行处理,得到特征数据。
- 特征数据存储,等待导出。
四、 系统操作流程

图3 软件操作流程
五、软件程序组成
GUI主界面:lung_nodules_features_extraction.m;
灰度特征界面:get_gray_feature.m;
纹理特征界面:get_texture_feature.m;
心态特征界面:get_Hu_Moment.m
CT影像读取及预处理:read_dcm_mask.m;
Hu不变矩算法:Hu_Moments;
纹理特征算法:getGLCMtextures.m;
六、软件使用图解
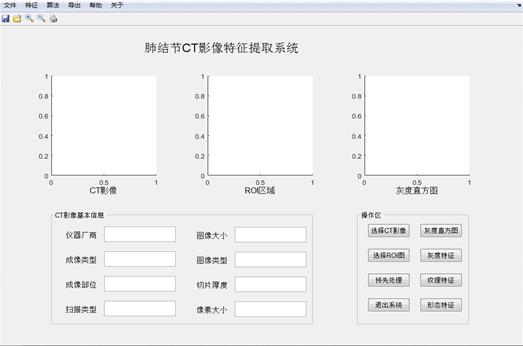
图4 系统界面示意图
系统打开后的界面如图3所示,最上面为菜单栏,上半部分三个图像显示区域,左下角是CT影像基本信息显示区域,右下角是按钮操作区。
操作流程如下:
- 在按钮区或者菜单栏选择操作读入CT影像;
- 在按钮区或者菜单栏选择操作读入ROI区域图像;
- 选择按钮或者菜单,对数据进行预处理,得到如图4所示,获得CT图像基本信息;
- 选择直方图按钮或菜单,得到灰度直方图图像,如图4上部第三幅图像所示;
- 在按钮区或者菜单栏选择灰度特征、纹理特征或者形态特征得到图5,图6,或者图7所示的界面,在特征显示界面进行操作,得到特征数据,并且可以导出特征数据为excel文件。
- 选择退出按钮,退出系统。
软件操作图示:

图5 特征提取界面

图6 灰度特征数据界面

图7 灰度特征数据界面
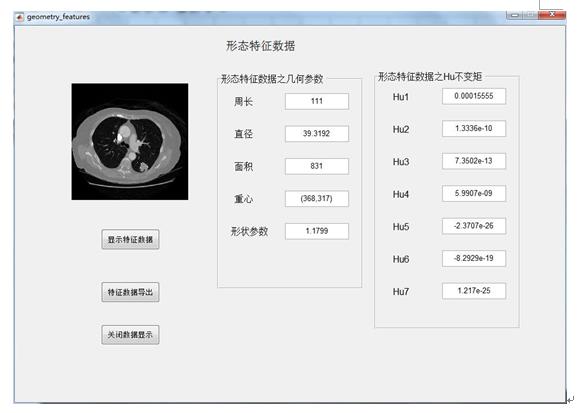
图8 灰度特征数据界面
以上是关于如何用深度学习进行CT影像肺结节探测(附的主要内容,如果未能解决你的问题,请参考以下文章