BioCodeElm格式中提取位点信息
Posted 于淼
tags:
篇首语:本文由小常识网(cha138.com)小编为大家整理,主要介绍了BioCodeElm格式中提取位点信息相关的知识,希望对你有一定的参考价值。
说明:
①Elm格式:
PLMD ID Uniprot Accession Position Type Sequence Species PMIDs
PlMD编号 Uniprot数据库编号 位点 翻译后修饰类型 序列信息 物种 PMID
PLMD-1 O00115 52 Ubiquitination MIPLLLAALLCVPAGALTC Homo sapiens 21963094;23266961
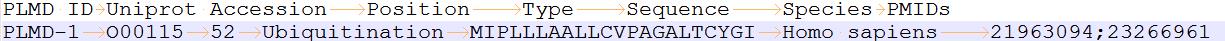
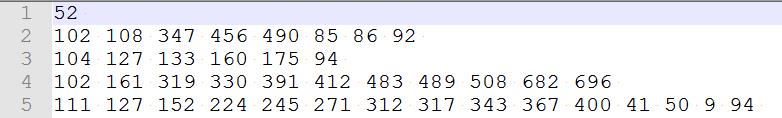
②代码说明:从上述格式中提取Position信息,当 Uniprot Accession相同时,Position位于同一行。不相同时,回车换行。每个位点之间用空格隔开。
代码:
package single; import java.io.BufferedReader; import java.io.BufferedWriter; import java.io.FileReader; import java.io.FileWriter; public class Elm_site { // 从Elm中获得位点 每一行是一个蛋白质的的位点 public void get_site(String path) { try { FileReader reader = new FileReader(path); BufferedReader br = new BufferedReader(reader); String str=null; String now="O00115"; FileWriter fileWritter = new FileWriter("E:\\\\experiment--help\\\\linglingbao\\\\site.txt"); BufferedWriter bufferWritter = new BufferedWriter(fileWritter); while((str=br.readLine())!=null){ String[] temp =str.split(" ");//每一列之间使用TAB隔开的,需要用TAB来截取 String uni_id=temp[1]; if(uni_id.equals(now)){ System.out.print(temp[2]+" ");//输出 bufferWritter.write(temp[2]+" ");//写入文件 bufferWritter.flush(); }else{ now=uni_id; System.out.print("\\n"+temp[2]+" "); bufferWritter.write("\\n"+temp[2]+" "); bufferWritter.flush(); } } br.close(); reader.close(); } catch (Exception e) { // TODO Auto-generated catch block e.printStackTrace(); } } public static void main(String[] args) { Elm_site site = new Elm_site(); String path = "E:\\\\experiment--help\\\\linglingbao\\\\ubiquitination.txt"; site.get_site(path); } }
输出结果格式:
以上是关于BioCodeElm格式中提取位点信息的主要内容,如果未能解决你的问题,请参考以下文章