ML(4.3): R Random Forest
Posted 天戈朱
tags:
篇首语:本文由小常识网(cha138.com)小编为大家整理,主要介绍了ML(4.3): R Random Forest相关的知识,希望对你有一定的参考价值。
随机森林模型是一种数据挖掘模型,常用于进行分类预测。随机森林模型包含多个树形分类器,预测结果由多个分类器投票得出。 决策树相当于一个大师,通过自己在数据集中学到的知识对于新的数据进行分类。俗话说得好,一个诸葛亮,玩不过三个臭皮匠。随机森林就是希望构建多个臭皮匠,希望最终的分类效果能够超过单个大师的一种算法。随机森林的分类效果(即错误率)与以下两个因素有关: ①森林中任意两棵树的相关性:相关性越大,错误率越大。 ②森林中每棵树的分类能力:每棵树的分类能力越强,整个森林的错误率越低。
减小特征选择个数m,树的相关性和分类能力也会相应的降低;增大m,两者也会随之增大。所以关键问题是如何选择最优的m(或者是范围),这也是随机森林唯一的一个参数。
在随机森林算法的函数randomForest()中有两个非常重要的参数,而这两个参数又将影响模型的准确性,它们分别是mtry和ntree。一般对mtry的选择是逐一尝试,直到找到比较理想的值,ntree的选择可通过图形大致判断模型内误差稳定时的值。
随机森林R包
randomForest::randomForest 该包中主要涉及5个重要函数,关于这5个函数的语法和参数请见下方:
- randomForest(): 此函数用于构建随机森林模型
① randomForest(formula, data=NULL, ..., subset, na.action=na.fail)
- formula:指定模型的公式形式,类似于y~x1+x2+x3...;
- data:指定分析的数据集;
- subset:以向量的形式确定样本数据集;
- na.action:指定数据集中缺失值的处理方法,默认为na.fail,即不允许出现缺失值,也可以指定为na.omit,即删除缺失样本;
- ntree:指定随机森林所包含的决策树数目,默认为500;
- mtry:指定节点中用于二叉树的变量个数,默认情况下数据集变量个数的二次方根(分类模型)或三分之一(预测模型)。一般是需要进行人为的逐次挑选,确定最佳的m值;
- replace:指定Bootstrap随机抽样的方式,默认为有放回的抽样;
- classwt:指定分类水平的权重,对于回归模型,该参数无效;
- nodesize:指定决策树节点的最小个数,默认情况下,判别模型为1,回归模型为5;
- maxnodes:指定决策树节点的最大个数;
- importance:逻辑参数,是否计算各个变量在模型中的重要性,默认不计算,该参数主要结合importance()函数使用;
- proximity:逻辑参数,是否计算模型的临近矩阵,主要结合MDSplot()函数使用;
- oob.prox:是否基于OOB数据计算临近矩阵;
- norm.votes:显示投票格式,默认以百分比的形式展示投票结果,也可以采用绝对数的形式;
- do.trace:是否输出更详细的随机森林模型运行过程,默认不输出;
- keep.forest: 是否保留模型的输出对象,对于给定xtest值后,默认将不保留算法的运算结果。
- importance():函数用于计算模型变量的重要性
① importance(x, type=NULL, class="NULL", scale=TRUE, ...)
- x:为randomForest对象;
- type:可以是1,也可以是2,用于判别计算变量重要性的方法,1:表示使用精度平均较少值作为度量标准;2:表示采用节点不纯度的平均减少值最为度量标准。值越大说明变量的重要性越强;
- scale:默认对变量的重要性值进行标准化
- MDSplot():函数用于实现随机森林的可视化,
① MDSplot(rf, fac, k=2, palette=NULL, pch=20, ...)
- rf: 为randomForest对象,需要说明的是,在构建随机森林模型时必须指定计算临近矩阵,即设置proximity参数为TRUE;
- fac: 指定随机森林模型中所使用到的因子向量(因变量)
- palette: 指定所绘图形中各个类别的颜色;
- pch:指定所绘图形中各个类别形状;
- rfImpute():函数可为存在缺失值的数据集进行插补(随机森林法),得到最优的样本拟合值.
① rfImpute(x, y, iter=5, ntree=300, ...)
② rfImpute(x, data, ..., subset)
- x: 为存在缺失值的数据集;
- y: 为因变量,不可以存在缺失情况;
- iter: 指定插值过程中迭代次数;
- ntree: 指定每次迭代生成的随机森林中决策树数量;
- subset: 以向量的形式指定样本集。
- treesize(): 函数用于计算随机森林中每棵树的节点个数,
① treesize(x, terminal=TRUE)
- x:为randomForest对象
- terminal:指定计算节点数目的方式,默认只计算每棵树的根节点,设置为FALSE时将计算所有节点(根节点+叶节点)
估值过程
- 指定m值,即随机产生m个变量用于节点上的二叉树,m的选择原则是使错误率最低。
- 应用bootstrap自助法在原数据集中又放回地抽取k个样本集,组成k棵决策树,每个决策树输出一个结果。
- 对k个决策树组成的随机森林对样本进行分类或预测:分类原则:少数服从多数;预测原则:简单平均。
oob error
- 如何选择最优的特征个数m,要解决这个问题,我们主要依据计算得到的袋外错误率oob error(out-of-bag error)
- OOB:在构造单棵决策树时我们只是随机有放回的抽取了N个样例,所以可以用没有抽取到的样例来测试这棵决策树的分类准确性,这些样例大概占总样例数目的三分之一(作者这么说的,我还不知道理论上是如何出来的,但是可以自己做试验验证)。所以对于每个样例j,都有大约三分之一的决策树(记为SetT(j))在构造时没用到该样例,我们就用这些决策树来对这个样例进行分类。我们对于所有的训练样例j,用SetT(j)中的树组成的森林对其分类,然后看其分类结果和实际的类别是否相等,不相等的样例所占的比例就是OOB错误估计。OOB错误估计被证明是无偏的。
- 随机森林有一个重要的优点就是,没有必要对它进行交叉验证或者用一个独立的测试集来获得误差的一个无偏估计。它可以在内部进行评估,也就是说在生成的过程中就可以对误差建立一个无偏估计
- 在构建每棵树时,对训练集使用了不同的bootstrap sample(随机且有放回地抽取)。所以对于每棵树而言,部分训练实例没有参与这棵树的生成,它们称为第k棵树的oob样本
- 袋外错误率(oob error)计算方式如下:
- 对每个样本计算它作为oob样本的树对它的分类情况
- 以简单多数投票作为该样本的分类结果
- 最后用误分个数占样本总数的比率作为随机森林的oob误分率
randomForest::randomForest
- 在R语言中,我们调用randomForest包中的randomForest()函数来实现随机森林算法,该函数中的决策树基于基尼指数(Gini index)构建,即CART分类决策树。不过该函数有两点不足:
它不能处理缺失值,如果数据集有缺失值的话,我们必须在使用该函数之前填补
②每个分类属性的最大数量不能超过32个,如果属性超过32个,那么在使用randomForest()之前那些属性必须被转化。
- 安装程序包,查看样本数据结构
#R package # install.packages("randomForest") library(randomForest) #选取训练样本(70%)和测试样本(30%) index <- sample(2,nrow(iris),replace = TRUE,prob=c(0.7,0.3)) traindata <- iris[index==1,] testdata <- iris[index==2,]
- 遍历比较确定最优mtry值。mtry参数是随机森林建模中,构建决策树分支时随机抽样的变量个数。选择合适的mtry参数值可以降低随机森林模型的预测错误率。示例的数据中共有4个自变量,可通过遍历设定mtry参数1至4进行4次建模,并打印出每次建模的错误率,选择错误率最低的mytry取值
> #选择最优mtry参数值 > n <- ncol(iris) -1 > errRate <- c(1) > for (i in 1:n){ + m <- randomForest(Species~.,data=iris,mtry=i,proximity=TRUE) + err<-mean(m$err.rate) + errRate[i] <- err + } > print(errRate) [1] 0.05462878 0.04320072 0.04302654 0.04316091 > #选择平均误差最小的m > m= which.min(errRate) > print(m) [1] 3
- 上面代码在数据量过大,出现如下异常,待解决
> dim(car.train) [1] 70146 7 > str(car.train) \'data.frame\': 70146 obs. of 7 variables: $ DV : Factor w/ 445 levels "293.00","294.00",..: 86 120 120 120 120 120 120 120 120 120 ... $ DC : Factor w/ 710 levels "0.00","1,052.90",..: 175 16 16 16 16 16 16 16 16 16 ... $ HV : Factor w/ 112 levels "310.00","312.00",..: 11 13 18 19 20 21 22 23 23 23 ... $ LV : num 62 62 62 62 62 62 62 62 62 62 ... $ HT : num 26 26 26 26 27 27 27 27 27 28 ... $ LT : num 23 23 23 23 23 23 23 23 23 23 ... $ Type: Factor w/ 20 levels "??-????","??-????EC7 EV",..: 9 9 9 9 9 9 9 9 9 9 ... > n <- ncol(car.train) -1 > errRate <- c(1) > for (i in 1:n){ + m <- randomForest(Type~.,data=car.train,mtry=i,ntree=100, proximity=TRUE,na.action=na.omit) + err<-mean(m$err.rate) + errRate[i] <- err + } Show Traceback Rerun with Debug Error in randomForest.default(m, y, ...) : Can not handle categorical predictors with more than 53 categories. >
-
- 根据遍历打印结果,当mtry=3时,错误率达到最低,因此本次建模过程中以3作为mtry参数值
- 选择合适的ntree参数值: ntree参数指出建模时决策树的数量。ntree值设置过低会导制错误率偏高,ntree值过高会提升模型复杂度,降低效率。以mtry=3进行随机森林建模,并将模型错误率与决策树数量的关系可视化,如下:
#选择最优ntree参数值 rf_ntree <- randomForest(Species~.,data=iris) plot(rf_ntree)
-
结果图如下:
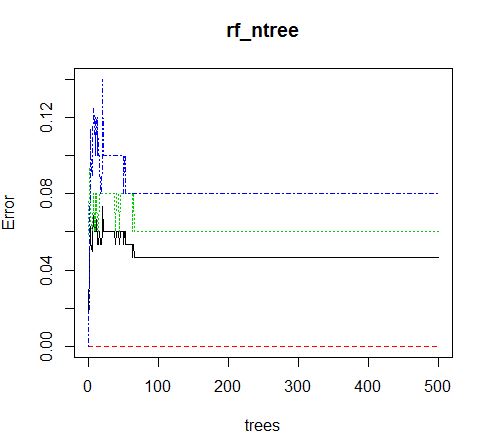
- 从图中可以看到,当ntree=100时,模型内的误差就基本稳定了,出于更保险的考虑,我们确定ntree值为100。
- 建模与观察:根据以上结果,以mtry=3,mtree=100为参数建立随机森林模型,并打印模型信息
> m <- randomForest(Species~.,data=traindata,mtry=3,ntree=100, proximity=TRUE) > print(m) Call: randomForest(formula = Species ~ ., data = traindata, mtry = 3, ntree = 100, proximity = TRUE) Type of random forest: classification Number of trees: 100 No. of variables tried at each split: 3 OOB estimate of error rate: 5.41% Confusion matrix: setosa versicolor virginica class.error setosa 39 0 0 0.00000000 versicolor 0 33 3 0.08333333 virginica 0 3 33 0.08333333
-
importance()函数:用于计算模型变量的重要性
> # importance()函数用于计算模型变量的重要性 > importance(m) MeanDecreaseGini Sepal.Length 1.201101 Sepal.Width 1.685455 Petal.Length 32.926760 Petal.Width 37.559478 > varImpPlot(m)
-
图例:
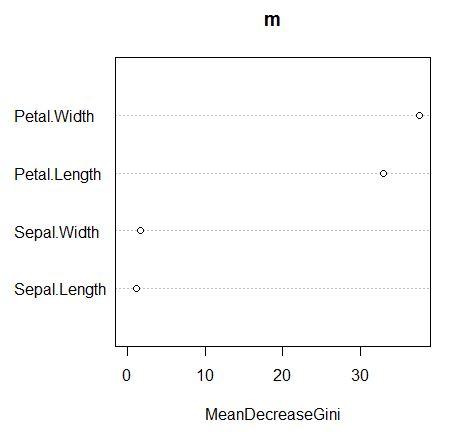
- 从返回的数据和图形可知,在四个变量中,Petal.Width和Petal.Length最为重要,其次分别是Sepal.Length和Sepal.Width
- 接着使用已建立好的随机森林模型进行新数据集的测试,如:pred <- predict(m,newdata=testdata)
- 可通过绘制测试数据的预测边距图,数据点的边距为正确归类后的比例减去被归到其他类别的最大比例。一般来说,边距为正数说明该数据点划分正确,如下图:
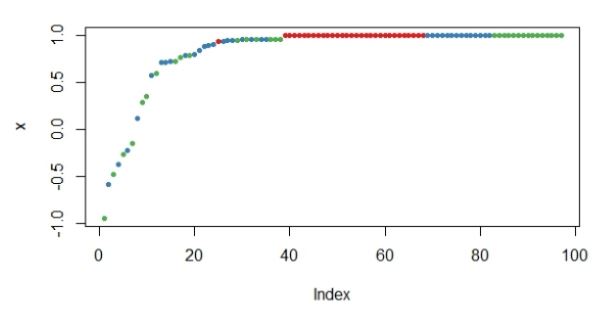
- 至此,一个简单的随机森林模型的R实现就完成了
参考资料
- https://zhuanlan.zhihu.com/p/24416833
- http://www.doc88.com/p-3436627023327.html
- http://blog.csdn.net/f_yuqi/article/details/54095477
- http://www.cnblogs.com/dudumiaomiao/p/5947369.html
- https://wenku.baidu.com/view/3fea70096bec0975f565e240.html
- http://www.36dsj.com/archives/32820
- https://www.analyticsvidhya.com/blog/2016/04/complete-tutorial-tree-based-modeling-scratch-in-python/
以上是关于ML(4.3): R Random Forest的主要内容,如果未能解决你的问题,请参考以下文章