FASTQ 数据质量统计工具
Posted Digital-LI
tags:
篇首语:本文由小常识网(cha138.com)小编为大家整理,主要介绍了FASTQ 数据质量统计工具相关的知识,希望对你有一定的参考价值。
主流工具:
拿到测序数据的第一步就是做质量控制
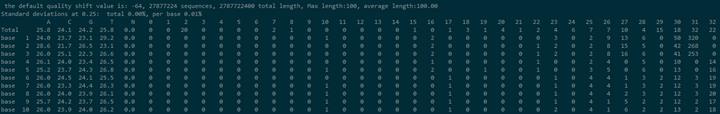
fqcheck之后得到的结果:
它会统计每条reads,按read 1-100位点计算每个位置的ACGTN含量,以及0-41质量值的个数
最终会得到整体的错误率,GC,Q20,Q30
the default quality shift value is: -64, 27877224 sequences, 2787722400 total length, Max length:100, average length:100.00 Standard deviations at 0.25: total 0.00%, per base 0.01% ··· Error Rate %GC Q20 Q30 0.61 48.35 96.26 89.88
adapter.list
#reads_id reads_len reads_start reads_end adapter_id adapter_len adapter_start adapter_end align_len mismatch FCD0JN9ACXX:6:1101:13637:2052#AGAGATCT/1 100 57 90 iPE-3+ 34 0 33 34 13 FCD0JN9ACXX:6:1101:15321:2200#AGAGATCT/1 100 53 86 iPE-3+ 34 0 33 34 0 FCD0JN9ACXX:6:1101:5318:2346#AGAGATCT/1 100 60 93 iPE-3+ 34 0 33 34 0 FCD0JN9ACXX:6:1101:5745:2411#AGAGATCT/1 100 64 97 iPE-3+ 34 0 33 34 0 FCD0JN9ACXX:6:1101:13286:2320#AGAGATCT/1 100 89 99 iPE-3+ 34 0 10 11 2 FCD0JN9ACXX:6:1101:15982:2390#AGAGATCT/1 100 80 99 iPE-3+ 34 0 19 20 0
接头序列,一般都要去掉
Illumina Adapter Sequences Document (1000000002694 v01)
过滤
使用SOAPnuke
SOAPnuke filter -l 20 -q 0.5 -n 0.1 -d -i -Q 1 -5 0 -1 1.fq.gz -2 2.fq.gz -f 1.adapter.list.gz -r 2.adapter.list.gz $tile -o 16_1.fq -D 16_2.fq -c 21
过滤后的reads同样要做质量统计
fqcheck -r 16_1.fq.gz -c 16_1.fqcheck
之后还会写个脚本作 fqcheck_distribute 分析
过滤后统计 FilterStat
得到
Type Raw data Clean data Number of Reads 52293338 48926594 Data Size 5229333800 4892659400 N of fq1 146135 35060 N of fq2 399754 16287 GC(%) of fq1 45.53 45.36 GC(%) of fq2 45.58 45.39 Q20(%) of fq1 97.03 97.99 Q20(%) of fq2 92.83 95.92 Q30(%) of fq1 91.66 93.58 Q30(%) of fq2 86.07 89.72 Discard Reads related to N 24406 Discard Reads related to low qual 2917634 Discard Reads related to Adapter 135524
catRS
drawPizza
参考:
以上是关于FASTQ 数据质量统计工具的主要内容,如果未能解决你的问题,请参考以下文章
fastq格式,如何快速计算fasta, fastq的reads数?