计算Gene co-expression features
Posted
tags:
篇首语:本文由小常识网(cha138.com)小编为大家整理,主要介绍了计算Gene co-expression features相关的知识,希望对你有一定的参考价值。
Gene co-expression features
下载 co-expression 数据
The following co-expression coefficient features were attained from COXPRESdb.
http://coxpresdb.jp/download.shtml
打开这个页面我们点击bulk download
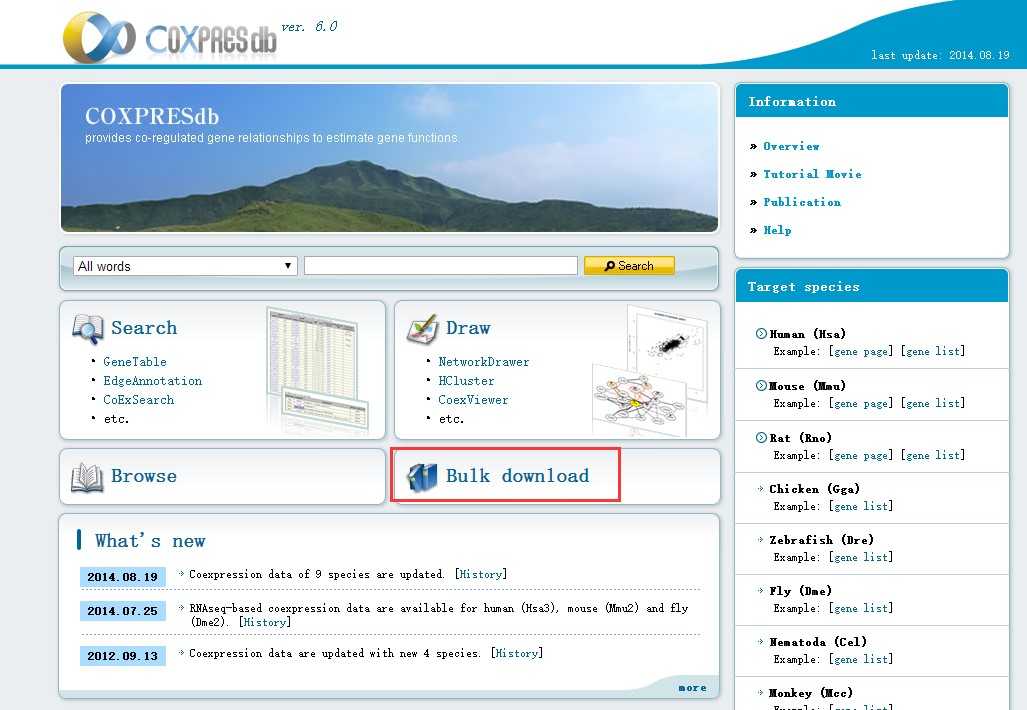

然后我们下载budding yeast 文件。
在最下面我们也可以看到文件格式的说明
Under the directory named Hsa.coex.v6, 19777 files will appear.
Hsa.coex.v6 ----- 1
|-- 10
|-- 100
|-- ...
|-- 9997
|
|
|
- Column 1; Entrez Gene ID of an opposite gene of coexpression (19776 genes)
- Column 2; MR (Mutual Rank) as a final measure of coexpression. Lines are sorted by this value.
- Column 3; Pearson‘s correlation coefficient of gene expression pattern
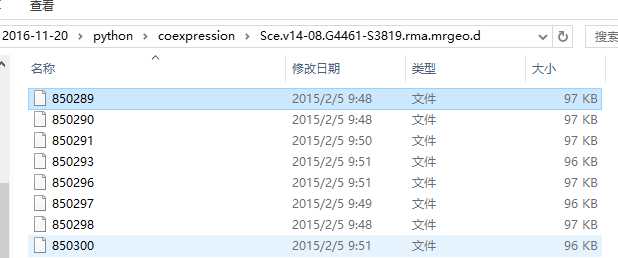
下载后的数据如下图
Sce.v14-08.G4461-S3819.rma.mrgeo.d文件夹包含4461个文件
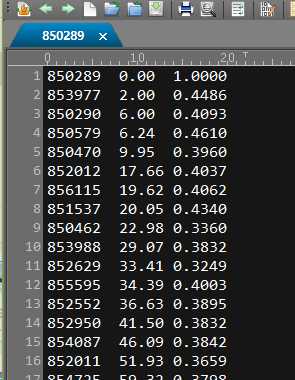
每个文件含有4461行对应4461个GENE ID
下载uniport数据
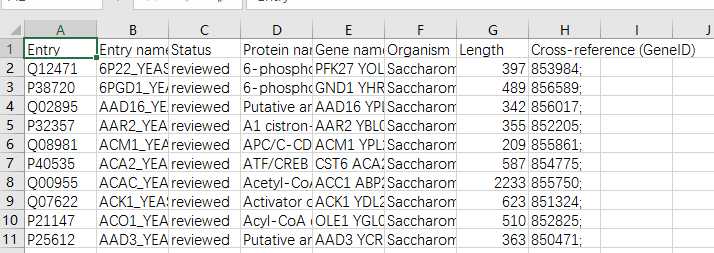
和前面同样一个问题,它这数据只给了3列,第一列 Entrez Gene ID,第二列MR,第三列COR。
蛋白质和 Entrez Gene ID的映射我们怎么得到呢?
通过各种资料。我们知道Entrez Gene ID就是ncbi里的ID,同样我们可以通过uniport下载对应的GENEID.
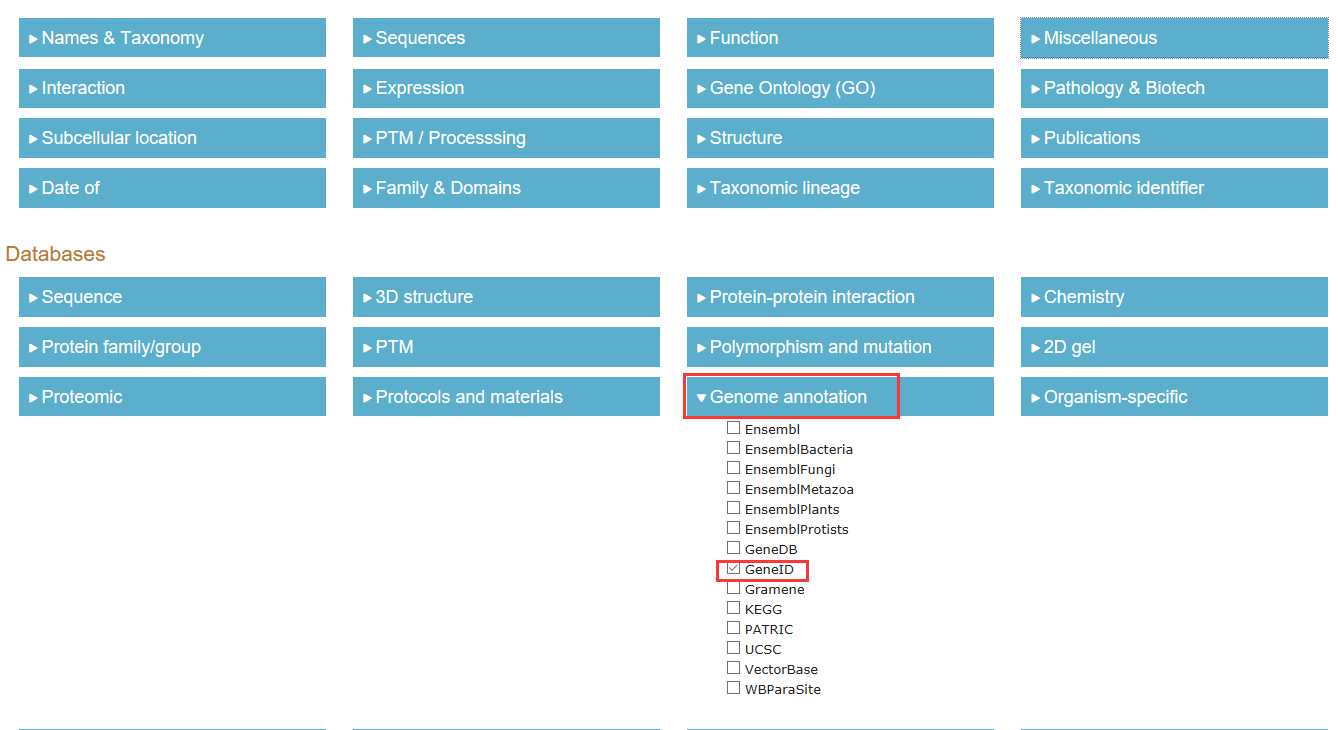
我们可以通过uniport上右边的笔头添加Gene ID列如下图所示
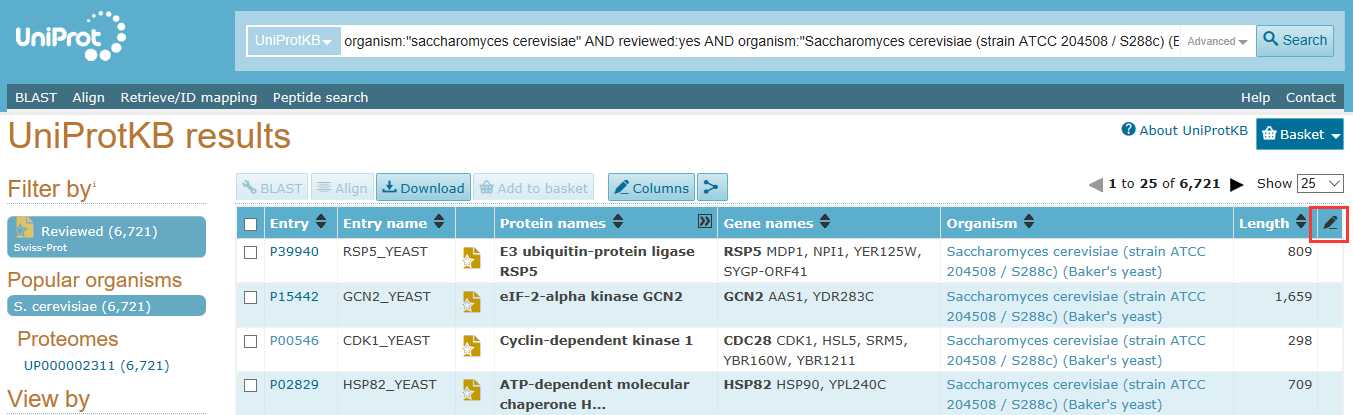
之后我们找到Genome annotation 下面的GeneID,打上勾(藏那么深也逃脱不了我的法眼。 )。这样我们就能得到GeneID信息了。
)。这样我们就能得到GeneID信息了。
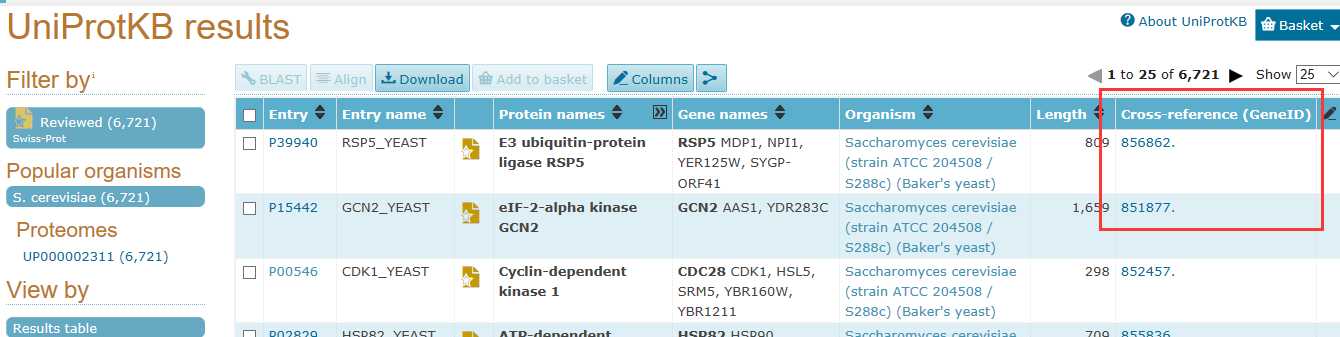
然后我们把下面的数据下载下来。
到此数据准备工作基本完成。
我们得到一个文件夹和一个uniprot和geneid对应关系的文件。
uniprot_to_geneid.csv
Ensemble learning prediction of protein–protein interactions using proteins functional annotations
获取论文的uniprot codes列(idA,idB)
yeast_gold_protein_pair.csv
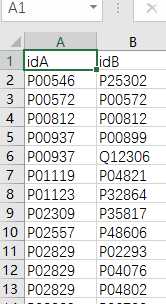
到这里我们的数据已经准备完成了。下面开始写代码!
代码的设计
# -*- coding: utf-8 -*-"""Created on Thu Nov 10 10:49:21 2016@author: sun"""import pandas as pdimport osyeast_gold_protein_pair=pd.read_csv(‘yeast_gold_protein_pair.csv‘,usecols=[‘idA‘,‘idB‘])GeneID=pd.read_csv(‘uniprot_to_geneid.csv‘,usecols=[‘Entry‘,‘Cross-reference (GeneID)‘],index_col=0)#注loc通过标签选择数据,iloc通过位置选择数据idA=GeneID.loc[yeast_gold_protein_pair.idA,:]idB=GeneID.loc[yeast_gold_protein_pair.idB,:]idA.index=range(len(idA))idB.index=range(len(idB))mr=[]cor=[]for i in range(len(idA)):GeneIDA=str(idA.iloc[i].values)GeneIDB=str(idB.iloc[i].values)ifGeneIDB!=‘[nan]‘andGeneIDA!=‘[nan]‘:GeneIDA=GeneIDA[2:8]GeneIDB=int(GeneIDB[2:8])path=‘Sce.v14-08.G4461-S3819.rma.mrgeo.d/‘+GeneIDAif os.path.exists(path):coex=pd.read_csv(path,header=None,sep=‘ ‘,index_col=0)ifGeneIDBin coex.index:mr.append(coex.loc[GeneIDB,1])cor.append(coex.loc[GeneIDB,2])else:mr.append("nan")cor.append("nan")else:mr.append("nan")cor.append("nan")else:mr.append("nan")cor.append("nan")yeast_gold_protein_pair[‘MR‘]=mryeast_gold_protein_pair[‘COR‘]=coryeast_gold_protein_pair.to_csv(‘coexpression.csv‘,index=False)
最后我们可以得到一个coexpression.csv的文件,通过查看文件我们可以知道,有686对蛋白找不到对应的MR和COR值
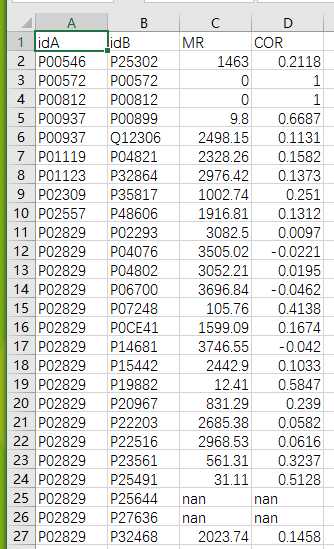
总结
Ensemble learning prediction of protein–protein interactions using proteins functional annotations
就像上面那篇论文所说的一样,缺失值太多,不适合使用,3006个样本有686 个缺失值,将近缺少了四分之一的值。
以上是关于计算Gene co-expression features的主要内容,如果未能解决你的问题,请参考以下文章