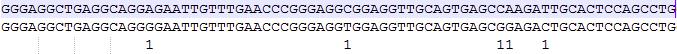Edit Distance编辑距离(NM tag)- sam/bam格式解读进阶
Posted Digital-LI
tags:
篇首语:本文由小常识网(cha138.com)小编为大家整理,主要介绍了Edit Distance编辑距离(NM tag)- sam/bam格式解读进阶相关的知识,希望对你有一定的参考价值。
sam格式很精炼,几乎包含了比对的所有信息,我们平常用到的信息很少,但特殊情况下,我们会用到一些较为生僻的信息,关于这些信息sam官方文档的介绍比较精简,直接看估计很难看懂。
今天要介绍的是如何通过bam文件统计比对的indel和mismatch信息
首先要介绍一个非常重要的概念--编辑距离
定义:从字符串a变到字符串b,所需要的最少的操作步骤(插入,删除,更改)为两个字符串之间的编辑距离。
(2016年11月17日:增加,有点误导,如果一个插入有两个字符,那编辑距离变了几呢?1还是2?我又验证了一遍:确实是2)
这也是sam文档中对NM这个tag的定义。
编辑距离是对两个字符串相似度的度量(参见文章:Edit Distance)
举个栗子:两个字符串“eeba”和“abca”的编辑距离是多少?
根据定义,通过三个步骤:1.将e变为a 2.删除e 3.添加c,我们可以将“eeba”变为“abca”
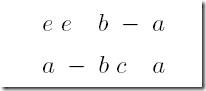
所以,“eeba”和“abca”之间的编辑距离为3
回到序列比对的问题上
下面是常见的二代比对到ref的结果(bwa):
D00360:96:H2YLYBCXX:1:2110:18364:84053 353 seq1_len154_cov5 1 1 92S59M8I17M1D6M1D67M seq30532_len2134_cov76 1 0 AAAAAAAAAAAAAAAAAAAAAAAACCCTGTCTCTAATAAAATACAAACAATTAGCCGGGCATGGTGGCACGCGCCTTTAGTCCCAGCTACTAGGGAGGCTGAGGCAGGGGAATTGTTTGAACCCGGGAGGTGGAGGTTGCAGTGAGCGGAGTTTTTTTCACTGCACTCCAGCCTGGTGACAAATCAAAAATCCATTTCAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAACAAAACAACAAA
DDDDDHIIIIIIII<EHHII?EHH0001111111<11<DEH1D1<FH1D<<<C<@GEHD</<11<101<1D<<C<E0D11<<1<D?1F1CC1DE110C0D1011100////0DD<1=@1=FGHCDHH<FG<D0<<<EF?CE<00<<0<D//0;<:D/////;///////;8F.;/.8.8......88.9........-8BBGADHIIHD?>D?HH<,>=HHDD,5CHDCDHD><,,,--8----8-8-- NM:i:25 MD:Z:16A21C16C0A3T15^A6^G1G0T0G3C2T0G1C41A4A2A3 AS:i:49 XS:i:42 SA:Z:seq13646_len513_cov63,125,-,103S125M21S,1,11; RG:Z:chr22
这是ref序列
>seq1_len154_cov5
GGGAGGCTGAGGCAGGAGAATTGTTTGAACCCGGGAGGCGGAGGTTGCAGTGAGCCAAGATTGCACTCCAGCCTGGATGACAAGAGTGAAACTCTGTCTCAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAA
cigar字段包含了序列比对的简化信息,M(匹配比对,包含match和mismatch),=(纯match),X(纯mismatch),I(插入),D(删除),还有N、P和S、H。(注:目前只在blasr比对结果中见过=和X)
根据cigar字段可以统计indel信息,但是无法统计mismatch。
这个时候就可以用到NM tag了,mismatch = NM – I - D = 25 – 8 – 1 – 1 = 15
有兴趣的可以对着cigar数一遍,下面是我无聊数着的结果,也是15个
使用python的pysam模块可以很容易的提取出每个比对结果的NM信息
参见之前的帖子:pysam - 多种数据格式读写与处理模块(python)
再次验证一遍:证明NM=len(mismatch) + len(insertion) + len(deletion), 而不是 count(mismatch) + count(insertion) + count(deletion) (count的意思是三个碱基的insertion算一个)
>>> bam_file = pysam.AlignmentFile(infile, "rb") >>> for line in bam_file: ... if 300 < line.query_alignment_length < 500: ... break ... >>> line.query_alignment_length 321 >>> line.seq \'CTCTTCATCACGTCACTTGTCCATGTCAATGGCTACAGGTTGGAAGTTTGGCCGCGGAGGGTGGGCAGGCAAGAGAAAGAAATCAGATAGGGCAGATGGTAGGGTAAAAGGAGGGGGTTAAGTGCAAATTGTCTACTGTTTGCAAATGGGAAGCATGTGATTGTTAAAATTTATACGATAAACCTTCTCATCATGTTGAGTCTCATGCTTGCGCCAAGAAGATCGGGTTCGGCGGGTCAAGCTGATAAGCAACTTGGGCAGCAAAGTCGTTCAGTGATACAAAATCATGTGCAAAAATCACAAGCATTCTTATAAACACCA\' >>> line.cigarstring \'5148H3M1I2M1I4M2I3M3I3M1D2M1I4M3I1M1I4M1D10M1D5M4I4M3D1M3I18M1I1M1I1M1I4M4D2M1I2M2I4M2I4M1I4M5I4M1I9M1I14M3I1M1I2M2I7M2I7M3I2M1I5M2I1M1I2M1I1M1I4M5I3M1I2M3I1M1I4M4I3M4I2M2I1M3I19M3I10M2I4M1I11M1D27M2I6M25212H\' >>> line.reference_name \'Backbone_1\' >>> line.reference_start 7164 >>> line.reference_end 7413 >>> line.get_tag(\'NM\') 100
取出了一条长度适中的比对结果,cigar字段比较全面。
取出了比对上的ref:
>>> ref = \'CTCTCTCACCACTCCTATTCAACACAGTGTTGGAAGTTCTGGACAGGGCAATCAGGCAAGAGAAAGAAATAAAGGGTATTCAATTAGGAAAAGAGGAAGTCAAATTGTCCCCGTTTGCAGATGACATGATTGTATATTTAGAAAACCCCACTGTTTCAGCCCCAAATCTCCTTAAGCTGATAAGCAACTTCAGCAAAGTCTCAGGATACAAAATCAATGTGCAAAAATCACAAGCATTCTTATACACCA\'
先通过我自己写的cigar校正函数校正:
def formatSeqByCigar(seq, cigar): \'\'\' Input: query_alignment_sequence and cigar from sam file Output: formatSeq Purpose: make sure the pos is one to one correspondence(seq to ref) \'\'\' formatSeq = \'\' pointer = 0; qstart = 0; qend = -1; origin_seq_len = 0 if cigar[0][0] == 4 or cigar[0][0] == 5: qstart = cigar[0][1] if cigar[-1][0] == 4 or cigar[-1][0] == 5: qend = - cigar[-1][1] - 1 # fushu count for pair in cigar: operation = pair[0] cigar_len = pair[1] if operation == 0: # 0==M formatSeq += seq[pointer:(pointer+cigar_len)] pointer += cigar_len origin_seq_len += cigar_len elif operation == 1: # 1==I pointer += cigar_len origin_seq_len += cigar_len elif operation == 2: # 2==D formatSeq += \'D\'*cigar_len elif operation == 4 or operation == 5: # 5==H origin_seq_len += cigar_len continue else: print (cigar) raise TypeError("There are cigar besides S/M/D/I/H\\n") return formatSeq, qstart, qend, origin_seq_len
然后分别计算deletion和mismatch:
>>> i = 0 >>> count = 0 >>> while i < len(ref): if formatSeq[i] != ref[i]: count += 1 i += 1 >>> count 17 >>> formatSeq.count(\'D\') 11
可以看出deletion有11个,退出mismatch有6个。
随手一推insertion有83个。
而
>>> cigarstring = \'5148H3M1I2M1I4M2I3M3I3M1D2M1I4M3I1M1I4M1D10M1D5M4I4M3D1M3I18M1I1M1I1M1I4M4D2M1I2M2I4M2I4M1I4M5I4M1I9M1I14M3I1M1I2M2I7M2I7M3I2M1I5M2I1M1I2M1I1M1I4M5I3M1I2M3I1M1I4M4I3M4I2M2I1M3I19M3I10M2I4M1I11M1D27M2I6M25212H\' >>> cigarstring.count(\'I\') 41 >>> cigarstring.count(\'D\') 6
用count计算显然不对。
验证成功
真切的明白了计算机有多么伟大,如果要是你肉眼去比对去数的话,我估计你会立马崩溃。
以上是关于Edit Distance编辑距离(NM tag)- sam/bam格式解读进阶的主要内容,如果未能解决你的问题,请参考以下文章
leetcode 72. Edit Distance 编辑距离(中等)
算法: 编辑距离使得单词相同 72. Edit Distance
算法: 编辑距离使得单词相同 72. Edit Distance