ADMIXTURE 是常用的群体遗传学分析工具,可以估计个体的祖先成分。与 STRUCTURE 相比,它的速度更快。
下面介绍一下它的使用。STRUCTURE 可以输入 Plink 或者 EIGENSTRAT 格式的数据,这里以 plink 格式的文件为例。
筛选SNP
SNP 数量太多,计算会非常慢。可以使用 plink 的 --indep-pairwise 命令,通过 LD 筛选位点:
plink --bfile data --indep-pairwise 50 10 0.1
plink --bfile data --extract plink.prune.in --make-bed --out data.pruned
寻找最佳k值
如果不知道k值设多少,可以在一系列不同的k值中进行交叉验证,选择最佳的k。
使用 --cv=n 参数,Admixture 会把基因型划分成均等大小的 n 份做交叉验证。不指定 n 时,默认为5。
为了加快计算的速度,还可以通过 -jn 的命令多线程计算,其中 n 为 线程数。
比如,使用默认的 5-fold cross-validation,以 40 个线程并行,从 1 至 15 中寻找最佳的k值:
for K in $(seq 1 15); do admixture --cv data.pruned.bed $K -j40 | tee log${K}.out; done
完成计算后,获取交叉验证的结果:
grep -h CV log*.out
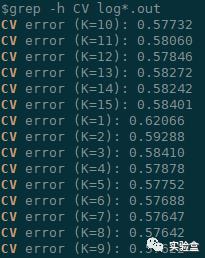
最低的 CV errors(cross-validation error)对应的 k 值,是其中最理想的选择。比如这里最低的是 K=9 时的 0.57622,因而选择 9 作为分析的 k 值。
利用最佳k值分析
知道最佳 k 值后,就可以直接计算群体成分。以 k=9,使用 20 个线程为例:
admixture data.pruned.bed 9 -j20
计算完成后,得到的 .Q 结尾的文件便是各个个体的群体成分。
如果只是简单看看,直接用 R 画个 barplot 就可以:
tbl=read.table("hapmap3.3.Q")
barplot(t(as.matrix(tbl)), col=rainbow(3),xlab="Individual #", ylab="Ancestry", border=NA)
如果要画更详细的图,可以用 R 包 pophelper。
