置换检验—结合GSEA解释
Posted zypiner
tags:
篇首语:本文由小常识网(cha138.com)小编为大家整理,主要介绍了置换检验—结合GSEA解释相关的知识,希望对你有一定的参考价值。
置换检验
参考网址:https://www.plob.org/article/3176.html
置换检验(permutation test):利用样本数据的全(或随机)排列,进行统计推断的方法。特别适用于总体分布未知的小样本资料。
结合GSEA,解释permutation test:
首先,有两种phenotype(treat和control)的RNA-seq的结果,即每个基因在这两种phenotype中的表达量。这两种phenotype可看做是置换检验中的A、B组数据。
零假设:GO过程(如脂肪代谢)的基因在这两组数据上的表达量没有差异。
步骤:1)从RNA-seq结果中抽取出这些GO过程的基因的表达值(比如,GO过程中有100个基因落入RNA-seq的结果中)。这样得到A、B两组数据,A组有100个基因的表达值,B组有100个基因的表达值。
2)将A、B的数据混在一起,打乱顺序。随机抽取100个数据作为A组的数据,剩余的作为B组的数据。共随机抽取1000次。统计量设为:A组的平均值-B组的平均值。
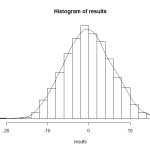
3)根据2),得到1000个统计量的值。这些值代表了抽样总体情况。画出这1000个统计值的分布图。如下:
4)再看看真实的RNA-seq数据中A组和B组的均值差(比如:14)。看看落入分布图的哪个区域。
5)假如真实的均值差落入尾部,则说明:在零假设条件下,这个数出现的概率很低。只有9个值大于14,计算概率:P-value=9/1000=0.009.
6)P-value值<0.01,差异显著。说明:GO过程(如脂肪代谢)的基因在这两组数据上的表达量是有差异的。
直观来讲,我想比较某个GO过程在A、B组中是否有差异。
用什么数据衡量?用GO过程基因集在A、B两组的表达值。
怎么衡量?看每个基因在A、B组上表达值是否有差异?
怎么衡量差异?看A组的基因表达均值,与B组的基因表达均值之差,看均值之差的大小?如果差大,说明A、B组有差异;如果差小,说明A、B组没有差异。(此时,AB组的均值之差记为真实的均值差值。)
怎么衡量均值差的大小呢,怎么样是大,怎么样是小?用分布吧。用什么分布?正态分布?不知道参数。(这一步我也不太理解)
用检验吧。将A、B两组数据打乱,然后分成两组,看看这两组的均值之差。执行1000次前面的步骤,就得到1000个均值之差。看这1000个值的分布如何。
看分布不是我的目的,我的目的是:A、B两组的真实的均值差值,说明A、B两组有差异呢,还是没有差异。
将真实的均值之差放到分布中看。如果在分布图的尾端,说明真实的均值之差不在大多数情况中。如何衡量呢?用分布曲线下的面积来衡量,即P-value。如果P-value小,说明真实的均值之差在分布图中占的比例小。
总结:其实,做1000次抽样,是为了构造上面的分布图,有了分布图,才能确定我的真实值处在分布图中的什么位置。P-value是衡量我的真实值在分布图中的概率。
再衍生出统计量的概念,即上面提到的A、B两组的均值。
再联想到,点突变频率的P-value如何理解?Mutect的算法如何理解?
后续再补吧!
结合GSEA说明:
随机抽取次数(比如上文的1000),即是GSEA中提到的number of permutation。次数越多,得到的统计量的值越多,抽样分布的值越多,抽样分布的分布图更接近真实值。看真实数据在此抽样分布图上的分布时,就更准确。
以上是关于置换检验—结合GSEA解释的主要内容,如果未能解决你的问题,请参考以下文章