脑肿瘤分割论文:TransBTS: Multimodal Brain Tumor Segmentation Using Transformer
Posted 北辰浅巷墨漓
tags:
篇首语:本文由小常识网(cha138.com)小编为大家整理,主要介绍了脑肿瘤分割论文:TransBTS: Multimodal Brain Tumor Segmentation Using Transformer相关的知识,希望对你有一定的参考价值。
标题:TransBTS: Multimodal Brain Tumor Segmentation Using Transformer(TransBTS:使用多模态的脑肿瘤分割)
published in:MICCAI 2021: Medical Image Computing and Computer Assisted Intervention – MICCAI 2021
论文地址:https://linkspringer.53yu.com/chapter/10.1007/978-3-030-87193-2_11
代码地址: https: //github.com/Wenxuan-1119/TransBTS
1.摘要
Transformer最近在自然语言处理和2D图像分类方面取得了成功,它可以利用自我注意机制进行全局(长期)信息建模。然而,局部和全局特征对于密集预测任务至关重要,尤其是对于三维医学图像分割。在本文中,我们首次利用3D CNN中的Transformer进行MRI脑肿瘤分割,并提出了一种基于编码-解码结构的新型网络TransBTS。为了捕获局部3D上下文信息,编码器首先利用3D CNN提取体积空间特征地图。同时,对特征映射进行了精心的改造,将标记输入Transformer进行全局特征建模。解码器利用Transformer嵌入的功能并执行渐进式上采样以预测详细的分割图。在BraTS 2019和2020数据集上的大量实验结果表明,TransBTS在3D MRI扫描中脑肿瘤分割方面取得了与之前最先进的3D方法相当或更高的结果。
2.Method
2.1TransBTS的总体架构
将给定的MRI图像作为输入X,空间分辨率为HW,深度维度为D(表示有多少个切片),C表示通道(也就是要分为几个模态)。我们首先利用3D CNN生成捕获空间和深度信息的紧凑特征图,然后利用Transformer编码器在全局空间中建模长距离依赖关系。然后,我们重复叠加上采样层和卷积层,以逐步生成高分辨率的分割结果。

2.2 网络编码器
 由于Transformer的计算复杂度和序列长度是二次的,因此将图片直接输入是不太现实的。首先要将图片分割为固定大小(16*16)的切片,*然后将每个面片重塑为一个标记,将序列长度减少到162。为了解决Transformer无法跨越空间和深度维度建模图像局部上下文信息的问题,本文采用的解决方案是将3×3×3卷积块与降采样(步幅为2的步幅卷积)叠加,以逐渐将输入图像编码为低分辨率/高层次特征表示。
2.3 网络解码器
特征映射模块:transformer 首先被重塑为d * H/8 * W/8 * D/8,为了降低解码器的计算复杂度,采用卷积块将信道维数从d降到K。通过以上操作,得到了在编码器部分与F维数相同的特征映射Z。在特征映射后,对Z进行级联上采样操作和卷积块,逐步恢复全分辨率分割结果R。此外,通过级联实现更精细的分割掩码和更丰富的空间细节,使用跳转连接将编码器特征与解码器特征进行融合。
3.实验部分
3.1数据集
实验中包含两个数据集,分别为BraTS2019和BraTS2020挑战赛提供的公开数据集,其中2019的数据集包括335个病例用于训练,125个用于验证。2020的数据集包含369个病例用于训练,125个病例用于验证,166个病例用于测试。
3.2 使用的技术
采用Adam优化器来训练模型,初始学习率设置为0.0004,其中初始速率通过每次迭代以0.9的幂衰减。
硬件设备:使用8个NVIDIA Titan RTX GPU(每个GPU有24GB内存)进行培训,将batch_size设置为16,训练8000个epoches.
实验中使用到以下几种增强技术:
- 将图片240x240x155随机裁剪到128x128x128像素.
- 沿着轴向、冠状和矢状进行随机翻转,概率为0.5.
- 随机强度在[-0.1,0.1]之间移动,标度在[0.9,1.1]之间。采用softmax Dice损耗对网络进行训练,L2范数也用于模型正则化,权重衰减率为10−5、在测试阶段,我们利用Test Time Augmentation(TTA)来进一步提高我们提出的TransBTS的性能。
3.3主要结果
TransBTS在ET、WT和TC上的Dice结果分别是78.93%、90.00%和81.94%,由于验证集中的标签不能获取,因此对所有 方法的训练集进行了5次交叉验证进行评估。各个方法的验证集结果如下:
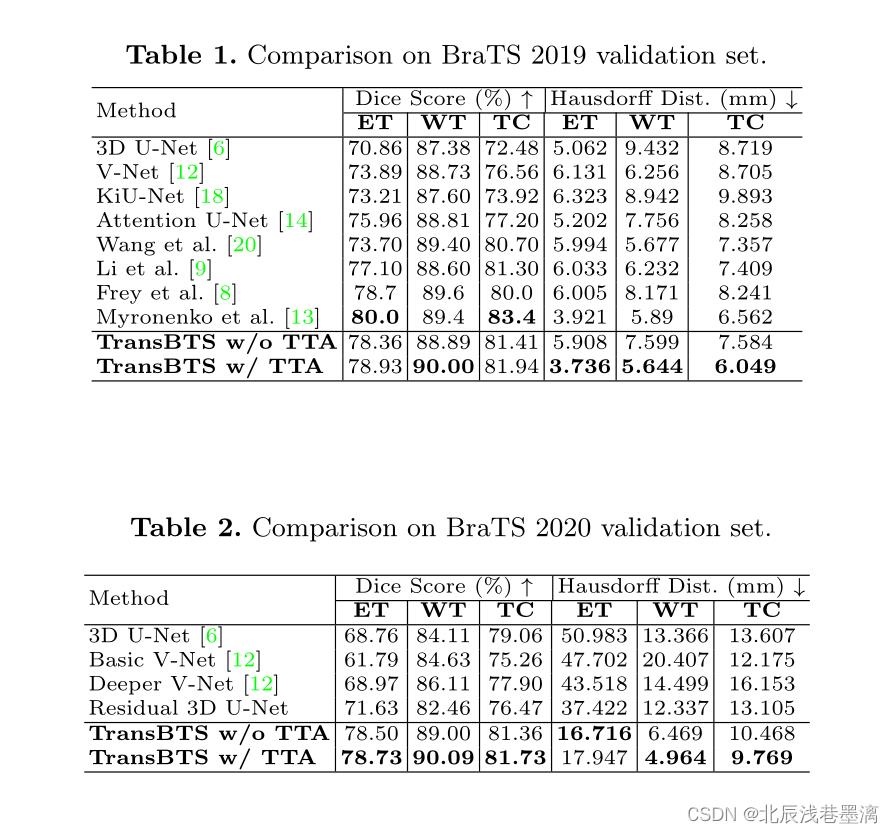
3.4消融实验
1)研究Transformer序列长度(N)的影响,该长度由网络编码器中3D CNN的总体步幅(OS)控制。
2) 我们在不同的模型尺度(即深度(L)和嵌入维度(d))下探索变压器。
3)我们还分析了跳过连接的不同位置的影响。
消融实验结果如下:

从表3中可以看出,OS从16调整到8来增加序列长度(从512增加到4096),可以显著的提高性能。将OS设置为4后,我们无法直接将每个volume重塑为特征向量。因此,我们做了一点修改,将序列长度保持在4096,即在传递到转换器之前,将每个2×2×2面片展开为一个特征向量。但是并没有提高性能,反而变得更差了。
嵌入维数(d)和tansformer层数(深度L)都决定着transformer的规模,因此进行消融实验验证transformer规模对于分割性能的影响。从表4中可以看出,L=4和d=512在ET和WT的性能最好。
5.结论
本文提出了一个新的分割框架,该框架在3D CNN中有效地结合了Transformer,用于MRI中的多模式脑肿瘤分割。最终的体系结构TransBTS不仅继承了3D CNN对本地上下文信息建模的优势,而且还利用Transformer学习全局语义关联。在两个数据集上的实验结果验证了TransBTS的有效性。
以上是关于脑肿瘤分割论文:TransBTS: Multimodal Brain Tumor Segmentation Using Transformer的主要内容,如果未能解决你的问题,请参考以下文章