清华AI蛋白质结构预测,连续4周夺得CAMEO第一
Posted QbitAl
tags:
篇首语:本文由小常识网(cha138.com)小编为大家整理,主要介绍了清华AI蛋白质结构预测,连续4周夺得CAMEO第一相关的知识,希望对你有一定的参考价值。
衡宇 梦晨 发自 凹非寺
量子位 | 公众号 QbitAI
AI蛋白质结构预测赛道,国产模型又有吸睛表现:
在蛋白质结构预测竞赛CAMEO上,有支队伍连续四周夺得全球第一。
达成这一成就的是来自清华大学智能产业研究院(AIR)的AIRFold。
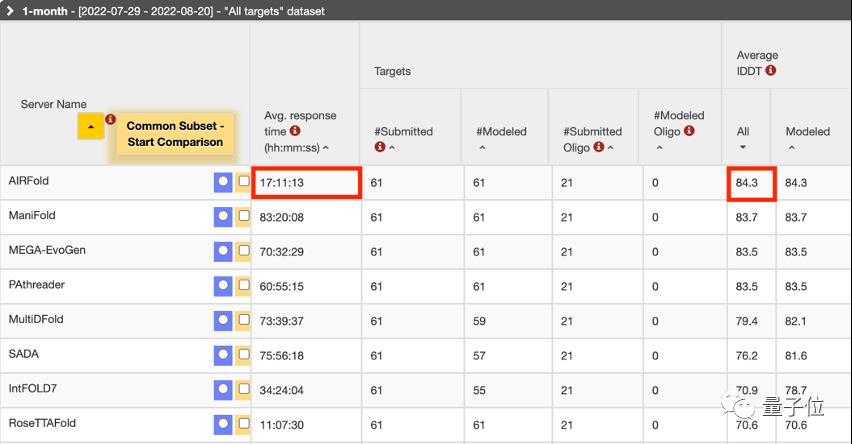
△AIRFold 在7.23-8.20的评估中连续四周全球第一
CAMEO竞赛(Continous Automated Model Evaluation)与CASP并列为蛋白质结构预测领域的两大权威竞赛。
不同之处在于CASP两年一届,CAMEO则是持续举办,每周都有结构生物学家最新破解出的蛋白质结构作为赛题。
CAMEO上得分与排名每周实时更新,华盛顿大学David Baker团队的RoseTTAFold、百度腾讯华为等行业顶尖选手都在其中参与角逐。
AIRFold在近4周的比赛中,不仅预测结果IDDT分数领先,系统响应时间上也远远领先后几名的团队。
亮眼成绩如何取得?后续又有哪些研究和应用方向?
带着这些问题,我们联系到项目负责人清华大学智能产业研究院清华大学智能产业研究院(AIR)的兰艳艳教授,与她进行了深入交流。
下面送上对话实录,为方便阅读,我们在不改变原意的基础上做了编辑整理。
对话实录
量子位:AIRFold项目是从什么时候开始做的?能否介绍一下团队基本情况?
兰艳艳教授:AIRFold项目是AIR智慧医疗方向的一个重要部分,大约是2021年9月份开始,距离现在刚好一年左右的时间。
团队成员是陆续到位的,目前总共有7-8人,除我之外还包括科研工程师,博士后和博士生等。成员背景基本都是AI方向的,也有生物学和化学背景的同学参与。
张亚勤老师和马维英老师在整个项目进行过程中也一直在帮我们把握方向,提供资源支持,给我们团队很多指导。同时我们的访问教授彭健老师以及他带领的Helixon(华深智药)团队也和我们进行过多次讨论,对我们进行了一些技术指导,帮助我们团队得到了很好的成长。
量子位:在AlphaFold2之后,出现了单序列预测的一些方法,AIRFold为什么坚持走同源信息挖掘这条路线?
兰艳艳教授:OmegaFold和ESMFold等单序列模型确实没有显式地使用MSA作为特征,但严格讲并非没有用同源序列中隐含的共进化信息,它采取了一种隐式的使用方式。具体来说,OmegaFold通过基于掩码语言模型(Mask Language Model, MLM)的蛋白质预训练模型编码了主序列然后用于预测结构,MLM天然地具有捕捉共进化信息的能力,这点在早期Meta 的蛋白质预训练工作ESM中也有体现。直接使用MSA或者使用具备捕捉共进化能力的编码器都是不同的方法而已。
我们选择同源挖掘路线主要有几个原因:
第一,从效果上讲,以Meta(原FAIR)的ESMFold为代表的基于单序列的结构预测方案,比基于单序列的AlphaFold2效果要好,但是与直接显式使用MSA序列的AlphaFold2方法相比还有不少差距。例如ESMFold在CAMEO以及CASP数据集上测试所得的TM-score分别是82.8以及67.8,对应AlphaFold2的TM-score是88.3以及84.7,有较大差距。我们认为ESMFold确实给我们指明了利用同源信息的新方式,但要达到替代MSA的效果还有较大的改进空间。
第二,当时选择同源挖掘这条路线,首先是因为我们团队有丰富的NLP背景,我们一看到AlphaFold2,就发现MSA这个模块作为同源信息的输入非常关键,而AlphaFold2的使用方式还停留在传统方法上,因此我们很自然会优先选择从我们擅长的MSA序列建模和检索这个方面入手,应用最新的NLP技术来进行突破。
第三,最重要的原因,我们做AIRFold和参加比赛最终的目的不仅仅是为了蛋白质结构预测本身。我们希望在这个过程中从建模和计算的角度探究哪些重要信息如何作用最后影响了折叠的结果,这些积累能够锻炼我们的队伍,让大家对结构预测这个问题有更深刻的认识,同时也会启发我们对蛋白质相互作用、序列到结构到功能等问题的思考,从而促进我们开展与AI赋能新药研发相关领域的研究和应用。
量子位:能否展开讲讲同源挖掘模块Homology Miner的技术细节和特色?
兰艳艳教授:挖掘同源信息是目前主流蛋白质结构预测模型以及参赛服务器都会关注的一个关键技术方向,AIRFold的特色集中在获取同源蛋白和对同源蛋白进行优化校正的方法上。
AIRFold 的Homology Miner在经典的同源检索算法之外,整合了一些基于NLP全新技术所形成的算法,包括稠密检索、面向多序列比对的同源蛋白生成等模型,这一系列的方法在一些初始缺乏同源信息的孤儿蛋白上,展示了比较明显的效果,说明目前主流的同源检索方法存在可提升空间。
除此之外,我们针对“什么是好的同源蛋白”这一问题,从信息论的角度给出了一个量化的定义,基于这一量化指标对于同源表征进行优化,可以稳定地提高结果以及鲁棒性,这也为同源表征学习也提供了一个全新的思路和角度。
量子位:除了同源挖掘模块外,AIRFold在AlphaFold2的基础上还做了哪些改进?
兰艳艳教授:除了同源挖掘模块,我们对AlphaFold2目前所存在的一些问题也进行了有针对性的探索和改进。
例如在结果预测上,AlphaFold2还无法很好的解决蛋白的多构象和点突变问题,模型精度(即pLDDT)的预测也存在偏差的问题等。
以pLDDT的偏差为例,pLDDT本身是结构预测结果的一个置信度,大家发现在AlphaFold2提供的预测结果中,pLDDT通常还是比较准的,高的地方预测结果相对比较准确,低的地方预测结果不够好,但是事实上作为神经网络的输出结果,pLDDT的鲁棒性很差,很难反映MSA的微小变化或攻击带来的影响,这样就导致在比赛或者实际应用中,如果完全以pLDDT为标准进行选择,会引发严重的问题。
在这方面我们也提出了一些对抗训练,多目标优化等新的思路,在这些问题的解决上取得了一些进展,欢迎大家关注我们后续的科研工作。
量子位:AIRFold保持高IDDT评估的同时,在系统响应时间上远远领先其他团队,是靠什么做到的?
兰艳艳教授:AIRFold是一个自动化的平台,包括同源序列增广、同源序列筛选、特征处理、结构预测、结果分析以及自动提交等模块。
比赛序列过来的时候没有任何人为的干预,我们的系统会自动的监控server是否有新来的序列,自动的补上提前设置好的参数配置,自动对蛋白质结构进行预测最后提交预测结果。
我们设计并实现AIRFold的初衷就是为蛋白质结构预测以及同源蛋白分析这一问题,找到通用的解决方案。尽管在比赛中的序列之间差异很大,比如有的同源很多,有的同源蛋白很少,我们在比赛中始终坚持使用同一套策略和系统,来减少人工对于不同的比赛序列进行不同的处理,力求得到一套通用的结构预测解决方案,这是我们响应迅速的主要原因。
量子位:AIRFold团队重点介绍了CAMEO比赛中一个较难预测的蛋白7TVI,它的预测难点在哪里?
兰艳艳教授:7TVI是来自 Planctomycetes(浮游菌门)细菌的Cas13bt3蛋白,相比其他序列来说有两个难点。
首先是这个序列同源序列相对少,多序列比对(Multiple Sequence Alignment ,MSA)结果无论是从深度还是覆盖度来说都是非常不理想的。我们第一次搜索的结果只搜索到了700条左右的同源序列,有三分之二以上的序列基本没有覆盖。我们都知道当下流行的AlphaFold2是高度依赖同源信息的,这样低质量的同源序列必然是不利于结构预测的。
AIRFold团队充分利用自主研发的HomoMiner的优势,对低质量的MSA进行筛选过滤,选择其中有价值的部分,去除冗余;同时利用深度稠密检索技术和同源序列生成技术对MSA进行补充,丰富其中的信息,因此能在这个序列上做出比较好的结果。
其次,这个蛋白结构域多,变构大,因此构象比较复杂灵活。从PDB的结构和以往的研究来看,这个蛋白有HEPN1和HEPN2两个核酸酶结构域,crRNA结合结构域又分为Helical1-1,Lid,Helical1-2,Helical2和Helcal1-3五个,中间有linker连接。目前主流的结构预测方法,比如AlphaFold2和ESMFold都主要使用单结构域蛋白进行训练,这是因为PDB数据库中单结构域蛋白远多于多结构域蛋白。
此外,MSA中往往也会出现每条序列只覆盖一个结构域的情况,不能提供多结构域之间关系的信息。这就造成多结构域之间关系不容易被准确预测。
AlphaFold-Multimer的成果对我们很有启发,秉承结构域间关系信息也蕴藏于MSA中的理念,我们使用HomoMiner对MSA进行筛选,去除信息量低、噪声大的序列,提高高质量同源序列中长程相互作用信息的信噪比。因此我们可以更准确地建模多结构域之间的关系。
这些经验也符合我前面说到的,充分挖掘MSA信息虽然更加耗时,但是在实际数据上能提供更具启发性的结果,也能够让我们在此过程中有思路去分析蛋白质的进化生物学问题。

量子位:除了CRISPR相关分子工具的挖掘与设计之外,AIRFold还在哪些领域有竞争优势?
兰艳艳教授:除了研究CRISPR/Cas系统本身之外,其实我们也在关注一些抗CRISPR蛋白(anti-CRISPRs,Acrs)。Acrs其实是非常有意思的蛋白。一方面,一些噬菌体会表达Acrs蛋白,从而增强其侵染细菌的能力。另一方面,一些细菌会产生针对自身基因组的CRISPR(self-targeing CRISPR),为了防止“自身免疫病”,细菌自己也会表达这样的Acrs蛋白。
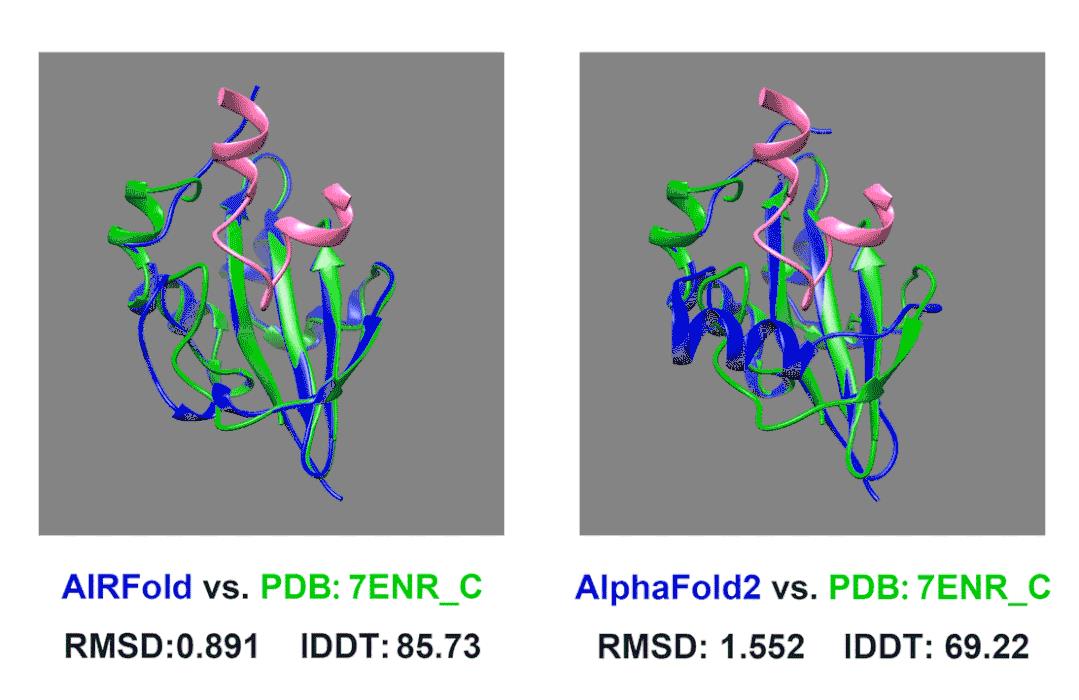
在我们内部的测试中,我们测试了一个上面描述的Acrs蛋白。7ENR_C这个蛋白是来自葡萄球菌的AcrIIA14,他可以结合Cas9抑制其活性(其中Cas9结合AcrIIA14的片段在图片中标记为粉色)。AcrIIA14这个蛋白同源蛋白非常少,搜索数据库后没有找到什么同源序列,AlphaFold2预测的结构的lDDT也只有不到70。我们使用HomoMiner对MSA进行补充,能够非常有效地提高预测效果,lDDT一下提高到了85。
除此之外,我们还观察到原来AlphaFold2预测的不太好的区域主要是结合Cas9的结合位点附近,AlphaFold2预测的口袋偏小,而这个位置我们预测的结构更接近真实结构,口袋大小也更合适。这样准确的预测可以允许我们更好的将预测Acrs蛋白结构并后续和已知的Cas9蛋白结构进行对接,分析其阻遏Cas9的原理,从而启发我们设计出更强的Acrs来强化噬菌体疗法;也可以助力相关抑制剂的设计。也许以后我们能让细菌患上“自身免疫病”,缓解日益严重的抗生素耐药问题。
我们后续会继续推进在CRISPR/Anti-CRISPR这一对欢喜冤家上的结构预测,增强和相关生物研究组的合作,共同发掘微生物这一神奇的系统。
量子位:在研发过程中遇到最大的困难是什么?有没有一个印象特别深刻的事件?
兰艳艳教授:最大的困难是最开始的时候,团队的主要成员背景都不是生物计算,对于蛋白质结构预测更是知之甚少。大家从头开始,花了很多力气一起学习领域知识,读paper,向生物计算背景的人请教,包括Helixon的彭健老师以及他们的团队,一点一点的把体系建立起来,研发新的模型,形成新的技术,搭建整套系统,再进一步再更多数据上进行预测和分析,也就开始有更多的理解和认识。
印象深刻的是参加CAMEO之后不久的一周,我们第一次拿到了周第二,当时特别开心,团队成员受到了很大的鼓舞,感觉很长时间的辛苦没有白费,再后面更加振奋,有信心去对原来不懂的问题设计新的解决方案,逐渐的成绩越来越好,变得稳定起来。到现在,大家越做越兴奋,因为除了能看到性能的提升,还能看到在具体某些重要蛋白上预测结果的变化,有了生物学背景同学的帮助和分析,我们能获取更多模型上的理解和改进,大家真正体会到了学科交叉的乐趣,也对AI for Science的信心更坚定了。
量子位:AIRFold是从什么时候开始参加CAMEO竞赛的,刚开始就取得了好成绩么?
兰艳艳教授:团队最早是从今年的三月末开始第一次提交CAMEO结果,我们一开始制定的目标是实现一个系统化的结构预测解决方案,同时锻炼我们的团队,让大家对蛋白质的结构预测问题有更深刻的理解。
那时候我们已经有一些模型上的积累了,但是接触真正的实际数据还是第一次,并不是一开始就特别有效,从实际数据中发现了很多问题,帮助我们进一步去改进了模型,后来由于团队成员也并行的参与其他的研究和CASP15的比赛,我们在算法上积累了更多的经验。
六月末,我们把这些经验逐步的变成新算法加入我们CAMEO比赛的服务器,逐渐展示出来不错的表现。
量子位:我们注意到在AIRFold在研究院官网和微信公众号都是第一次出现,为什么选择这一时间亮相?
兰艳艳教授:包括AIRFold在内的蛋白质结构预测和设计等研究方向一直是我们的AIR智慧医疗组的重要研究方向。选择在现在公开AIRFold,一方面是展示我们在这一方向上持续布局投入的一个阶段性成果。
另外,单体的蛋白预测以及对于共进化信息的深入理解是我们团队后续开展在蛋白质以及大分子药物相关研究的基础,我们也希望利用这个契机增加和学界业界的交流与沟通,在结构与计算相关的领域持续发力,为AI赋能创新药物研发做出贡献。
量子位:官方消息说AIRFold的相关技术还在蛋白单点突变、多构象评估等问题取得初步进展,简单展开讲讲?
兰艳艳教授:在后AlphaFold2时代,其实蛋白结构预测已经是一个几乎被解决的问题了。虽然所谓孤儿序列(Orphan Sequence)的结构并不容易预测,但是从进化的角度,有重要生物学功能的序列几乎不可能是独立存在的,因此这并不是很大的问题。AIRFold团队在这样一个时间点开始研究蛋白质结构,我们更多的是想关注蛋白的结构如何决定功能,如何助力药物与疗法的研发,并不是只关注结构预测这一个孤立的问题。在这样一个背景下,我们就更加关注突变蛋白和多构象预测等问题。
蛋白点突变实际上和很多疾病是有关系的。我们现在耳熟能详的一些遗传病,比如囊性纤维化和家族性阿兹海默综合征都是由蛋白发生点突变导致的。现在AlphaFold2预测突变后蛋白的能力还很有限,主要因为来自单序列的突变信息会被MSA中信息掩盖住。如果一个点突变发生后导致原有的残基间的相互作用消失了,这类突变是相对容易研究的;AIRFold团队目前在这类问题中有一些进展,我们可以利用HomoMiner有针对性的破坏MSA中对应的信息。但是如果一个点突变导致这个残基可以和其他残基发生原来没有的相互作用,这也会影响蛋白的结构和功能,但是这一信息却不容易引入MSA中。我们目前也在聚焦这类问题对HomoMiner进行有针对性的优化和改进。
多构象预测其实是蛋白结构和功能以及药物研发之间的另一道鸿沟。我们知道无论是AlphaFold2预测的还是实验解析的蛋白结构其实都是静态的。但是在酶、离子通道等重要蛋白发挥活性的时候,他们都很发生非常大的构象变化,变化过程中不稳定的中间状态,往往可能是更好更高效的药物靶点。尽管通过分子动力学模拟的方法可以研究蛋白的构象变化,但是分子动力学模拟往往会消耗较大的计算资源,而且不容易研究时间尺度较大的过程。目前我们也积累了一些关于多构象预测的技术方法,我们团队通过深入分析AlphaFold在多构象任务上的不足,开发了一系列输入信息微调的流程,以可控的方式获得大量具有高度多样性的构象。比如说在刚刚结束的CASP15中,我们遇到了经典的蛋白激酶Scr蛋白变构的问题(T1197),我们就使用目前开发的方法获得了很多不同的构象。后续我们也会深入挖掘概率生成式模型在多构象预测上的潜力和可能性。
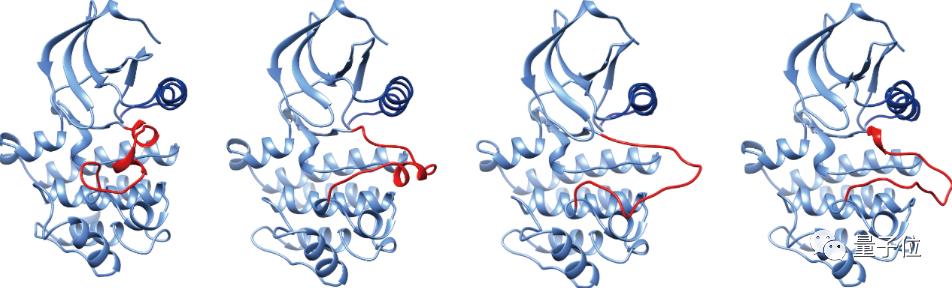
△CASP15比赛中的T1197蛋白
量子位:对AIRFold后续技术上还有哪些改进方向?又有哪些应用方向?
兰艳艳教授:AIRFold本身更关注于深入利用同源信息,提高通用的蛋白质结构预测预测表现。虽然相关的技术可以直接应用到更复杂的场景下,但还存在一些局限性。从我们的角度来看,我们更期待对于一些学界业界关注的重点难题有更加令人满意的解决方案,如对于抗体等特殊蛋白等处理,可变区域等预测,以及ligand和protein在结合状态的下的构象预测等。我们团队正在持续地推进这一部分的研究,同时我们也跟相关企业和科研单位进行密切交流,希望能够在大分子制药等方向找到共同感兴趣的关键性问题,并且开展深入合作。我们也期待未来有更多的优秀研究人员加入到这个新兴交叉科学领域,进一步发挥AI的价值。
团队介绍
AIRFold来自清华大学智能产业研究院兰艳艳教授团队。
兰艳艳教授毕业于中国科学院数学与系统科学研究院,获得概率论与数理统计专业理学博士学位,师从著名数学家马志明院士,研究方向为信息检索,机器学习和自然语言处理。

△AIRFold团队
清华大学智能产业研究院(AIR)AI+生命科学团队招聘博士后/科研工程师/实习生,主要从事AI for Science的交叉学科研究,利用深度学习、自然语言处理、信息检索等领域的前沿方法,解决交叉学科的各类挑战性问题,技术创新将落地在AI制药、健康计算等领域。
AIR将提供一流的科研平台与创新氛围,并提供有竞争力的薪酬。其中,本科和硕士实习生,有机会成为拟2023年入学的博士生候选人(团队多位老师有计算机系直博名额)。
指导教师:马维英教授/兰艳艳教授/周浩副教授
简历请发送至 airhr@air.tsinghua.edu.cn
邮件主题:医疗健康+姓名+申请职位
参考链接:
[1]https://mp.weixin.qq.com/s/ROeZYKvVJm-EBbhnDKZR1w
[2]https://air.tsinghua.edu.cn/info/1046/1200.htm
[3]https://www.sciencedirect.com/science/article/pii/S1097276522007547
— 完 —
「量子位·视点」直播报名
高阶智能驾驶的量产有什么特点?量产路线如何选择?落地进程又到了哪一阶段?
9月15日周四19:00,参与直播,为你解答~

点这里👇关注我,记得标星哦~
以上是关于清华AI蛋白质结构预测,连续4周夺得CAMEO第一的主要内容,如果未能解决你的问题,请参考以下文章
AlphaFold2被超越!中国团队刷新全球蛋白质结构预测纪录,大牛彭健创业项目一鸣惊人...
国产AI蛋白质结构预测再现突破,用单条序列解决3D结构,彭健团队:“AlphaFold2以来最后一块拼图补齐了”...
AI 最新突破集锦 AI控制核聚变 AI预测蛋白质3D结构 Alpha Fold2 AI证明数学公式 自动驾驶
AI 最新突破集锦 AI的瓶颈和进展 AI控制核聚变 AI预测蛋白质3D结构 Alpha Fold2 AI证明数学公式 自动驾驶 AI替代的硬件
AI 最新突破集锦 AI的瓶颈和进展 AI控制核聚变 AI预测蛋白质3D结构 Alpha Fold2 AI证明数学公式 自动驾驶 AI替代的硬件
AI 最新突破集锦 AI的瓶颈和进展 AI控制核聚变 AI预测蛋白质3D结构 Alpha Fold2 AI证明数学公式 自动驾驶 AI替代的硬件