群体遗传之ped格式
Posted djx571
tags:
篇首语:本文由小常识网(cha138.com)小编为大家整理,主要介绍了群体遗传之ped格式相关的知识,希望对你有一定的参考价值。
1、PED简介
PED文件格式是广泛使用的用于连锁系谱数据分析的格式,并用作plink程序的输入。PLINK是一个免费的,开源的全基因组关联分析工集,旨在以高计算效率的方式执行一系列基本的,大规模的分析。PED能够处理二倍体SNP数据。
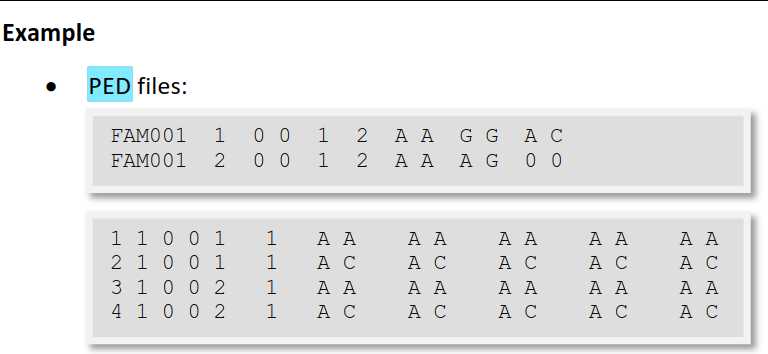
空格(空格或制表符)分隔的文本文件*.ped 每一行对应一个individual 以下前6列是必须的(id是字母数字): o Family ID (Family ID用来表示家族,同一个家族用同一个family ID表示) o Individual ID (用来表示个体,family ID和Individual ID连起来必须能够唯一表示每个样本) o Paternal ID (表示父本ID,) o Maternal ID (母本ID,) o Sex (1代表male,2代表female, 其他数字表示unknown。) o Phenotype (代表表型,其中表型可以是离散型的(比如关联性状),也可以是连续型的(比如数量性状),plink会自动识别对应的类型。通过以上6个必须的字段,可以完整的映射到某一性状的家系图上。) 关联性状应该这样编码: o -9 missing o 0 missing o 1 unaffected o 2 affected column 7 onwards: Genotypes (对于关联分析而言,除了表型相关信息,还需要基因型信息) any character (e.g.: 1,2,3,4 or A,C,G,T or anything else) missing genotype: 0 所有的标记必须是双等位的(二倍体)。要么两个等位基因都缺失,要么两者都不缺失。单倍体数据:编码为二倍体纯合子。两个等位基因依次出现。 Comments: line starts with #
在ped文件中,每个snp位点的基因型需要两列来表示,分别表示major allel 和 minor allel。在表示基因型时,既可以使用A,C,G,T字母的形式,也可以采用1,2数字编码的形式。默认情况下,用0来表示基因型的缺失。
2、MAP简介
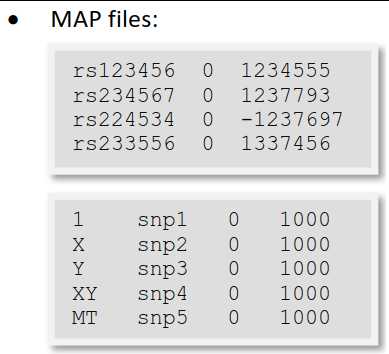
MAP文件的每一行描述一个 single marker且必须包含4列:
chromosome (1-22, X, Y, MT or 0 if unplaced) #染色体编号为数字, 未知为0 rs# or snp identifier #SNP名称为字符或数字, 可以从1编号, 注意要和bed文件SNP列一一对应 Genetic distance (morgans) (missing: 0) #遗传距离(摩尔) SNP物理坐标
MAP必须包含与PED文件中一样多的markers。‘
PED文件中的标记不需要按照基因组顺序排列,但是MAP应该与PED文件maker顺序一致
PGDSpider软件中对于不同的文件格式有一个详细的说明。
http://pngu.mgh.harvard.edu/~purcell/plink/data.shtml#ped
以上是关于群体遗传之ped格式的主要内容,如果未能解决你的问题,请参考以下文章