植物GO注释
Posted zhanmaomao
tags:
篇首语:本文由小常识网(cha138.com)小编为大家整理,主要介绍了植物GO注释相关的知识,希望对你有一定的参考价值。
本文主要是对没有GO term库的植物进行注释。
1、选用AgriGo 进行注释,在agriGO中点击species后,查看与你目标物种相近的物种作为库

2、比如我以甜菜为例
 为了找到和GO term对应的ID,先找到PLAZA,进入网站https://bioinformatics.psb.ugent.be/plaza/versions/plaza_v3_dicots/download/index
为了找到和GO term对应的ID,先找到PLAZA,进入网站https://bioinformatics.psb.ugent.be/plaza/versions/plaza_v3_dicots/download/index
点击data ->identifier Conversion, 找到甜菜,下载改ID对应的文件,进而可以确定甜菜基因组版本,并进行写脚本更换ID
3、将DGE更换好的ID输入AgriGO中,即可获得差异基因,可点击downlodw下载,
选取FDR<=0.05, p <0.05进行作图
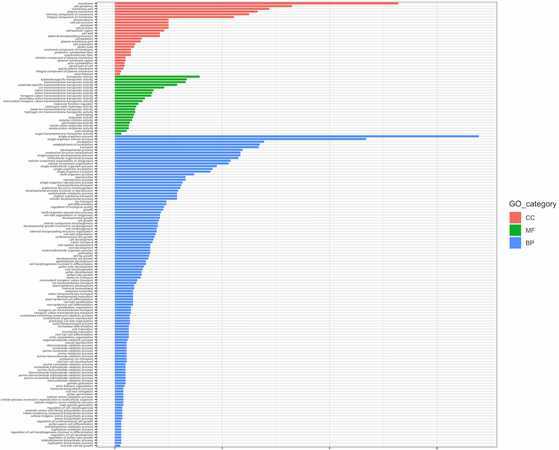
作图
使用ggplot2
rm(list = ls())
library(ggplot2)
data <- read.table("Go_input.txt",header = T,sep = " ")
attach(data)
dorder=factor(as.integer(row.names(data)),labels = data$Term) ###sorted by the Term,
p <- ggplot(data,aes(x=dorder,y=queryitem,fill=term_type))+geom_bar(stat = "identity",width = 0.8)+
xlab("") + ylab("Gene count") +theme_bw()+
scale_fill_discrete(name="GO_category",breaks=c("C","F","P"),labels=c("CC","MF","BP"))+ ##change the legend text
theme(panel.background = element_rect(fill = "transparent",colour = NA),axis.text.y = element_text(size = 7),axis.text.x = element_text(face = "bold",angle = 70,vjust = 1,hjust = 1,size = 7))
以上是关于植物GO注释的主要内容,如果未能解决你的问题,请参考以下文章