非模式物种的注释
Posted djx571
tags:
篇首语:本文由小常识网(cha138.com)小编为大家整理,主要介绍了非模式物种的注释相关的知识,希望对你有一定的参考价值。
一句话:我们经常会面对非模式物种的GO或者KEGG富集与注释。
1、载入我们所需要的包
if("clusterProfiler" %in% rownames(installed.packages()) == FALSE) {source("http://bioconductor.org/biocLite.R");biocLite("clusterProfiler")}
biocLite("colorspace")
suppressMessages(library(clusterProfiler))
ls("package:clusterProfiler")
library(AnnotationHub)
library(biomaRt)
library(enrichGO)
2、收索目标物种
hub <- AnnotationHub::AnnotationHub() query(hub, "Apis cerana")
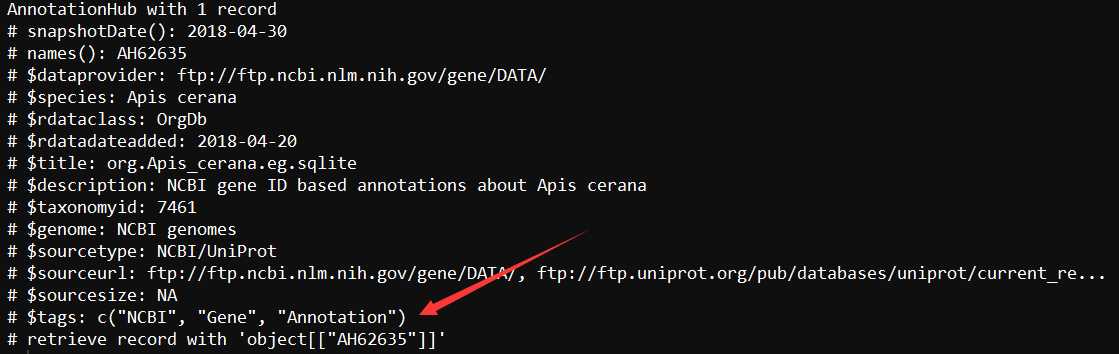
3、抓取目标OrgDb
Apis_cerana.OrgDb <- hub[["AH62635"]] columns(Apis_cerana.OrgDb) Apis_cerana.OrgDb
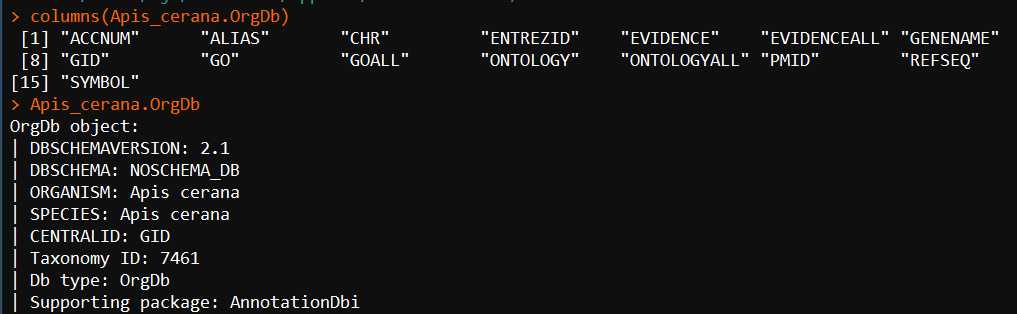
4、抓取目标OrgDb
keys(Apis_cerana.OrgDb)
head(keys(Apis_cerana.OrgDb,keytype = "SYMBOL")) # 查看注释信息的symbol
hainan_genelist =read.table(‘C:/Users/djx/Desktop/海南.txt‘,header=TRUE,sep=‘ ‘)
head(hainan_genelist)
colnames(hainan_genelist)
rownames(hainan_genelist)
bitr(keys(Apis_cerana.OrgDb)[15], ‘SYMBOL‘, c("ENTREZID","REFSEQ", "GO", "ONTOLOGY"), Apis_cerana.OrgDb)
hainan_geneid=as.character(hainan_genelist$Gene.ID)
hainan_geneid[1]
str(hainan_geneid)
rm(hainan_geneid)
sample_genes <- keys(Apis_cerana.OrgDb)[1:100]
hainan_enrich.go = enrichGO(sample_genes,
OrgDb = Apis_cerana.OrgDb,
keyType = ‘ENTREZID‘,
#ont= "BP",
pvalueCutoff = 0.05,
qvalueCutoff = 0.1,
readable = T)
以上是关于非模式物种的注释的主要内容,如果未能解决你的问题,请参考以下文章