R_Studio(癌症)以等宽类别值自定义类别值等频类别值(分为5类)
Posted 1138720556gary
tags:
篇首语:本文由小常识网(cha138.com)小编为大家整理,主要介绍了R_Studio(癌症)以等宽类别值自定义类别值等频类别值(分为5类)相关的知识,希望对你有一定的参考价值。
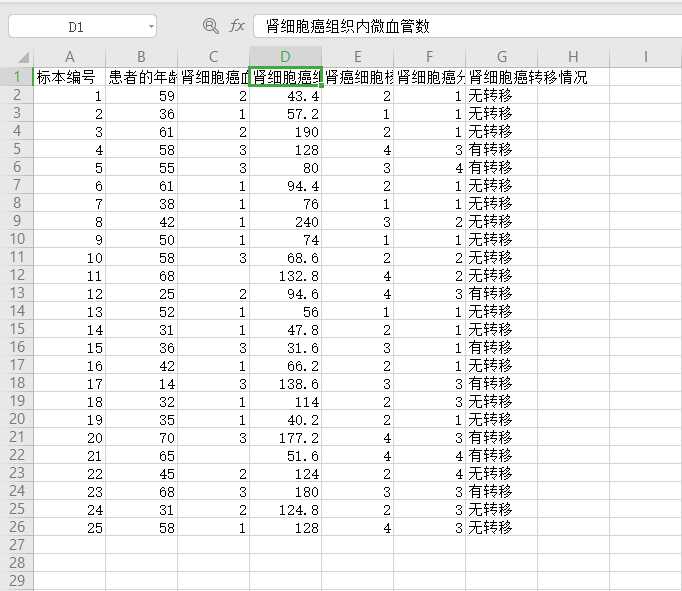
对“癌症.csv”中的肾细胞癌组织内微血管数进行连续属性的离散化处理
增加“微血管数分类1”属性,取值为等宽类别值(分为5类),增加“微血管数分类2”属性,取值为自定义类别值(0~40,41~60,61~120,121~150,151~200,201~250),增加“微血管数分类3”属性,取值为等频类别值(分为5类)

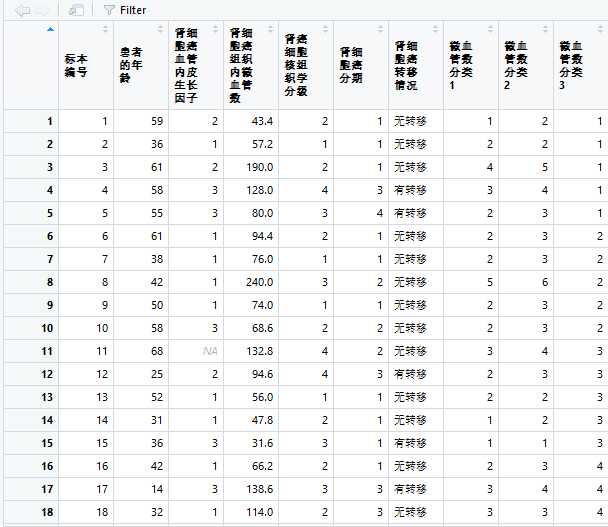
setwd(‘D:\\data‘) list.files() #读取数据 dat=read.csv(file="癌症.csv",header=TRUE) #等宽类别值 v1=dat[,4]/max(dat[,4])*5 v1=ceiling(v1)#将得到的值向上取整 dat=data.frame(dat,‘微血管数分类1‘=v1)#变量重命名,存入数据 #自定义类 c2=c(0,40,60,120,150,200,250) v2=cut(dat[,4],c2,labels = F,right = T) dat=data.frame(dat,‘微血管数分类2‘=v2)#变量重命名,存入数据 names(dat)=c("f1","f2","f3","f4","f5","f6") #变量重命名 #attach(data) n=length(dat[,1]) k=5#等频划分为5组 m=length(k) data=dat[order(dat$f4),]#按大小排序作为离散化依据 v3=rep(1:k, each = n/k, len = n)#定义新变量 dat=data.frame(dat,"微血管数分类3"=v3)
实现过程
增加“微血管数分类1”属性,取值为等宽类别值(分为5类)
v1=dat[,4]/max(dat[,4])*5 v1=ceiling(v1)#将得到的值向上取整 dat=data.frame(dat,‘微血管数分类1‘=v1)#变量重命名,存入数据
输出:[1] 1 2 4 3 2 2 2 5 2 2 3 2 2 1 1 2 3 3 1 4 2 3 4 3 3
(区间值 / max()*等宽 分类 控制数值区间在1~5)
增加“微血管数分类2”属性,取值为自定义类别值(0~40,41~60,61~120,121~150,151~200,201~250)
c2=c(0,40,60,120,150,200,250) v2=cut(dat[,4],c2,labels = F,right = T) dat=data.frame(dat,‘微血管数分类2‘=v2)#变量重命名,存入数据
输出: [1] 2 2 5 4 3 3 3 6 3 3 4 3 2 2 1 3 4 3 2 5 2 4 5 4 4
cut()函数:切割将x的范围划分为时间间隔
参数:
breaks:两个或更多个唯一切割点或单个数字(大于或等于2)的数字向量,给出x被切割的间隔的个数。
breaks:采用fivenum():返回五个数据:最小值、下四分位数、中位数、上四分位数、最大值。
labels:为区间数,打标签
right=T:右区间取闭区间(10,20]
增加“微血管数分类3”属性,取值为等频类别值(分为5类)
n=length(dat[,1]) k=5#等频划分为5组 m=length(k) data=dat[order(dat$肾细胞癌组织内微血管数),]#按大小排序作为离散化依据 v3=rep(1:k, each = n/k, len = n)#定义新变量 dat=data.frame(dat,"微血管数分类3"=v3)
输出:[1] 1 1 1 1 1 2 2 2 2 2 3 3 3 3 3 4 4 4 4 4 5 5 5 5 5
rep()函数 传送门

rep(x, ...)
rep.int(x, times)
rep_len(x, length.out)
rep函数有4个参数:
x:向量或者类向量的对象
each:x元素每个重复次数
times:each后的向量的处理,如果times是单个值,则each后的值整体重复times次数,如果是x each后的向量相等长度的向量,则对each后的每个元素重复times同一位置的元素的次数,否则会报错
length.out指times处理后的向量最终输出的长度,如果长于生成的向量,则补齐。也就是说rep会先处理each参数,生成一个向量X1,然后times再对X1进行处理生成X2,length.out在对X2进行处理生成最终输出的向量X3> rep(1:4,times=c(1,2,3,4)) #与向量x等长times模式
[1] 1 2 2 3 3 3 4 4 4 4
> rep(1:4,times=c(1,2,3)) #非等长模式,出现错误
Error in rep(1:4, times = c(1, 2, 3)) : invalid ‘times‘ argument
> rep(1:4,each=2,times=c(1,2,3,4)) #还是非等长模式,因为each后的向量有8位,而不是4位
Error in rep(1:4, each = 2, times = c(1, 2, 3, 4)) :
invalid ‘times‘ argument
> rep(1:4,times=c(1,2,3,4)) #等长模式,我写重了o(╯□╰)o
[1] 1 2 2 3 3 3 4 4 4 4
> rep(1:4,times=c(1,2,3,4),each=3) #重复的例子啊,莫拍我
Error in rep(1:4, times = c(1, 2, 3, 4), each = 3) :
invalid ‘times‘ argument
> rep(1:4,each=2,times=1:8) #正确值,times8位长度向量
[1] 1 1 1 2 2 2 2 2 2 2 3 3 3 3 3 3 3 3 3 3 3 4 4 4 4 4 4 4 4 4 4 4 4 4 4 4
> rep(1:4,each=2,times=1:8,len=3) #len的使用,循环补齐注意下
[1] 1 1 2
> rep(1:4,each=2,times=3) #先each后times
[1] 1 1 2 2 3 3 4 4 1 1 2 2 3 3 4 4 1 1 2 2 3 3 4 4
>rep函数完毕!
以上是关于R_Studio(癌症)以等宽类别值自定义类别值等频类别值(分为5类)的主要内容,如果未能解决你的问题,请参考以下文章
R_Studio(cart算法决策树)对book3.csv数据用测试集进行测试并评估模型
