机器视觉——医学检测
Posted zhangmingfeng
tags:
篇首语:本文由小常识网(cha138.com)小编为大家整理,主要介绍了机器视觉——医学检测相关的知识,希望对你有一定的参考价值。
利用openCV或其他工具编写程序实现细化血管并输出血管轮廓图像的功能。
实现过程
1、编写程序
目标图片如下
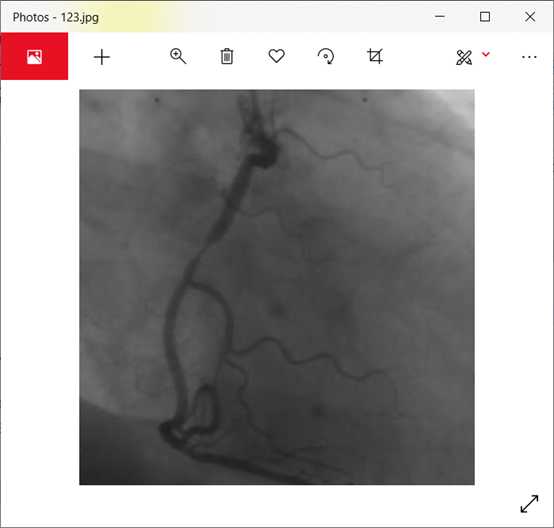
根据展示的程序功能编写对应的程序:
第一步,读取显示图像的功能openCV已经提供了函数imread()和imshow(),代码如下
img1=cv2.imread(‘123.jpg‘)
cv2.imshow(‘origin‘,img1)
cv2.waitKey(0)
第二步,使用open CV自带的局部阈值的方法adaptiveThreshold()处理原图片,得到一张带“杂质”的血管二值图,需要注意的是这个方法只能处理灰度图,所以我们需要先将原图进行灰度处理:
gray = cv2.cvtColor(img1, cv2.COLOR_RGB2GRAY)
binary = cv2.adaptiveThreshold(gray, 255, cv2.ADAPTIVE_THRESH_GAUSSIAN_C,cv2.THRESH_BINARY, 31,2.7)
cv2.namedWindow("binary1", cv2.WINDOW_NORMAL)
cv2.imshow("binary1", binary)
cv2.waitKey(0)
第三步,对图像binary进行反色处理:
def pointInvert(img):
point = img.copy()
for i in range(0, img.shape[0]):
for j in range(0, img.shape[1]):
point[i,j] = 255 - img[i,j]
return point
img2 = pointInvert(binary)
cv2.imshow("img2", img2)
cv2.waitKey(0)
第四步,扩展图片,将图片四周向外扩展十个像素单位:
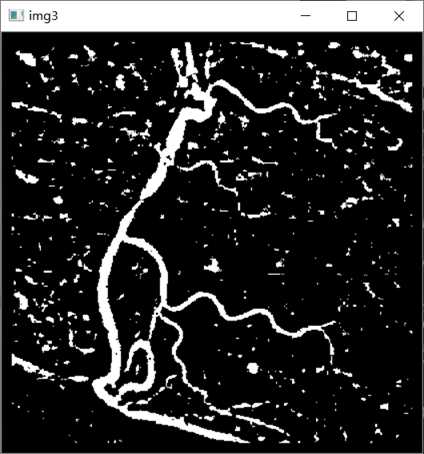
img3 = cv2.copyMakeBorder(img2,10,10,10,10, cv2.BORDER_CONSTANT,value=[0,0,0])
cv2.imshow("img3 ", img3 )
cv2.waitKey(0)
第五步,对图像img3进行轮廓查找操作,寻找处理后的图片的轮廓:
contours,hierarchy = cv2.findContours(img3, cv2.RETR_CCOMP, cv2.CHAIN_APPROX_SIMPLE)
for i in range(len(contours)):
area = cv2.contourArea(contours[i])
if area < 700:
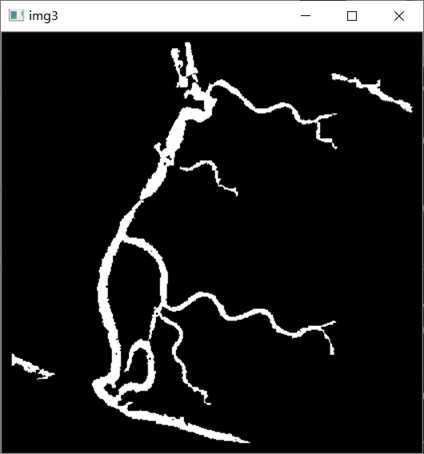
cv2.drawContours(img3,[contours[i]],0,0,-1)
cv2.imshow("img3 ",img3 )
cv2.waitKey(0)
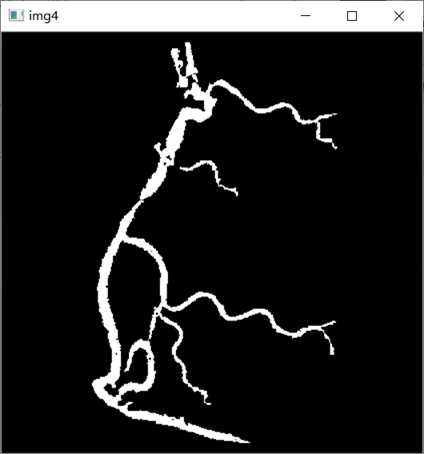
第六步,输出给定范围内查找到的连通图:
img4=img3.copy()
contours1,hierarchy = cv2.findContours(img4, cv2.RETR_CCOMP, cv2.CHAIN_APPROX_SIMPLE)
for j in range(len(contours1)):
area1 = cv2.contourArea(contours1[j])
print(area1)
if area1 ==155.0:
cv2.drawContours(img4,[contours1[j]],0,0,-1)
elif area1==266.5:
cv2.drawContours(img4,[contours1[j]],0,0,-1)
elif area1==437.0:
cv2.drawContours(img4,[contours1[j]],0,0,-1)
cv2.imshow(‘img4‘,img4)
cv2.waitKey(0)
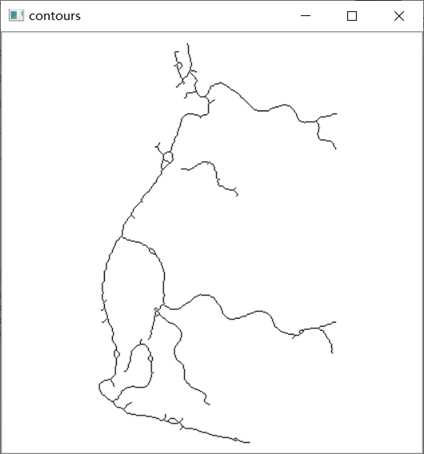
第七步,查找最大连通图的轮廓并输出:
temp=temp=np.temp = np.ones(a.shape,np.uint8)
contours1, hierarchy = cv2.findContours(img4, cv2.RETR_CCOMP, cv2.CHAIN_APPROX_SIMPLE)
for idx in range(len(contours)):
if verify(contours[idx],150,10000):
cv2.drawContours(temp,contours,idx,(255,255,255),1)
cv2.imshow("contours",temp)
cv2.waitKey(0)
第八步,对temp图像进行细化操作,这里我使用的是索引表的方式,在每行水平扫描时,先判断每一点的左右像素,如果都是黑点,则该点不做处理。如果某个黑点被删除了,则跳过它的右边的像素点,处理下一个点。再将水平扫描改为垂直扫描重复上一个操作。
多次重复以上的步骤,直到图像不再变化就得到了我们所需要的骨架图。
def VThin(image, array):
h,w= image.shape[:2]
NEXT = 1
for i in range(h):
for j in range(w):
if NEXT == 0:
NEXT = 1
else:
M = image[i, j-1] + image[i,j] + image[i, j+1] if 0<j<w-1 else 1
if image[i, j] == 0 and M != 0:
a = [0] * 9
for k in range(3):
for l in range(3):
if-1<(i-1+k)<h and -1<(j-1+l)<w and image[i-1+k, j-1+l] == 255:
a[k*3 + l] = 1
sum = a[0]*1 + a[1]*2 + a[2]*4 + a[3]*8 + a[5]*16 + a[6]*32 + a[7]*64 + a[8]*128
image[i,j] = array[sum]*255
if array[sum] == 1:
NEXT = 0
return image
def HThin(image, array):
h, w = image.shape[:2]
NEXT = 1
for j in range(w):
for i in range(h):
if NEXT == 0:
NEXT = 1
else:
M = image[i-1, j] + image[i, j] + image[i+1, j] if 0<i<h-1 else 1
if image[i, j] == 0 and M != 0:
a = [0] * 9
for k in range(3):
for l in range(3):
if -1<(i-1+k)<h and -1<(j-1+l)<w and image[i-1+k, j-1+l] == 255:
a[k*3 + l] = 1
sum = a[0]*1 + a[1]*2 + a[2]*4 + a[3]*8 + a[5]*16 + a[6]*32 + a[7]*64 + a[8]*128
image[i, j] = array[sum] * 255
if array[sum] == 1:
NEXT = 0
return image
def Xihua(binary, array, num=10):
iXihua = binary.copy()
for i in range(num):
VThin(iXihua, array)
HThin(iXihua, array)
return iXihua
iThin = Xihua(img6, array)
cv2.imshow(‘contours‘, iThin)
cv2.waitKey(0)
第九步,获取iThin图像的轮廓:
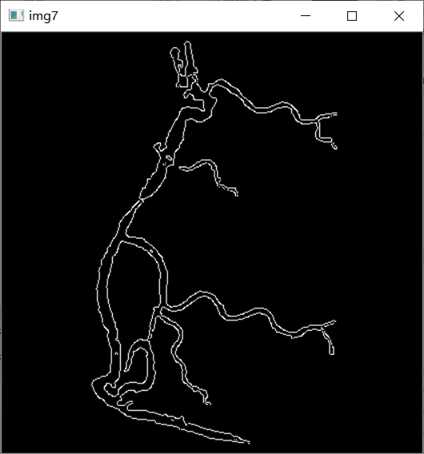
img7 = cv2.Canny(iThin,80,255)
cv2.imshow(‘img7‘, img7)
cv2.waitKey(0)
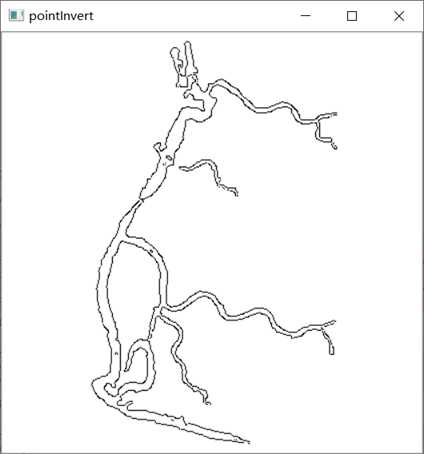
第十步,对图像img7进行反色处理:
point = pointInvert(img7)
cv2.imshow(‘pointInvert‘,point)
cv2.waitKey(0)
运行结果
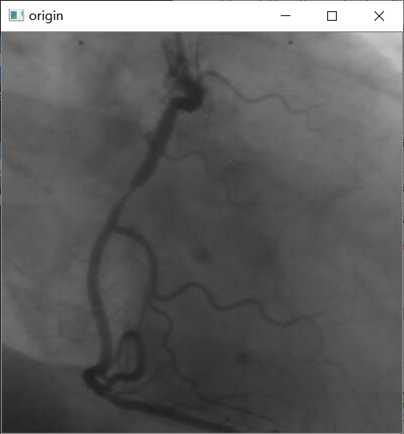
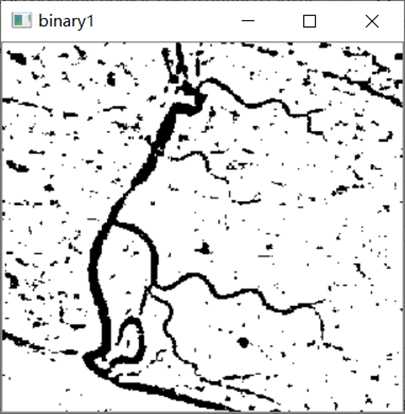
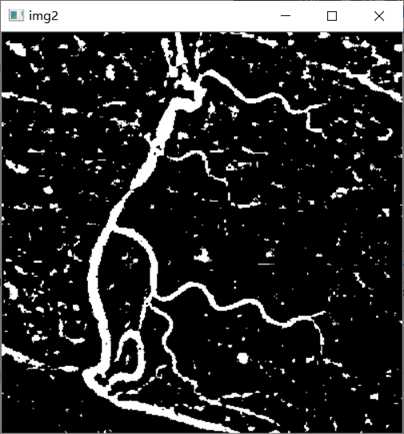
问题及解决方法
1、二值化
刚开始处理原图片时,使用openCV自带的二值化处理方法得到的结果总是不尽如人意,图片的下半区始终是一整块的黑块,没有办法进行下一步的处理,后来又使用增强对比度的方法来完成这一步的操作,发现下半区的问题还是没有解决。后来抱着试一试的心态搜索open CV中的Niblack处理,发现有类似这个操作的局部阈值的方法,最后使用这个方法成功完成图像二值化的步骤。
2、部分“杂质”消除不掉
这个问题在我更改了findContours()函数中的参数无果后,我选择了一个很傻的方法:循环输出图像输出区域的面积,同时显示当时的图片,知道了需要的连通图面积后给定面积的值输出仅需要的连通图。
实验总结
这一次的实验与之前的其次实验相比难了很多,代码的难度、逻辑思维的难度以及编写程序的过程中出现的问题的难度,都大大提高了。很开心的是我顺利完成了这次实验,不足的是实验中的问题解决的并不完美,比解决消除图像中的“杂质”这一步出现的部分“杂质”消除不掉的问题,我使用的是最傻的办法。希望以后可以改进这一部分的缺陷。
以上是关于机器视觉——医学检测的主要内容,如果未能解决你的问题,请参考以下文章