提取bam/sam文件指定区域reads
Posted
tags:
篇首语:本文由小常识网(cha138.com)小编为大家整理,主要介绍了提取bam/sam文件指定区域reads相关的知识,希望对你有一定的参考价值。
参考技术A 若想要从sam或bam文件中提取指定区域内的reads,可以使用samtools和bedtools来实现。 首先准备一个区域信息文件。region.bed #第一列为染色体ID,第二三列分别为起始终止位置
若想要从sam或bam文件中提取指定区域内的reads,可以使用samtools或bedtools来实现。
使用samtools
使用bedtools
target.bed
认识Panda3D引擎bam相关命令
看一下Panda自带命令,其中有bam相关的,来了解一下;
输入一个命令看一下,提示需要输入一个bam文件名;
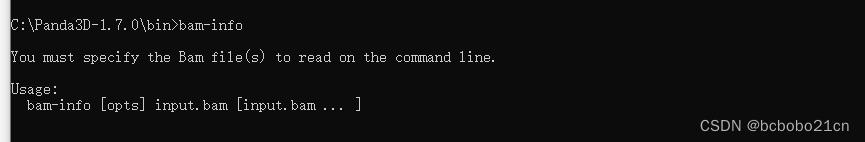
查一下,查到一个介绍一种bam文件的资料如下,
SAM (Sequence Alignment/Map) 格式是一种通用的比对格式,用来存储reads到参考序列的比对信息。
SAM是sanger制定,是以TAB为分割符的文本格式。主要应用于测序序列mapping到基因组上的结果表示,当然也可以表示任意的多重比对结果。
BAM文件
通过BGZF格式压缩后的SAM文件,一种二进制文件,可以直接理解为对SAM文件的压缩。
BAM文件无法直接用less、cat、head、tail等常规命令打开,可用samtools -view sample.bam命令打开
上面命令需要的bam文件是否是这个?
资料说,
Bam文件的head部分以"@"开头的行,包含有"@HD"、"@SQ"、"@RG"、"@PG" 等。
@HD行表示整个文件信息,VN表示格式版本,SO表示排序信息,支持queryname、coordinate、unknown、unsorted。
@SQ行记录了参考序列的信息,一般三列,分别是@SQ、RNAME、Sequence_length。
@RG记录了样本名,测序平台等信息,内容与bwa生成bam文件时的-R参数后更的内容一致。
用记事本打开一个bam文件,
再用十六进制编辑器打开看看,

Panda的bam-info命令是查看bam文件信息;看一下,
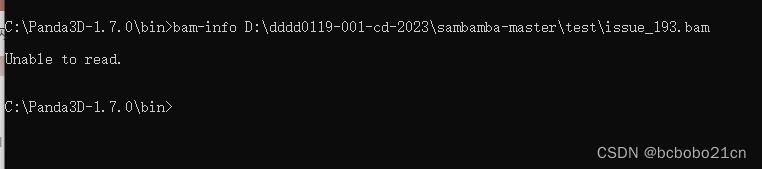
不能读上面的示例bam文件;
Panda还有一个命令是转换egg文件为bam文件,egg是panda自身的模型文件格式,可以和3dmax和maya等相互转换,
egg2bam -ps rel -o bamFileName.bam eggFileName.egg
那么这里的bam也应是一种3d模型文件,并非前面的示例bam文件;
查一下资料,常见3d模型文件格式里面没有bam;
看一下panda手册;
Converting Egg to Bam
Panda’s native egg file format is human-readable. This is convenient, but the files can get very large, and they can a little bit slow to load. To accelerate loading, Panda supports a second native format, bam. These files are smaller and are loaded very rapidly, but they cannot be viewed or edited in a text editor. Also, bam files are specific to the version of Panda they are created with, so they are not a good choice for long-term storage of your models.
转换Egg到Bam
Panda自己的egg文件格式是可读的。这是方便的,但是文件可能比较大,加载会有延迟。为了加速加载,Panda支持第二种格式,bam。
native format,应翻译为自带格式,bam是Panda3d引擎自带的第二种格式,第一种是egg。
bam是panda自带的二种模型文件格式之一;如果egg格式加载慢,可转换为bam格式;
操作一下看一下,
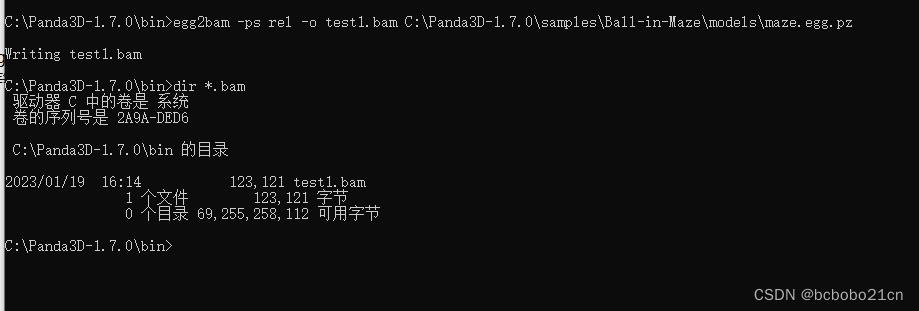
出来一个bam文件;
以上是关于提取bam/sam文件指定区域reads的主要内容,如果未能解决你的问题,请参考以下文章