图像分割U-Net: Convolutional Networks for Biomedical Image Segmentation
Posted coldMoon
tags:
篇首语:本文由小常识网(cha138.com)小编为大家整理,主要介绍了图像分割U-Net: Convolutional Networks for Biomedical Image Segmentation相关的知识,希望对你有一定的参考价值。
| 原始题目 | U-Net: Convolutional Networks for Biomedical Image Segmentation |
|---|---|
| 中文名称 | U-Net:用于生物医学图像分割的卷积网络 |
| 发表时间 | 2015年5月18日 |
| 平台 | Miccai 2015 |
| 来源 | University of Freiburg, Germany |
| 文章链接 | https://arxiv.org/pdf/1505.04597.pdf |
| 开源代码 | 官方实现: https://lmb.informatik.uni-freiburg.de/people/ronneber/u-net/ (Caffe) |
摘要
人们普遍认为,深度网络的成功训练需要数千个标注的训练样本。
本文提出一种网络和训练策略,依赖于数据增强的强大使用,以更有效地使用可用的标注样本。
该架构由 捕获上下文的收缩路径和能够精确定位的对称扩展路径(a contracting path to capture
context and a symmetric expanding path that enables precise localization) 组成。
这样的网络可以从 很少的图像 中进行端到端训练,并在 ISBI challenge 中对电子显微镜堆栈中的神经元结构进行分割的性能优于之前的最佳方法(滑动窗口卷积网络 a sliding-window convolutional network)。
使用在透射光学显微镜图像(phase contrast and DIC)上训练的相同网络,我们在这些类别中以较大优势赢得了 2015 年 ISBI 细胞跟踪挑战赛。
此外,网络速度很快。在最近的 GPU 上,512x512 图像的分割耗时不到一秒。
总结:
- 数据增强
- contracting path 捕获上下文, symmetric expanding path 能够精确定位
- 需要的图像更少,可以端到端训练
- 性能好,速度快(最近的 GPU 上,512x512 图像的分割耗时不到一秒。)
5. 结论
u-net 架构在不同的生物医学分割应用上取得了非常好的性能。
由于使用 弹性变形(elastic deformations) 进行数据增强,它只需要很少的标注图像,并且在 NVidia Titan GPU (6 GB) 上的训练时间仅为10 小时。
给出了基于 Caffe[6]的完整实现和经过训练的网络。
我们确信 u-net 架构可以轻松地应用于更多的任务。
总结:
- 数据增强:elastic deformations,需要很少的标注图像
- 训练时间短
- 最重要的还是可以用于别的任务。
2. 网络架构
网络架构如 图1 所示。

图1所示。U-net架构(例如最低分辨率为 32x32 像素)。每个蓝色框对应于一个多通道特征图。通道数在方框的顶部表示。x-y 尺寸在 box 的左下边缘。白色框表示复制的特征映射。箭头表示不同的操作。
它由 一个收缩路径(contracting path)(左侧)和 一个扩展路径(expansive path)(右侧)组成。
收缩路径 遵循卷积网络的典型架构。
- 它由两个 3x3 卷积(无填充卷积)的重复应用组成,每个卷积后面都有一个 ReLU 和一个 stride 2 的 2x2 max pooling 操作用于下采样。
- 在每个下采样步骤,我们将 特征通道的数量 增加一倍。
扩展路径中的每一步都包括对特征图的上采样,然后是将特征通道数量减半的 2x2 卷积(“up-convolution”),与 收缩路径中相应裁剪的特征图的 concatenation,以及两个 3x3 卷积,每个卷积后面都是一个 ReLU。
裁剪是必要的,因为在每个卷积中会丢失边界像素。
在最后一层,使用 1x1 卷积将每个 64 个分量的特征向量映射到所需的类别数量。该网络总共有 23 个卷积层。
为了允许输出分割图的 无缝拼块(seamless tiling)(参见 图2 ),选择输入 分块(tile) 大小很重要,以便将所有 2x2 max pooling 操作应用于具有偶数 x 和 y 大小的层。
总结:
- 架构图一目了然
- 使用的是 无填充卷积
- 注意其中有个裁剪特征的操作
3. 训练
利用 输入图像及其对应的分割图,采用 随机梯度下降 实现Caffe[6]来训练网络。由于无填充卷积,输出图像比输入图像小一个恒定的边界宽度。
为了最小化开销并最大限度地利用 GPU 内存,我们倾向于使用较大的输入图像块,而不是较大的 batch size ,从而将 batch 减少为单个图像。因此,我们使用高 momentum(0.99),以便之前看到的大量训练样本确定当前优化步骤中的更新。
能量函数 通过在 最终特征图上 pixel-wise soft-max 并结合 cross entropy loss function 来计算。
soft-max 被定义为 \\(p_k(\\mathbfx)=\\operatornameexp(a_k(\\mathbfx))/\\left(\\sum_k^\\prime=1^K\\operatornameexp(a_k^\\prime(\\mathbfx))\\right)\\),其中 \\(a_k\\) 表示在 像素位置 \\(\\textbfx\\in\\Omega\\ \\ \\textwith\\ \\ \\Omega\\subset\\mathbbZ^2\\) 的 特征通道 k 中的激活函数。
K 是类别的数量,\\(p_k(x)\\) 是 approximated maximum-function。
即: 对于具有最大激活 \\(a_k\\) 的 k, \\(p_k(x) ≈1\\); 对于所有其他k, \\(p_k(x) ≈ 0\\)。
然后,cross entropy 在每个位置惩罚 \\(p_\\ell(\\textbfx)(\\textbfx)\\) 与 1 的 误差(deviation)。
这里 \\(\\ell:\\Omega\\to\\1,\\ldots,\\textK\\\\) 是每个像素的标签, \\(w:\\Omega\\rightarrow\\mathbbR\\) 是一个权重图,我们引入它是为了在训练中赋予一些像素更多的重要性。
我们预先计算每个 ground truth 分割的权重图,以弥补训练数据集中特定类别像素的不同频率,并迫使网络学习我们在接触细胞之间引入的 小分离边界(参见 图3c和d )。
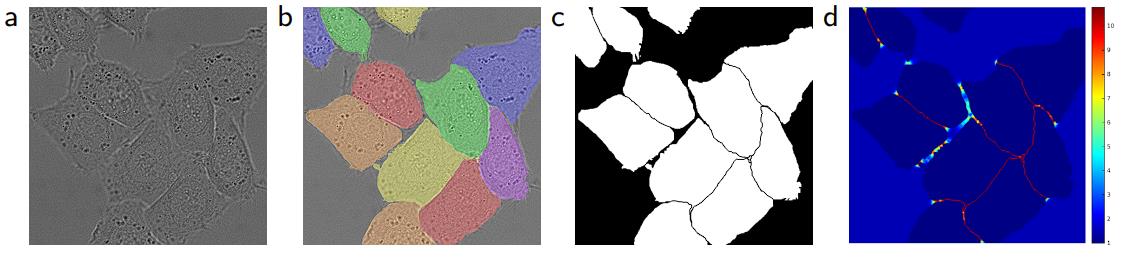
图3所示。用 DIC (differential interference contrast) microscopy 记录玻璃上的 HeLa 细胞。(a) 原始图像。(b) 覆盖 ground truth 分割。不同的颜色表示 HeLa 细胞的不同实例。(c) 生成的分割 mask(白色:前景,黑色:背景)。(d)使用 pixel-wise loss 权重进行映射,以迫使网络学习边界像素。
分离边界使用形态学操作计算。weight map 的计算如下:
其中 \\(w_c: Ω→R\\) 是平衡类频率的 weight map,\\(d_1: Ω→R\\) 表示到最近单元格边界的距离,\\(d_2: Ω→R\\) 到第二最近单元格边界的距离。在我们的实验中,我们设置 \\(w_0 = 10, σ≈5\\) 像素。
在具有许多卷积层和不同路径的深度网络中,良好的 权值初始化 是非常重要的。
否则,网络的某些部分可能会过度激活,而其他部分则永远不会做出贡献。
理想情况下,初始权重应该调整,使网络中的每个特征图都具有近似的单位方差。
对于具有我们的架构(alternating convolution and ReLU layers)的网络,这可以通过从标准差为\\(\\text\\sqrt2/N\\) 的高斯分布中提取初始权重来实现,其中 N 表示一个神经元[5]的传入节点数。
例如,对于一个 3x3 卷积和 前一层中的 64 个特征通道,N = 9·64 = 576。
3.1 数据增强
在只有少量训练样本的情况下,数据增强对于让网络获得所需的 invariance 和 robustness 至关重要。
在显微图像中,我们主要需要 平移和旋转不变性,以及对变形和灰度变化的鲁棒性。特别是训练样本的随机弹性变形似乎是用很少的标注图像训练分割网络的关键概念。我们在一个粗糙的 3 × 3网格 上使用随机位移向量生成平滑变形。位移从具有 10像素 标准差的高斯分布中采样。然后使用 双三次插值 计算每个像素的位移。收缩路径(contracting path) 末尾的 Drop-out layers 执行进一步的隐式数据增强。
4. 实验
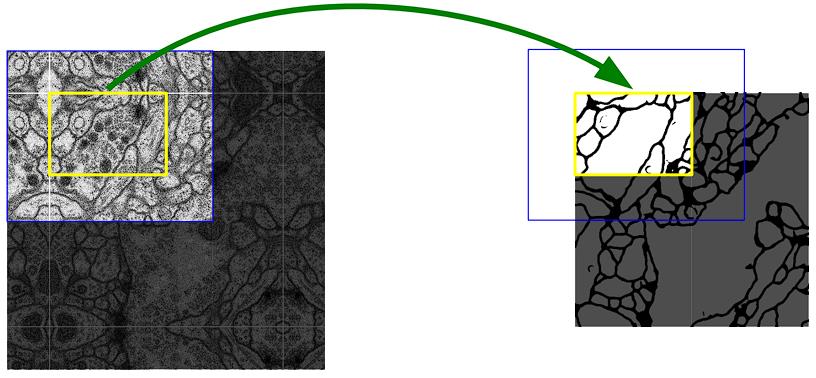
- 数据集1:EM segmentation challenge [14] that was started at ISBI 2012 。训练集: a set of 30 images (512x512 pixels) 。Each image comes with a corresponding fully annotated ground truth segmentation map for cells (white) and membranes (black). 测试集标签未公开,需要发给比赛组织者。
- 数据集2:ISBI cell tracking challenge 2014 and 2015 的 光显微图像中的细胞分割任务。“PhC-U373” 和 DIC-HeLa 两个数据集。
参考:
以上是关于图像分割U-Net: Convolutional Networks for Biomedical Image Segmentation的主要内容,如果未能解决你的问题,请参考以下文章
U-Net: Convolutional Networks for Biomedical Image Segmentation