一文读懂FastQC Report
Posted
tags:
篇首语:本文由小常识网(cha138.com)小编为大家整理,主要介绍了一文读懂FastQC Report相关的知识,希望对你有一定的参考价值。
参考技术A总览,来判断测序质量
Encoding :测序平台信息,我也不知道这个样本的测序信息对不对,所以这里就没有办法深入解释了。
Total Sequences :测序获得的reads数目,测序质量的重要评价手段。
Sequences flagged as poor quality :标记为差的碱基序列,这个貌似没有神马用处
Sequence length :测序获得长度
%GC :整个测序中碱基GC所占比例,一般有物种特异性。
注意 :Sequence length GC
横坐标 :碱基所在的测序位置,这个一般根据不同测序平台会有不同。
纵坐标 :测序质量。20%代表错误为1%的错误读取率,30%代表0.1%的错误读取率。
图例 :箱线图,25-50-75%代表数据所处位置。
注意 :理论来说好的测序结果应该都在绿色和黄色区域,红色区域代表测序质量较差。
横坐标 :碱基所在的测序位置,这个一般根据不同测序平台会有不同。
纵坐标 :tile的index编号(tile应该是flow cell - lane - tile中的单位。
注意 :也是判定测序质量好和不好的依据,整张图应该以冷色调为主,如果出现大量暖色,则证明某些tail测序有些问题,在后期的处理过程中可以删掉那些暖色的tail。
从read的总体质量判定这次测序的质量,是质量分析的重要标准之一。
横坐标 :测序质量。20%代表错误为1%的错误读取率,30%代表0.1%的错误读取率。
纵坐标 :在该质量值下的read数目。
注意 :峰应该集中在高质量区间,即图像起始点最好大于20。
横坐标 :还是碱基的数目
纵坐标 :碱基的百分比
注意 :ATCG碱基分布应该差异不大。两条紧挨着的线:AT、CG误差应小于10%,若大于该比值则认为测序风险大。由于测序仪调整造成前几个测序结果略有误差,同时也有同学指出可能是含有接头序列所导致的碱基偏好,可以通过后期工作将前几个碱基删掉。在本案例中由于测序质量很好,就没有进行删除操作(ps 查了几个资料,大家争议比较大,建议还是根据自身情况进行确定,至于删除与否建议参考biostar相关的帖子)。
横坐标 :每个read的平均GC比
纵坐标 :该GC比下,read的数量
注意 :蓝色是理论值,测序结果趋向蓝色越好。如果有其它污染会导致双峰图样。
横坐标 :又是碱基数目。
纵坐标 :N(未知碱基)所占比例。
注意 :未知碱基数目越少越好。
[图片上传失败...(image-ee9921-1564222758518)]
横坐标 :read的长度,本文中应该是125。
纵坐标 :代表在该长度下read数目的多少。
横坐标 :序列重复等级
纵坐标 :重复数量
注意 :在测序建库PCR过程中,由一些基因扩增次数过多导致。重复次数为一次的比例越高越好。但由于本文中使用的是转录组数据,偏高正常,一旦移除会对后面定量分析计算带来困扰。
大量重复序列 :FastQC检测出的大量重复序列。
横坐标 :又又又是碱基所在位置。
纵坐标 :接头所占比例。
注意 :当FastQC参数中 -a中没有参数时,默认使用四种lillmina接头进行匹配。
横坐标 :又又又又是相同的内容。
纵坐标 :观测值和预测值的比值。
注意 :啥是K-mer啊?具体可以看参考资料5。其实就是一段短小的重复序列。
这是前面k-mer=5时(软件默认)生成的表格,主要用于检查数量情况。
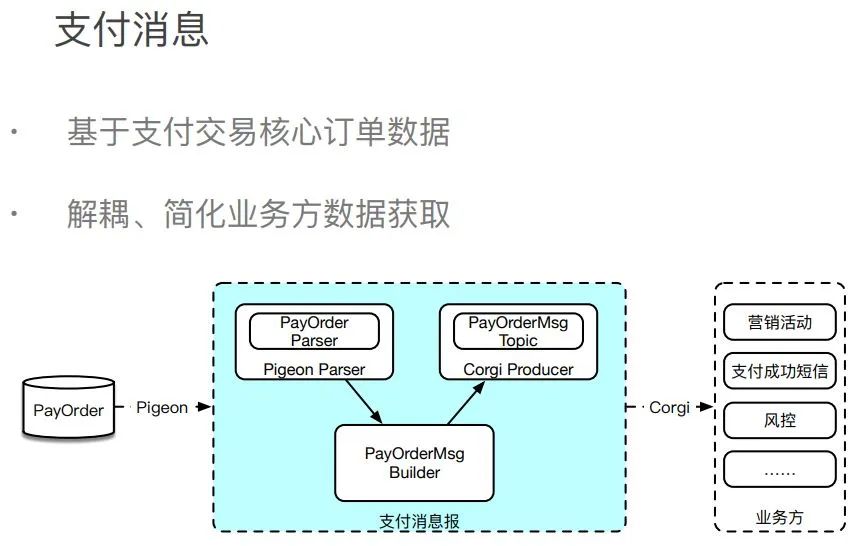
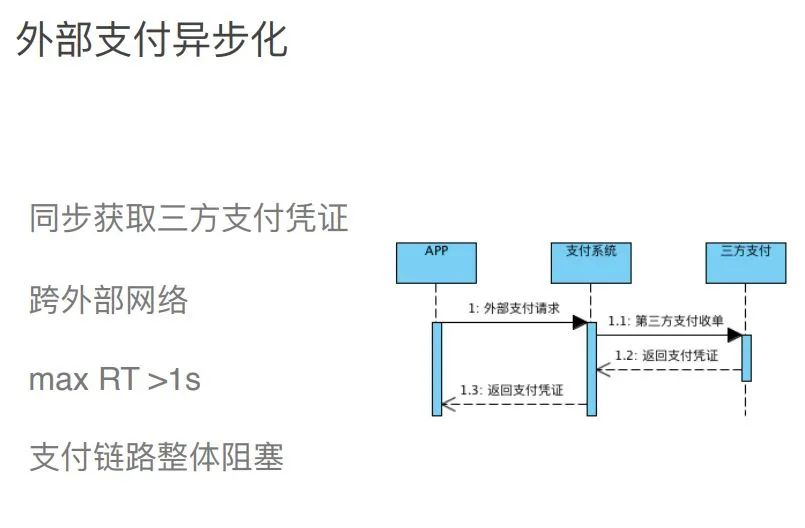
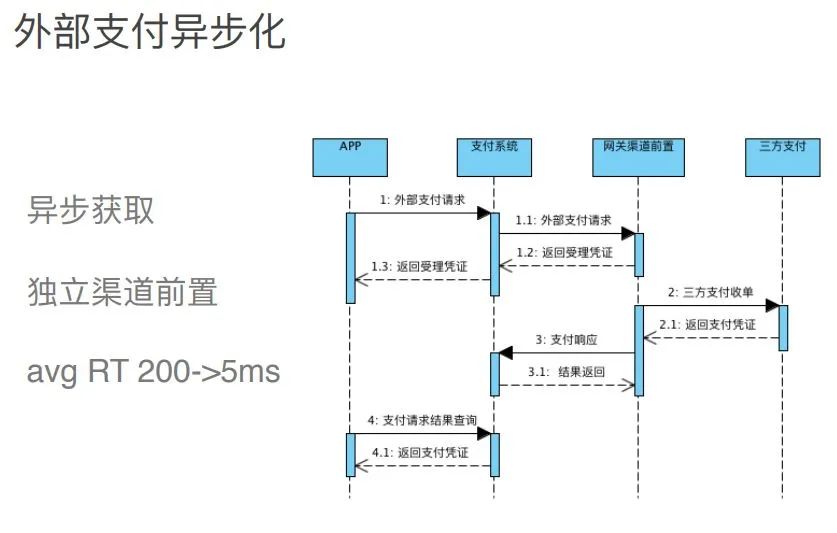
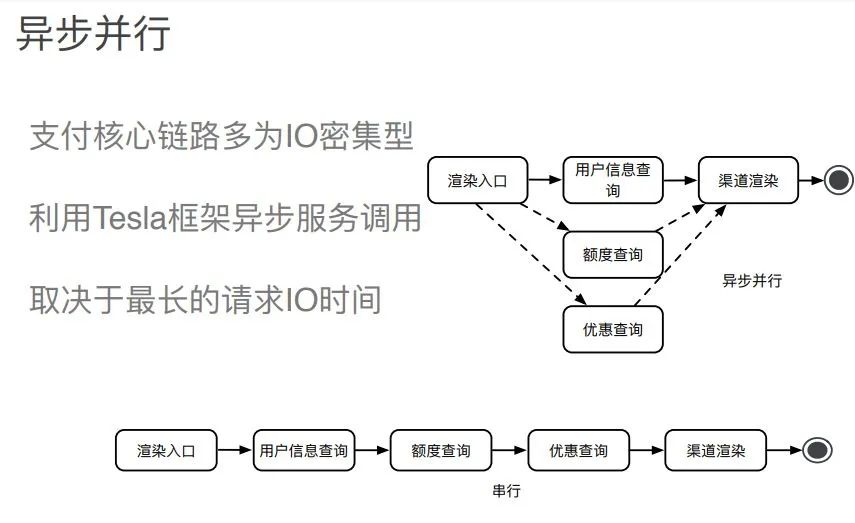
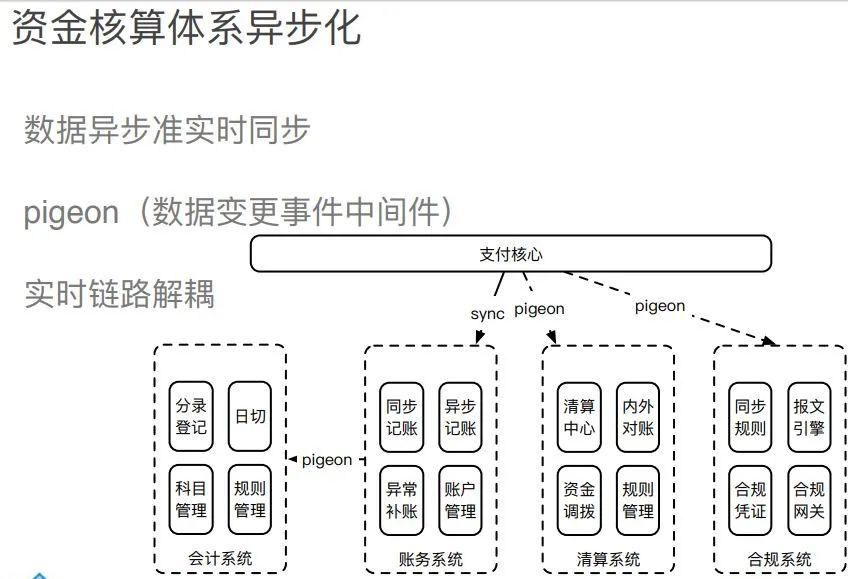
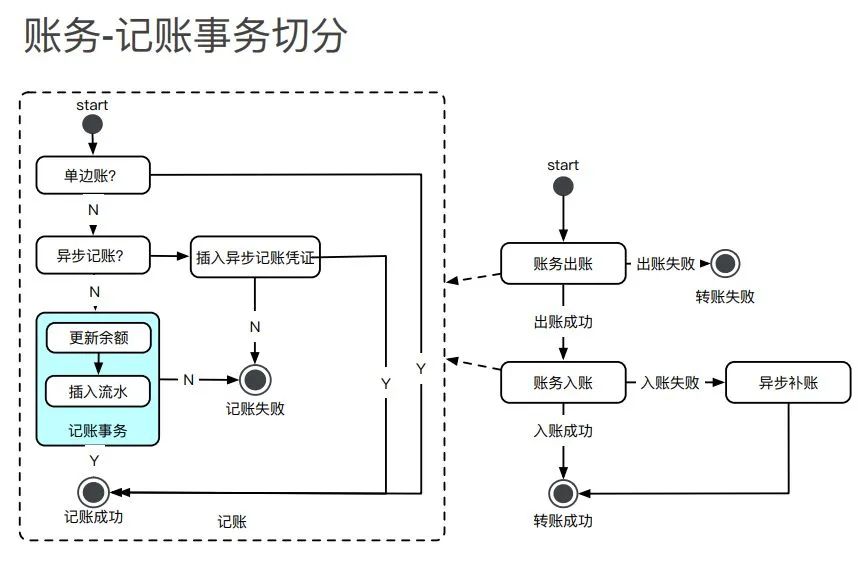
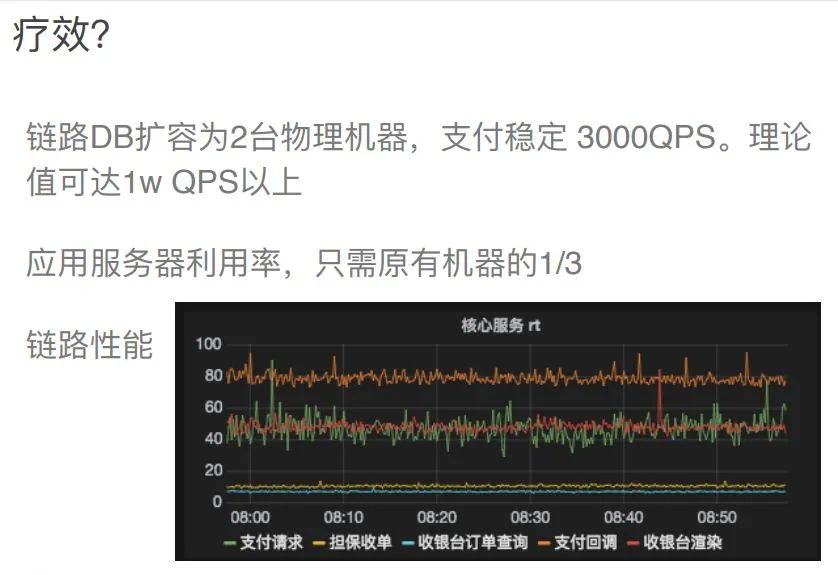
一文读懂电商支付架构设计
图解电商支付架构设计
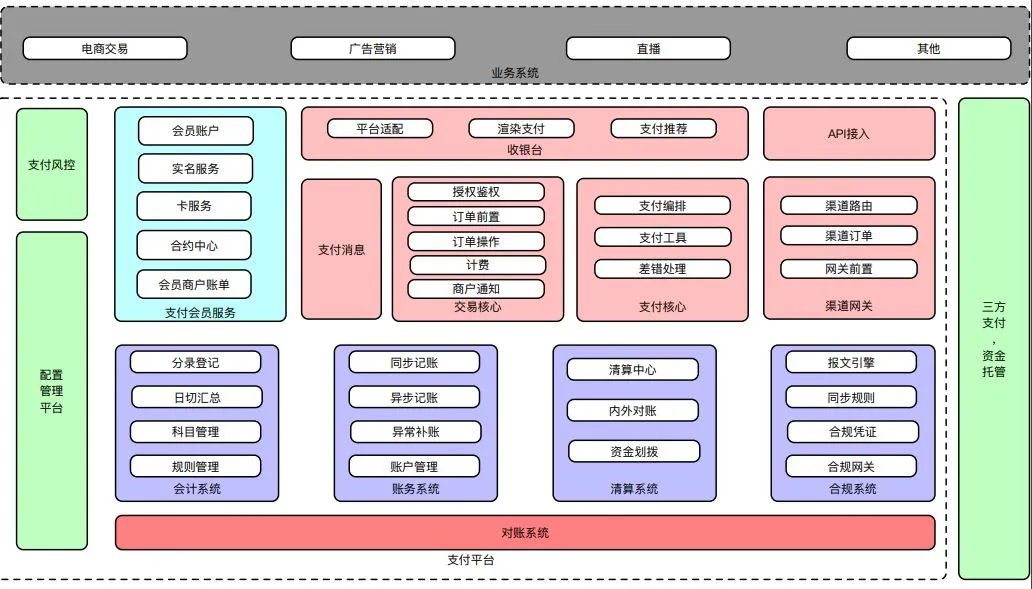
交易核心
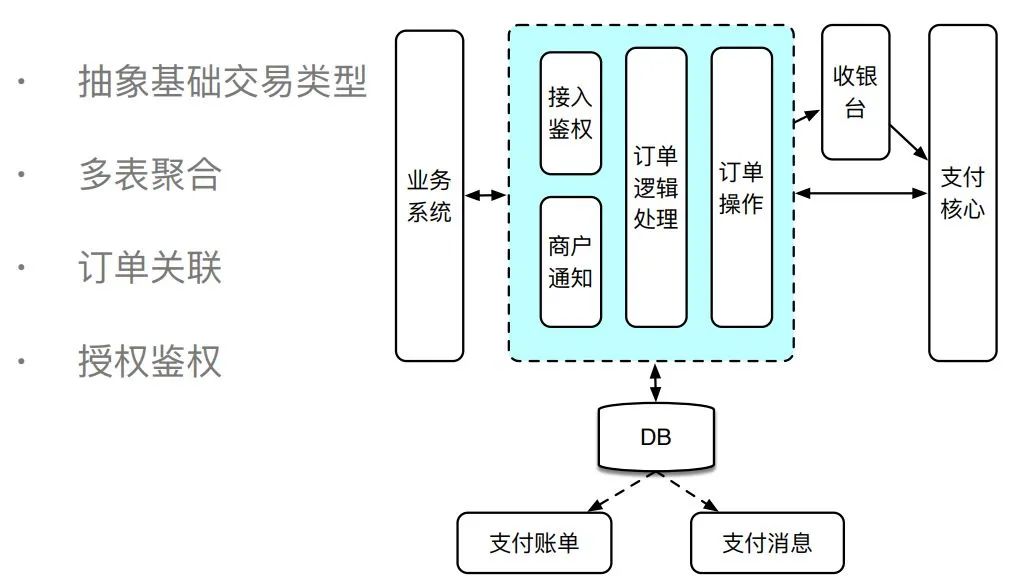
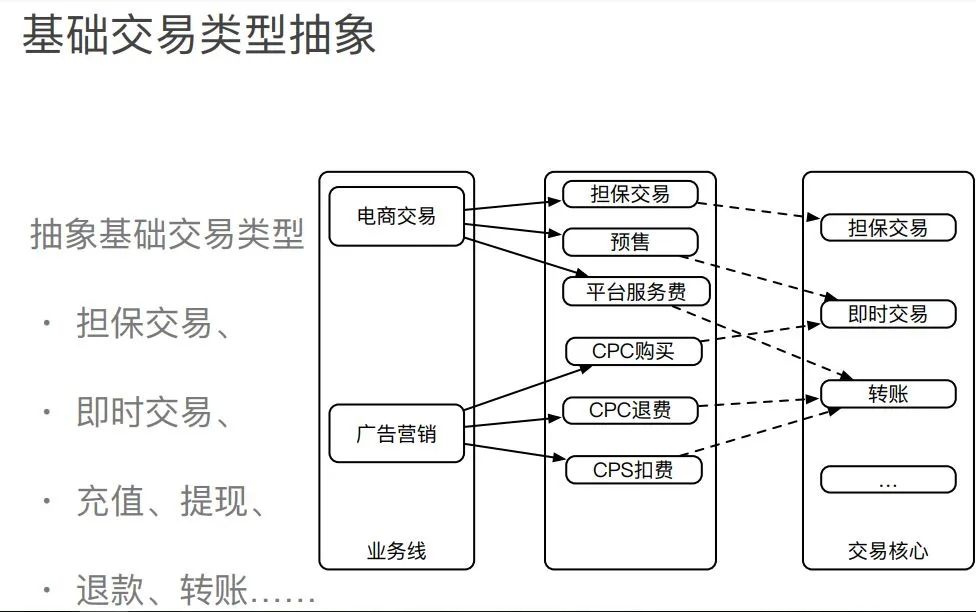
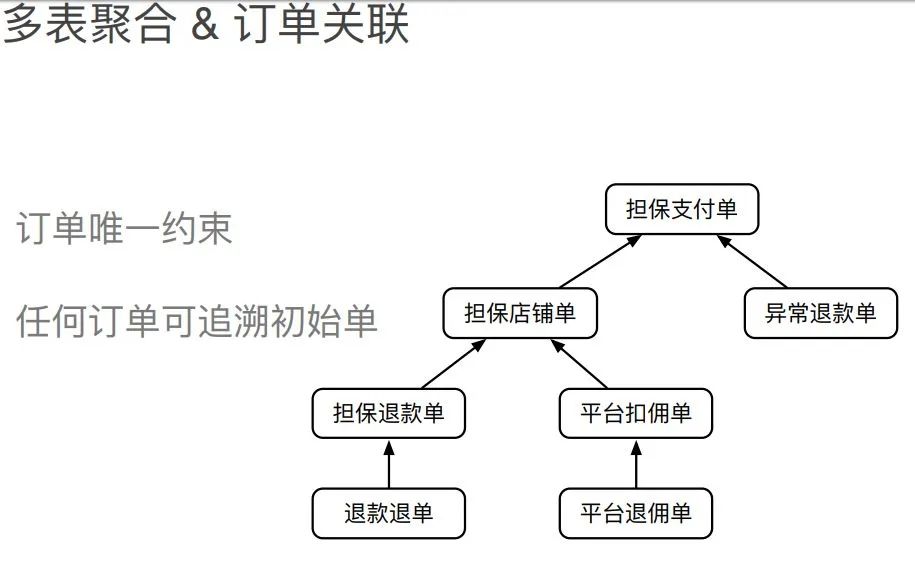
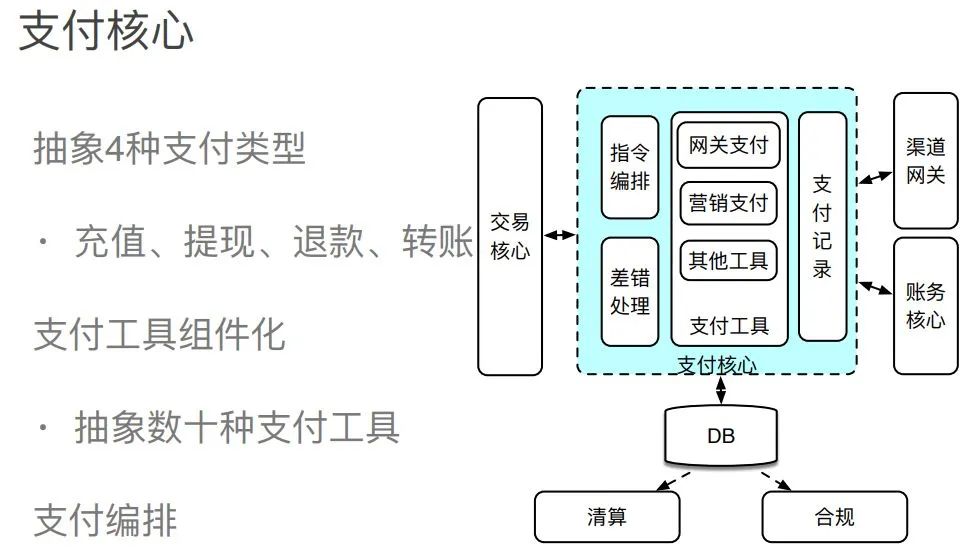
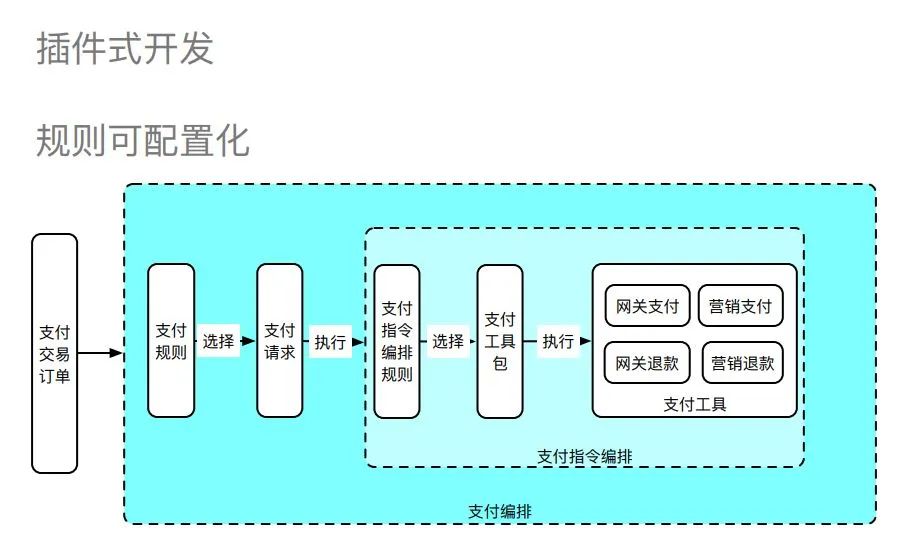
支付编排
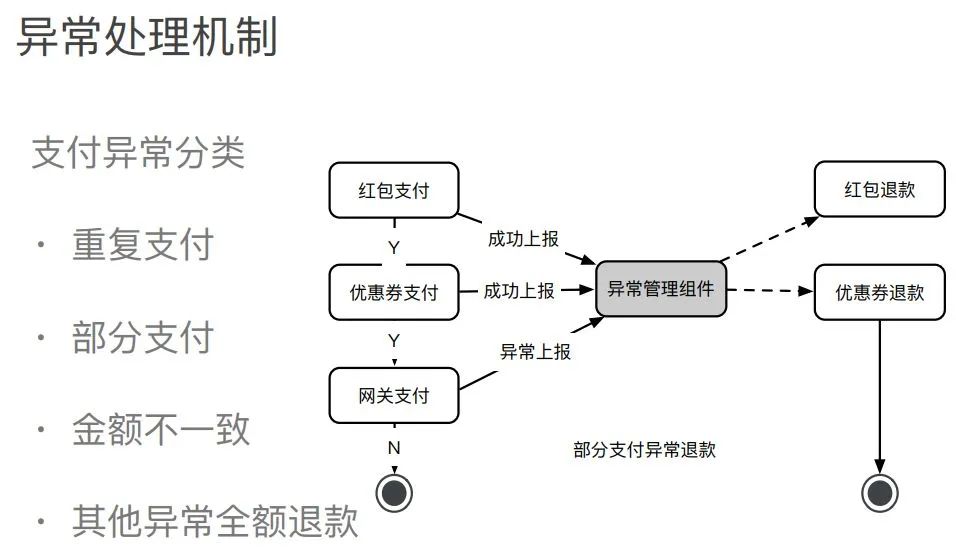
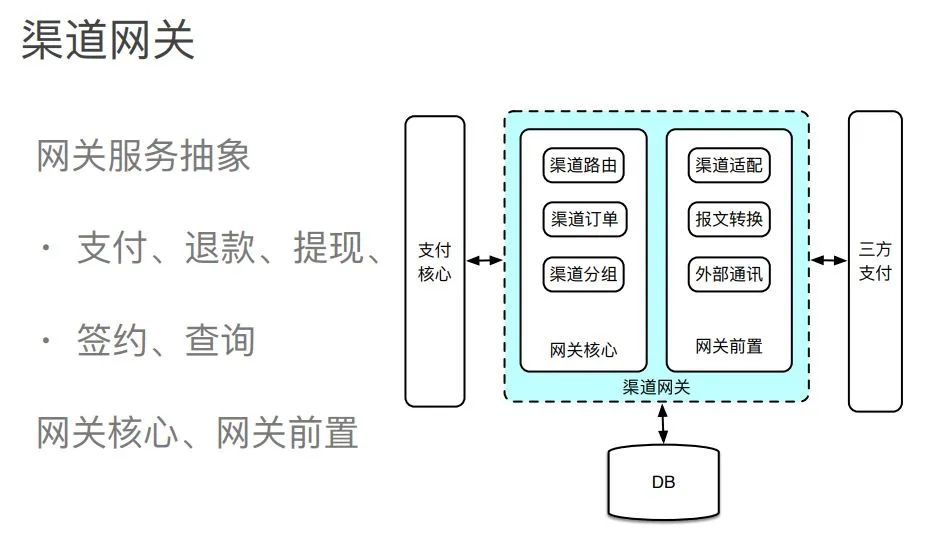








以上是关于一文读懂FastQC Report的主要内容,如果未能解决你的问题,请参考以下文章