变异位点的归一化(normalization of indel)
Posted
tags:
篇首语:本文由小常识网(cha138.com)小编为大家整理,主要介绍了变异位点的归一化(normalization of indel)相关的知识,希望对你有一定的参考价值。
参考技术A 一、定义二、indel的左对齐
三、多等位位点的左对齐
在二代测序中,使用 bcftools call 或者 gatk HaplotypeCaller 工具检出变异位点的时候,会得到原始的vcf文件,这些vcf文件中的indel一般是没有进行归一化的,这里的归一化主要针对两个方面:(1) indel的左对齐和(2) 多等位位点的左对齐。
如果不进行归一化,相同的变异可能会因为形式的不同而导致无法比较,例如下面的两个位点其实是相同的位点,如果不进行处理,对后续的注释和分析会带来很大的麻烦。
如下面所示的vcf文件中检出的indel(这里仅展示前5列内容)
通过左对齐后可以变成
第一个indel可以进行左对齐简化,由 G 变成 GC ,表明在该位置发生了一个C的插入,而第一个indel可以去掉右边冗余的 C ,由 TCC 变为 T ,发生了一个CC的缺失。
该过程我们可以使用 bcftools norm 来实现,关于该命令的详细用法请见 bcftools文档 ,这里重点介绍几个参数:
接下来使用 bcftools norm 来实现上述过程:
命令中的 -Ov 为 bcftools的通用命令 ,我们可以指定输出的文件类型, b 为 compressed bcf(压缩的bcf文件), u 为uncompressed bcf(不压缩的bcf文件), v 为 uncompressed vcf(未压缩的vcf文件), z 为compressed vcf(压缩的vcf文件)。
由于人类是二倍体生物,同源染色体都是以成对的方式出现的,因此会出现多等位变异。请观察如下的几个位点:
上述的几个位点为多等位位点,两条同源染色体在相同的位置分别发生了不同的变异。通过左对齐可以变成如下的样子:
snp的多等位位点不发生改变,而indel的多等位位点的REF和ALT可以同时去掉冗余的两个 CC 碱基。
上述的过程可以使用 bcftools norm 命令来实现
可以看到和普通的indel的归一化并没有什么区别,这里你可能有疑问既然两者的归一化的实现方式相同,为什么要将indel的归一化和多等位位点的归一化分开来分析呢?
让我们来看下面的例子:
上述的位点是一个多等位位点,两条同源染色体在10003这个位置上分别发生了 AC 到 AG 和 AC 到 ACT 的变异,这里可以注意到 AC 到 AG 可以去掉碱基 A ,而 AC 到 ACT 可以通过左对齐来变成 C 到 CT ,接下来让我们将该位点放到vcf文件中并使用命令 bcftools norm -f b37.fasta -m +both test.vcf -Ov -o test.normed.vcf 来进行归一化,结果如下:
你会发现该位置的多等位位点变成了一个snp和一个indel!这显然不是我们想要的,为了解决这个问题,我们可以先对多等位位点进行拆分,在左对齐后,再进行合并,合并的时候,我们可以选择 -m +both 而不让snp和indel合并到一起(如果你使用 -m +any 参数,结果它们将再次合并到一起)。
最后得到如下的结果
巧妙的借助管道符来解决这个问题,这条命令请及时做下笔记。
深度学习之 BN(Batch Normalization)批归一化
从字面意思看来Batch Normalization(简称BN)就是对每一批数据进行归一化,确实如此,对于训练中某一个batch的数据x1,x2,…,xn,注意这个数据是可以输入也可以是网络中间的某一层输出。在BN出现之前,我们的归一化操作一般都在数据输入层,对输入的数据进行求均值以及求方差做归一化,但是BN的出现打破了这一个规定,我们可以在网络中任意一层进行归一化处理,因为我们现在所用的优化方法大多都是min-batch SGD,所以我们的归一化操作就成为Batch Normalization。
在我目前的水平看来,深度学习的本质上就是刻画问题的内部复杂结构特征,进行任意的非线性变换,简单来说可以看成是学习数据的分布(个人看法,勿喷)。机器学习领域有个很重要的假设:IID独立同分布假设【数据的独立同分布(Independent Identically Distributed)】————假设训练数据和测试数据是满足相同分布的,这是通过训练数据获得的模型能够在测试集获得好的效果的一个基本保障。如果训练数据与测试数据的分布不同,那么网络的泛化能力就会大大降低。另一方面,如果每批训练数据的分布各不相同,网络就要在每次迭代时都去学习适应不同的分布,那么网络的训练速度就会大大降低,这就是为什么我们需要对输入数据做一个归一化处理。
那BN的作用到底是什么呢?根据刚才提到的机器学习领域的IID独立同分布假设,我们可以发现BN的作用就是在深度神经网络训练过程中使得每一层神经网络的输入均保持相同分布。
归一化可以将我们的数据转换为均值为0,方差为1的服从标准正态分布。
比如可采用Z-score标准化方法:

证明过程:

下面我们以sigmoid激活函数来说明为何我们需要进行批归一化
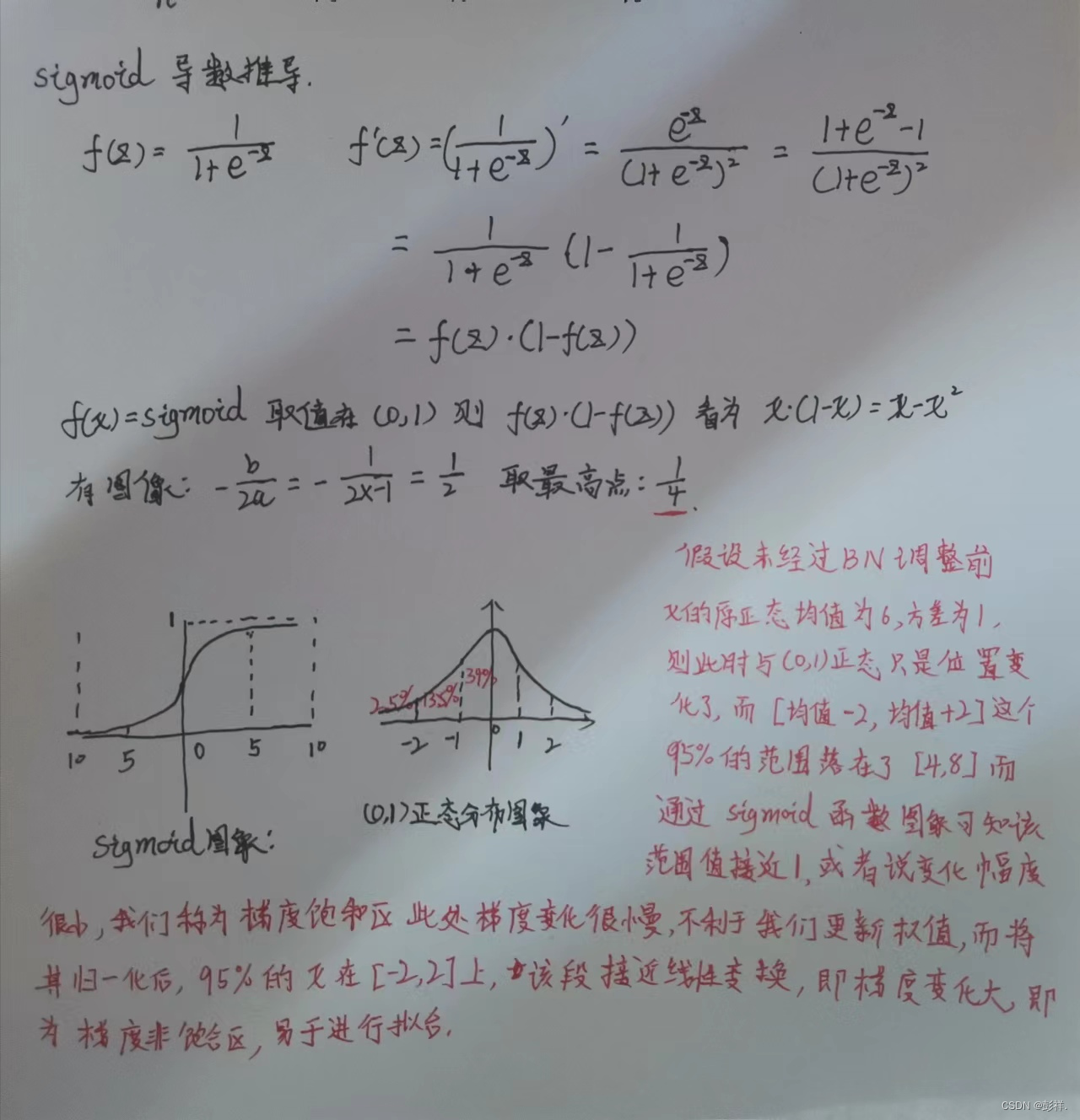
从上面提到的过程中学到了什么呢?BN的作用其实简单来说就是把隐层神经元激活输入从各种奇怪的非【均值为0方差为1的正态分布】拉回到【均值为0方差为1的正态分布】。
但是又出现了一个问题,如果都通过NBN实现归一化的话,不就是把非线性函数替换成线性函数了?这意味着什么呢?这就要说到我们深度学习的学习过程是怎么实现的了,即通过多层非线性实现一个泛化的过程。如果是多层的线性函数其实这个深层就没有任何的意义,因为多层线性网络和一层线性网络是等价的。这意味着网络的表达能力下降了,也就是意味着深度的意义就没有了。所以BN为了保证非线性的获得,对变换后的满足【均值为0方差为1的正态分布】的x又进行了scale+shift操作。每个神经元增加了两个参数scale和shift,这两个参数通过训练学习到的,关键就是在线性和非线性之间找到一个比较好的平衡点,既能享受非线性的较强表达能力的好处,又避免太靠非线性区两头使得网络收敛速度太慢。最不济的情况,这两个参数可以等效成最开始的状态,也就是训练数据集本来的特征,这只是需要简单的计算就可以实现。
这个时候你可能会觉得真是好简单,不就是在网络中间对数据做一个归一化处理?然而其实并不是那么简单的。如果是仅仅使用上面的归一化公式,对网络某一层A的输出数据做归一化,然后送入网络下一层B,这样是会影响到本层网络A所学习到的特征的,这样会导致网络表达能力下降。比如:网络中间某一层A学习到特征数据本身就分布在S型激活函数的两侧,你强制把它给归一化处理了、标准差也限制在了1,把数据变换成分布于s函数的中间部分,这样就相当于搞坏了这一层网络所学习到的特征分布。为了防止这一点,论文提出了一个解决方法,在每个神经元上增加两个调节参数(scale和shift),这两个参数是通过训练来学习到的,用来对变换后的激活反变换,使得网络表达能力增强,即对变换后的激活进行如下的scale和shift操作,这其实是变换的反操作:

BatchNormalization网络层的前向传导过程公式就是:

BN算法在训练时的操作就如我们上面所说,首先提取每次迭代时的每个mini−batch的平均值和方差进行归一化,再通过两个可学习的变量恢复要学习的特征。
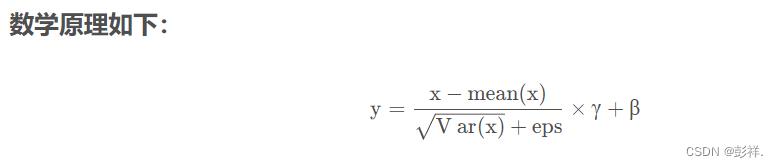

以上是关于变异位点的归一化(normalization of indel)的主要内容,如果未能解决你的问题,请参考以下文章
使用Sklearn的MinMaxScaler做最简单的归一化