泛基因组工具: Roary, panX, PEPPAN, PGAP
Posted
tags:
篇首语:本文由小常识网(cha138.com)小编为大家整理,主要介绍了泛基因组工具: Roary, panX, PEPPAN, PGAP相关的知识,希望对你有一定的参考价值。
参考技术A 引用文章
标题: Insights on the Evolutionary Genomics of the Blautia Genus: Potential New Species and Genetic Content Among Lineages
中文:Blautia菌基因组进化分析
杂志:Front. Microbiol.
时间:2021.4
该文章分析之一:使用三种泛基因组方法同时处理一批菌株基因组
稀释曲线分析
KEGG module注释
泛基因组分析
1 输入文件由prokka生成
2 Roary PEPPAN使用gff文件,panX使用genebank文件
3 PEPPAN.gene_content.curve生成稀释曲线输入数据,pandas matplotlib绘制曲线
4 panX geneCluster.json和pandas生成基因presence absence矩阵
5 R micropan估计泛基因组openness/closedness
三种方法的结果
文章:Roary: rapid large-scale prokaryote pan genome analysis. bioinformatics 2015
引用:1780
GITHUB: http://sanger-pathogens.github.io/Roary/
Tutorial: https://github.com/microgenomics/tutorials/blob/master/pangenome.md
文章:panX: microbial pan-genome analysis and exploration. Nucleic acids research, 2018
引用:131
GITHUB: https://github.com/neherlab/pan-genome-analysis
conda git安装:
文章:Accurate reconstruction of bacterial pan- and core genomes with PEPPAN. Genome Res. 2020
引用:5
GITHUB: https://github.com/zheminzhou/PEPPAN
地址: http://pgaweb.vlcc.cn/
PGAP本地: https://sourceforge.net/projects/pgap/
PGAP: pan-genomes analysis pipeline.Bioinformatics,2012
被引:328
PanGP: a tool for quickly analyzing bacterial pan-genome profile.Bioinformatics,2014
被引:95
PGAP-X: Extension on pan-genome analysis pipeline.BMC Genomics,2018
被引:18
PGAweb: A Web Server for Bacterial Pan-Genome Analysis.Front Microbiol,2018
被引:15
PGAweb在线使用 - 测试数据
1 使用example gbk数据:输入文件格式
2 参数:identity 0.8,其他默认
3 运行等待结果
PGAP本地使用
1 软件获取
sourceforge: https://sourceforge.net/projects/pgap/
Pangenome
生信和数据挖掘必备:一款超好用的基因组分析工具网站!
编辑:小怡
本文为小狗科研平台原创,转载请在【后台】留言
目前,很多科研工作者热衷于做肿瘤研究,利用生物信息学和数据挖掘方法,如GEO,TCGA等,做到不用做实验,也能发SCI文章。
本文介绍一款适用于肿瘤基因组分析的在线网站:GSCALite
(链接:http://bioinfo.life. hust.edu.cn/web/GSCALite/)
GSCALite是一个肿瘤基因组分析平台,该平台整合了来自TCGA库的33种肿瘤类型的基因组数据,GDSC,CTRP的药物反应数据,以及来自GTEX的正常组织数据,用于统一的数据分析流程中的基因组分析。待分析完成后,数据和结果的可以从官网直接下载。
该工具网站是华中科技大学生命科学学院的郭安源团队开发的,用于基因组癌症分析,相关介绍发表在《Bioinformatics》杂志上。
图片来源于《Bioinformatics》官网
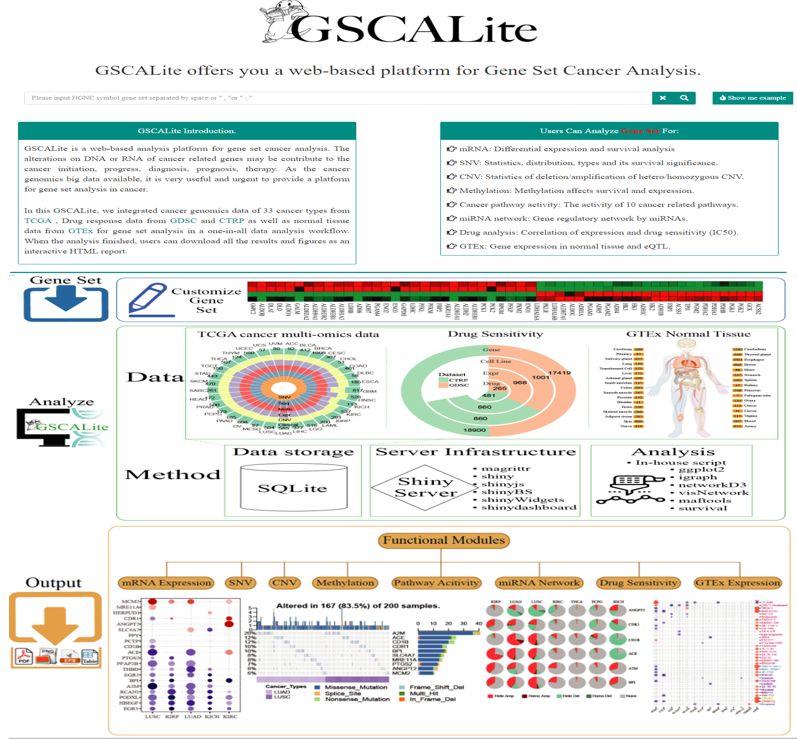
图片来源于网站
2)SNV:统计学,分布,类型及其生存意义
3)CNV:杂合/纯合 CNV缺失/扩增的统计资料
4)甲基化:甲基化影响生存和表达
5)肿瘤通路活性:10种癌症通路的活性
6)miRNA网络:通过miRNA的基因调控网络
7)药物分析:表达与药物敏感性的相关(IC50)
8)GTEx: 基因在正常组织和eQTL的表达。
本文以RAS家族基因为例,来分享一下该网站的功能。
在框中输入想要研究的基因,注意至少输入5个基因,否则无法输出结果。点击TCGA库中的肿瘤或组织类型,以及右侧分析类型。在下方会显示所选的基因总个数,有效和无效个数,并可以导出数据。
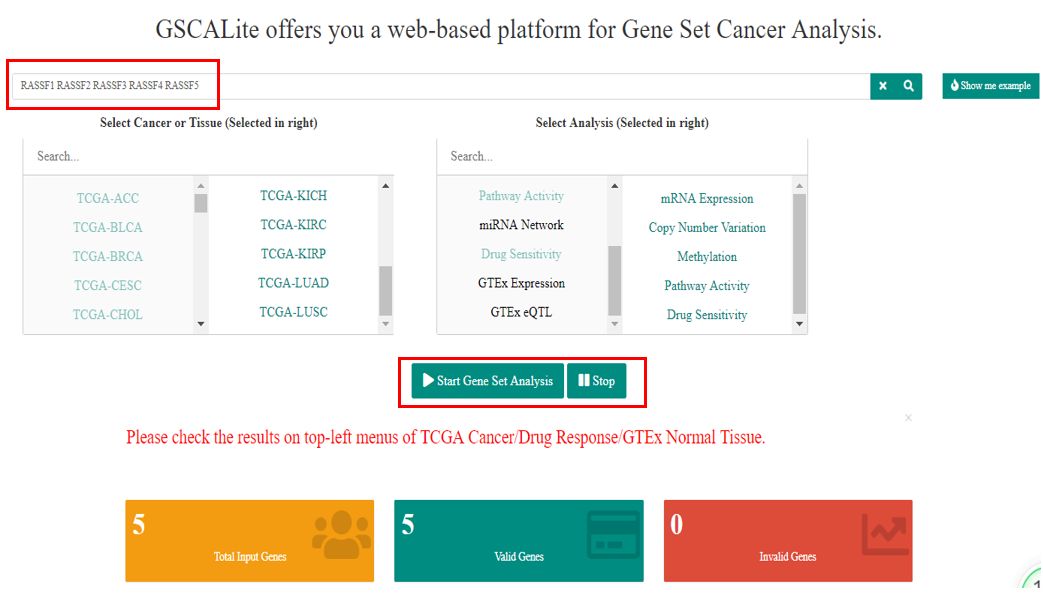
图片来源与官网
接下来,点击“Start Gene Set Analysis”按钮,查看分析结果。
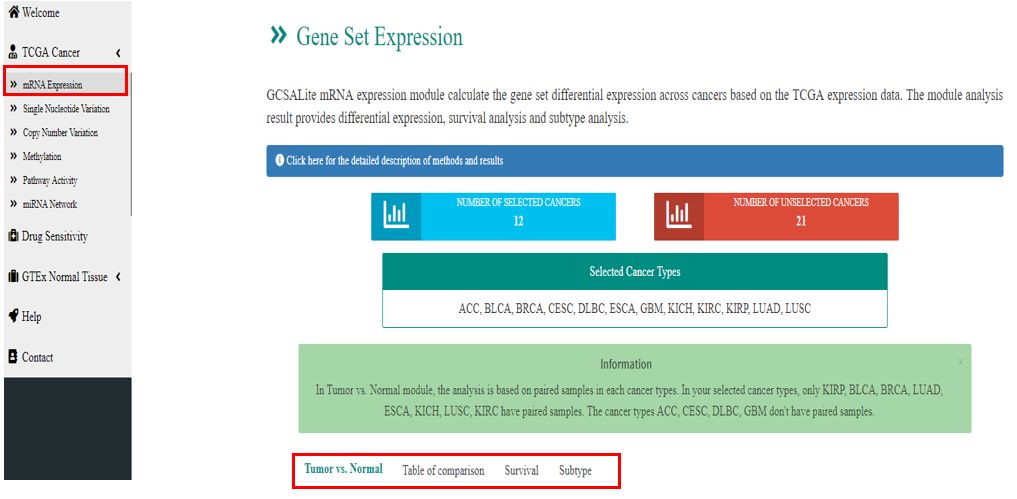
TCGA Cancer
miRNA表达
该模块分析结果提供基因差异表达,生存分析和亚型分析,相应的图形结果提供PDF,png和EPS多种文件格式下载。
单核苷酸变异(SNV)分析
SNV模块能显示特定肿瘤类型中的SNV频率和基因集的变异类型。突变对总体生存OS的作用通过对数秩检验来评估。
①SNV percentage profile: 该部分展现特定肿瘤类型中所选基因的相对SNV频率,每一行表示一个基因。
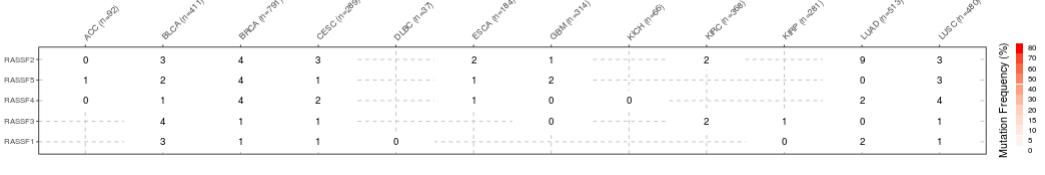
图片结果来源于官网
②SNV summary:显示变体类型(SNP或DEL),变体分类和SNV类等。
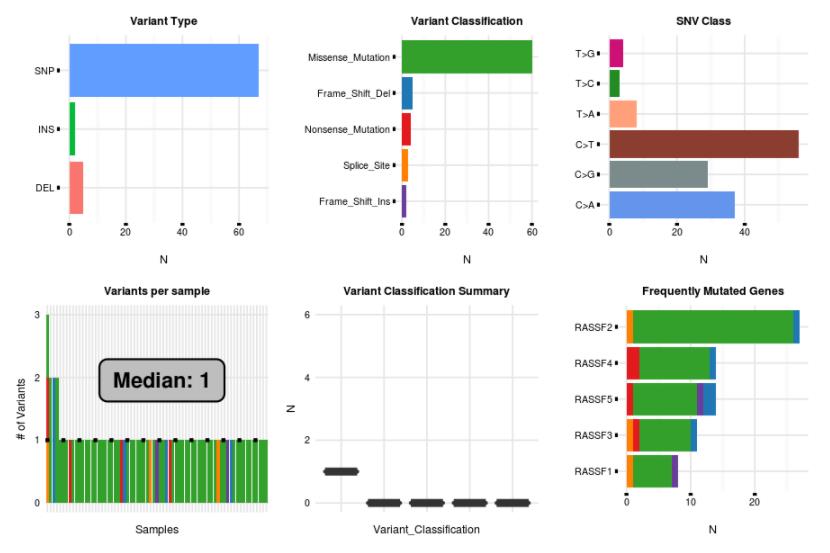
图片结果来源于官网
③SNV>显示在选定的TCGA肿瘤类型中详细的SNV信息的基因集。
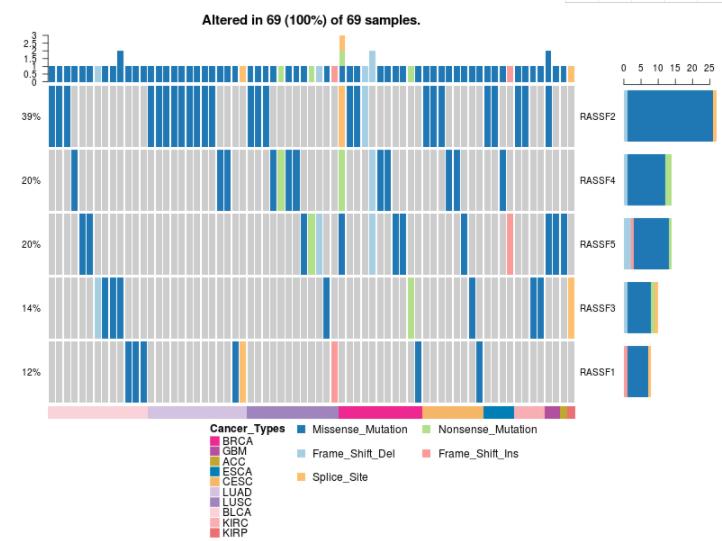
图片结果来源于官网
④SNV survival plot:该部分显示选定的肿瘤类型中有或无SNV的样本的Kaplan-Meier生存评估。
CNV分析
该部分结果图形包括CNV Pie distribution(CNV)类别(杂合/纯合,删除/扩增)。Heat CNV部分表示CNV分类在选定癌症类型中的分布。CNV to expression 则显示CNV于基因表达的关系。
甲基化分析
该模块主要探讨所选肿瘤与配对正常人之间的甲基化差异。包括Differential Methylation, Methylation survival和Methylation to Expression三部分。本研究图形如下:
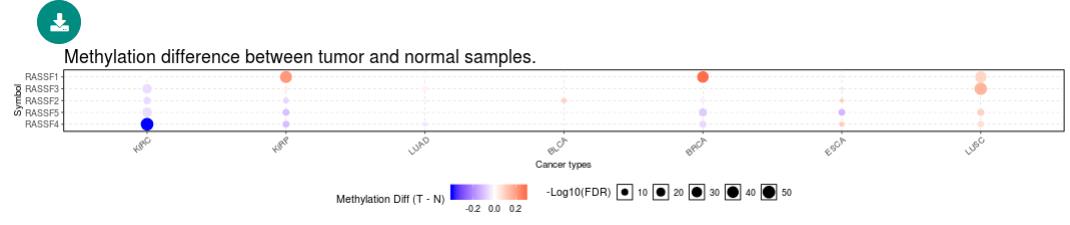
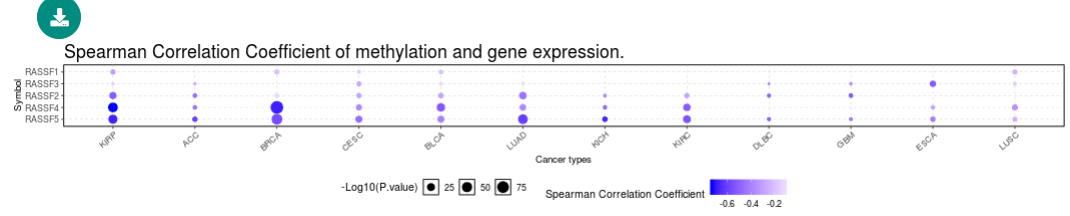
图片截图来源于官网
通路活性
该模块包括Global percentage, Heatmap percentage和Relation network三个部分,主要提示选定肿瘤类型的基因整体活性,基因对通路的影响百分比(激活/抑制)和基因的表达。
miRNA网络
提供一个miRNA调节网络,可视化miRNA对基因的潜在调节,见图。
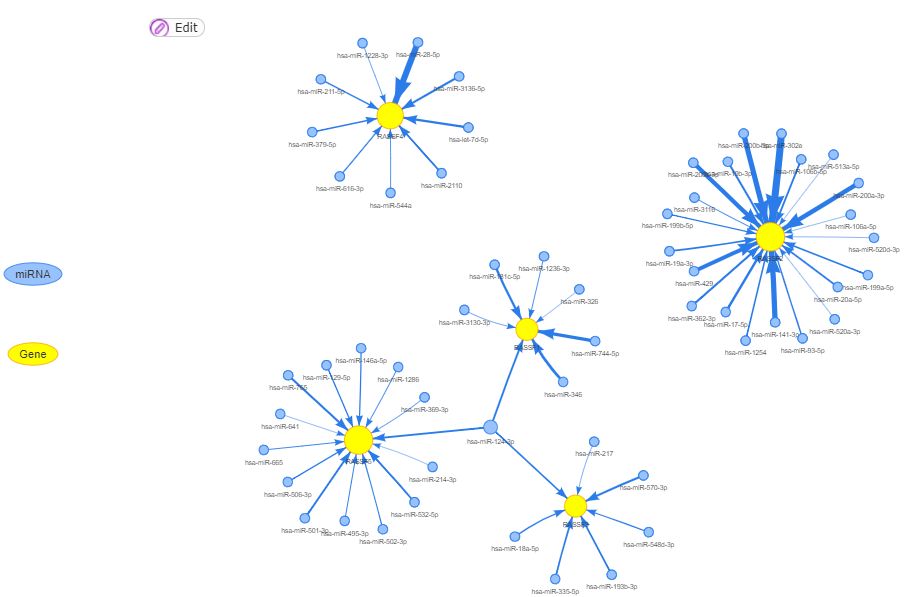
图片来源于官网
药物敏感性分析
该模块整合了GDSC和CTRP中癌细胞系的药物敏感性和基因表达谱数据进行研究。通过Spearman相关分析以药物的敏感性进行基因组中各个基因的表达。
GETX Normal Tissues
该模块包括GETX expression, 以热图和箱式图显示所选GETX正常组织中查询基因集的表达谱和分析得分。同时Table of eQTL in GTEX dataset会以表格形式展示。
对很多临床医生而言,数据都是现有的。所以不需要花时间做试验做科研,只要潜心做临床,在门诊、住院、随访中发现问题,通过挖掘数据、分析数据,你也能发表高分的 SCI 论文。
但在这条道路上,半路出家的大有人在,非常多同学反馈,想要系统的进行学习。为了给大家降低学习门槛,帮助更多医学人员掌握生物医学大数据,小狗阅读团队联合首都医科大学生物医学工程学院副教授华琳,传授大家如何利用大数据发表自己的第一篇生信SCI!
课程原价399元的
《生物医学大数据挖掘及生物信息学案例分析》
限时拼团仅需79元(1月7日24:00截止)
14节精品视频课程,好评率99%!
让你灵活运用大数据,快速发表SCI

以上是关于泛基因组工具: Roary, panX, PEPPAN, PGAP的主要内容,如果未能解决你的问题,请参考以下文章