CTCF ChIP-seq
Posted
tags:
篇首语:本文由小常识网(cha138.com)小编为大家整理,主要介绍了CTCF ChIP-seq相关的知识,希望对你有一定的参考价值。
CTCF ChIP-seq
CTCF(CCCTC binding factor),是CTCF基因编码的转录因子 ,与绝缘子的活性相关。
CTCF蛋白在印记调控区域(imprinting control region,ICR)和分化甲基化区域1(differentially-methylated region-1,DMR1)和MAR3结合抑制胰岛素样生长因子2(Igf2)基因的过程中起重要作用 。CTCF与靶顺序因子的结合可阻断增强子和启动子的相互作用。从而将增强子的活性限制在一定的功能区域。除了阻断增强子外,CTCF还可作为染色质屏障阻止异染色质的传播。人类基因组有将近一万五千个CTCF绝缘体位点,说明CTCF在基因调控方面的功能广泛。而且CTCF结合位点还可作为核小体定位锚。
CTCF是一种重要的转录抑制因子,具有多个可与DNA结合的锌指结构,通过多种机制调控基因表达。CTCF最具特色的功能是通过同源二聚引起DNA成环,参与基因组拓扑结构域(TAD)边界形成,改变染色质空间结构。
作为重要的绝缘子(insulator)元件,CTCF是近年染色质结构领域热门研究对象之一,其核酸结合特征、蛋白互作关系、功能调控机制等都广受关注
CTCF是控制染色质高级结构和表观遗传的关键蛋白,具有组织染色质高级结构、调控基因转录、作为绝缘子蛋白、参与遗传印记和参与选择性剪接等多种功能。CTCF突变会引起人类小头畸形症和智力低下;条件敲除CTCF,能够引发小鼠神经元缺失并阻断淋巴细胞的增殖和分化,增加肿瘤发生的机会;CTCF还可以帮助细胞抵抗凋亡和UV带来的伤害。针对这些现象,现有研究多从CTCF的绝缘子功能和基因表达调控层面进行解释。
CTCF (CCCTC-binding factor)是一种重要的染色质架构蛋白,其与绝缘子的方向性结合在哺乳动物基因组三维空间结构形成和维持中起着至关重要的作用。正向-反向相对方向的CTCF结合位点(简称CTCF位点)可以在染色质黏连蛋白(cohesin)的协助下,形成染色质环,介导远距离DNA元件之间的相互作用;而在染色质拓扑结构域边界区域的CTCF位点呈现反向-正向相背方向分布,发挥绝缘子的功能。
11锌指蛋白CTCF是个高度保守的转录因子并且广泛结合基因组中不同位置。CTCF在基因组中行使相当多样的功能,既可以作为激活因子也可以作为绝缘子并且可以调控染色质的三维结构。
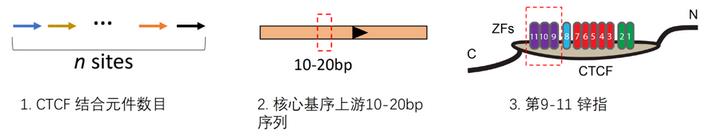
研究团队利用这个系统比较了不同的CTCF结合序列。单个CTCF结合序列即便是存在于拓扑结构域的边界上通常也只有微弱的绝缘子效应。将多个CTCF结合元件顺式安排在一起可以提供更强的绝缘功能,表明CTCF绝缘子在阻隔增强子功能上具有协同效应。
另外作者比较了位于拓扑结构域边界上的和边界外的CTCF结合元件,发现CTCF的核心基序附近的序列而非核心基序本身决定了CTCF结合元件的绝缘子效应。进一步分析发现,CTCF的第9-11个锌指可能结合核心基序上游10bp左右的短序列从而行使绝缘子功能。
REF
https://www.cn-healthcare.com/articlewm/20210519/content-1221946.html
https://www.lascn.net/Item/66934.aspx
https://baike.baidu.com/item/CTCF/10719734?fr=aladdin
R语言学习 - 线图一步法
profile = "Pos;H3K27ac;CTCF;Enhancer;H3K4me3;polII
-5000;8.7;10.7;11.7;10;8.3
-4000;8.4;10.8;11.8;9.8;7.8
-3000;8.3;10.5;12.2;9.4;7
-2000;7.2;10.9;12.7;8.4;4.8
-1000;3.6;8.5;12.8;4.8;1.3
0;3.6;8.5;13.4;5.2;1.5
1000;7.1;10.9;12.4;8.1;4.9
2000;8.2;10.7;12.4;9.5;7.7
3000;8.4;10.4;12;9.8;7.9
4000;8.5;10.6;11.7;9.7;8.2
5000;8.5;10.6;11.7;10;8.2"
profile_text <- read.table(text=profile, header=T, row.names=1, quote="",sep=";")
# tab键分割,每列不加引号
write.table(profile_text, file="line_data.xls", sep="\t", row.names=T, col.names=T,quote=F)
# 如果看着第一行少了ID列不爽,可以填补下
system("sed -i ‘1 s/^/ID\t/‘ line_data.xls")
profile = "Pos;variable;value;set
-5000;H3K27ac;8.71298;A
-4000;H3K27ac;8.43246;A
-3000;H3K27ac;8.25497;A
-2000;H3K27ac;7.16265;A
-1000;H3K27ac;3.55341;A
0;H3K27ac;3.5503;A
1000;H3K27ac;7.07502;A
2000;H3K27ac;8.24328;A
3000;H3K27ac;8.43869;A
4000;H3K27ac;8.48877;A
-5000;CTCF;10.6913;A
-4000;CTCF;10.7668;A
-3000;CTCF;10.5441;A
-2000;CTCF;10.8635;A
-1000;CTCF;8.45751;A
0;CTCF;8.50316;A
1000;CTCF;10.9143;A
2000;CTCF;10.7022;A
3000;CTCF;10.4101;A
4000;CTCF;10.5757;A
-5000;H3K27ac;8.71298;B
-4000;H3K27ac;8.43246;B
-3000;H3K27ac;8.25497;B
-2000;H3K27ac;7.16265;B
-1000;H3K27ac;3.55341;B
0;H3K27ac;3.5503;B
1000;H3K27ac;7.07502;B
2000;H3K27ac;8.24328;B
3000;H3K27ac;8.43869;B
4000;H3K27ac;8.48877;B
-5000;CTCF;10.6913;B
-4000;CTCF;10.7668;B
-3000;CTCF;10.5441;B
-2000;CTCF;10.8635;B
-1000;CTCF;8.45751;B
0;CTCF;8.50316;B
1000;CTCF;10.9143;B
2000;CTCF;10.7022;B
3000;CTCF;10.4101;B
4000;CTCF;10.5757;B"
profile_text <- read.table(text=profile, header=T, quote="",sep=";")
# tab键分割,每列不加引号
write.table(profile_text, file="line_data_melt.xls", sep="\t", row.names=T, col.names=T,quote=F)
# 如果看着第一行少了ID列不爽,可以填补下
system("sed -i ‘1 s/^/ID\t/‘ line_data_melt.xls")
使用正常矩阵默认参数绘制个线图
# -f: 指定输入的矩阵文件,第一列为行名字,第一行为header。列数不限,列名字不限;行数不限,行名字默认为文本 # -A FALSE: 指定行名为数字 sp_lines.sh -f line_data.xls -A FALSE
# -l: 设定图例的顺序 # -o TRUE: 局部拟合获得平滑曲线 # -A FALSE: 指定行名为数字 # -P: 设置legend位置,相对于原点的坐标 # -x, -y指定横纵轴标记 sp_lines.sh -f line_data.xls -l "‘CTCF‘,‘Enhancer‘,‘polII‘,‘H3K4me3‘,‘H3K27ac‘" -P ‘c(0.8,0.3)‘ -o TRUE -A FALSE -x ‘Up and down 5 kb of TSS‘ -y ‘Relative density‘
# -A FALSE: 指定行名为数字 # -V ‘c(-1000, 500)‘: 设置垂线的位置 # -D: 设置垂线的文本标记,参数为引号引起来的vector,注意引号的嵌套 # -I: 设置横轴的标记的位置 # -b: 设置横轴标记的文字 sp_lines.sh -f line_data.xls -A FALSE -V ‘c(-1000,500)‘ -D "c(‘+1 kb‘,‘-0.5 kb‘)" -I "c(-5000,0,5000)" -b "c(‘-5 kb‘, ‘TSS‘, ‘+5 kb‘)"
# -f: 指定输入文件 # -m TRUE: 指定输入的矩阵为melted format, 三列,第一列为Pos (给-a) # 第二列为variable (给-H,-H默认即为variable) # 第三列为value,名字不可修改 # -A FALSE: 指定行名为数字 # -P ‘c(0.8,0.2)‘: 设置legend位置,相对于原点的坐标 sp_lines.sh -f line_data_melt.xls -a Pos -m TRUE -A FALSE -P ‘c(0.8,0.2)‘
# -C: 自定义线的颜色 sp_lines.sh -f line_data_melt.xls -a Pos -m TRUE -A FALSE -P ‘c(0.8,0.2)‘ -o TRUE -V ‘c(-1000,500)‘ -D "c(‘+1 kb‘,‘-0.5 kb‘)" -I "c(-5000,0,4000)" -b "c(‘-5 kb‘, ‘TSS‘, ‘+4 kb‘)" -x ‘Up 5 kb and down 4 kb of TSS‘ -y ‘Relative density‘ -C "‘pink‘, ‘blue‘"
- 外层引号与内层引号不能相同
- 凡参数值中包括了空格,括号,逗号等都用引号括起来作为一个整体
以上是关于CTCF ChIP-seq的主要内容,如果未能解决你的问题,请参考以下文章