如何获得基因组CpG位点信息 —— Package:BSgenome.Hsapiens.UCSC.hg19
Posted
tags:
篇首语:本文由小常识网(cha138.com)小编为大家整理,主要介绍了如何获得基因组CpG位点信息 —— Package:BSgenome.Hsapiens.UCSC.hg19相关的知识,希望对你有一定的参考价值。
参考技术A 运行结果如下:如此根据染色体序号循环即可得到保存整个基因组的CpG位点的RData。
BSgenome.Hsapiens.UCSC.hg19 包是基于IRanges,GenomeInfoDb,GenomicRanges, Biostrings,XVector这些包所所构建的。
Biostrings 是 BSgenome.Hsapiens.UCSC.hg19 的一个基础包,其中的 matchPattern() 函数用于根据设定的 pattern 寻找目标 string 的起始和结束位点。结合 BSgenome.Hsapiens.UCSC.hg19 包的基因组数据可用于建立一些数据集,如CpG位点data;或保存其他的定序列的位点信息。
eQTL
首先QTL是数量性状位点,比如身高是一个数量性状,其对应的控制基因的位点就是一个数量性状位点,而eQTL就是控制数量性状表达位点,即能控制数量性状基因(如身高基因)表达水平高低的那些基因的位点。
数量性状基因座:控制数量性状的基因在基因组中的位置称数量性状基因座。常利用DNA分子标记技术对这些区域进行定位,与连续变化的数量性状表型有密切关系
表达数量性状基因座(expression Quantitative Trait Loci,eQTL)是对上述概念的进一步深化,它指的是染色体上一些能特定调控mRNA和蛋白质表达水平的区域,其mRNA/蛋白质的表达水平量与数量性状成比例关系。eQTL可分为顺式作用eQTL和反式作用eQTL,顺式作用eQTL就是某个基因的eQTL定位到该基因所在的基因组区域,表明可能是该基因本身的差别引起的mRNA水平变化;反式作用eQTL是指某个基因的eQTL定位到其他基因组区域,表明其他基因的差别控制该基因mRNA水平的差异。
eQTL就是把基因表达作为一种性状,研究遗传突变与基因表达的相关性: 就好像研究遗传突变与身高的相关性一样
早年可以通过同时做一个个体的SNP芯片和cDNA芯片, 在全基因组尺度研究突变与表达的相关性, 这种研究需要较多个体(例如1000个); 现在随着深度测序的出现,很多人开始用RNA-Seq在较少量个体中研究allele-specific expression,本质上就是eQTL.
简单地说, 遗传学研究经常发现一些致病或易感突变, 这些突变怎样导致表型有时候不太直观; 所以用某个基因的差异表达作为过渡: 突变A-->B基因表达变化-->表型;
From https://www.cnblogs.com/Acceptyly/p/3904108.html
=====================================================
从基因的改变到疾病等现象的出现,中间缺失了重要的一环,那就是基因的表达。也许在测序中,我们可以看到某一个基因上某一个位置的变化(比如说SNP单核苷酸变化),但是这种变化并不一定会影响mRNA的产生或者蛋白的改变。也就有可能不会影响到疾病或其他生物学过程。于是科学家想到了另一个指标——mRNA的序列数据。因为只有被表转译到mRNA上的基因,才可能进一步表达为蛋白(图1)。
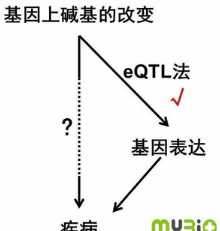
图1:eQTL是沟通基因改变与疾病的桥梁
但是要怎么搞清DNA改变是怎么影响mRNA的出现呢?这一过程被称为Expression quantitative trait loci(eQTL) 分析,目的在于得到单个DNA突变与单个基因表达量之间的相关性。与单个基因mRNA表达量相关的DNA突变,就被称为eQTL。
简单来讲,我们首先通过全基因组测序获得每个个体的DNA全序,然后以同种族的其他个体作为参照,标记出该个体所有的DNA变异位点, 称为SNP位点。同时,我们通过全基因组mRNA表达量测序得到该个体的特定组织样本中的基因表达量。以全部DNA变异位点为自变量,轮流以每种mRNA表达量为因变量,用大量的个体数据做样本进行线性回归,就可以得到每一个SNP位点和每一个mRNA表达量之间的关系。
GTEx是第一个收集了多个人体器官mRNA测序的数据库,并提供了跨器官的eQTL研究平台。
当前使用的GTEx v6p版本的原始数据来自于449名生前健康的遗体捐献者的44个不同的器官。图2是不同器官里面样本数的直观展示。由这个图可以看出,这一数据库中涉及的数据覆盖面非常广,数据量大,具有重要的应用潜力。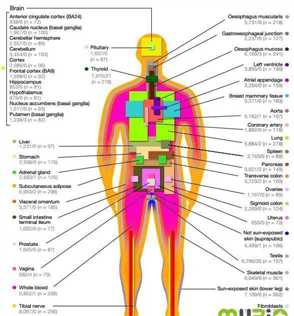
图2:GTEx 样品取材来源图示。灰色字体为 cis-eQTL 数/trans-eQTL数
链接:https://www.jianshu.com/p/2e1e9d3ccd63
=====================================================
常见问题
eQTL分析可以研究哪些疾病?
近年来许多科学家运用eQTL联合GWAS分析的方法,寻找致病风险位点,这些联合分析的方法已应用在类风湿性 关节炎、肾病、精神分裂等疾病的研究中。
eQTL对非编码区功能怎样进行研究?
通过eQTL分析,可以解释非编码区基因突变与基因表达之间的调控关系。系统的了解基因转录的调控机制,构建 基因表达调控网络。
eQTL分析怎样进行药物处理前后反应的研究?
对药物处理前后的机体/细胞进行eQTL的差异分析,可以很好的研究药物处理前后反应的差异,为寻找药物作用靶 点,以及药物作用机制提供基因层面的证据。
怎样利用eQTL分析进行人群队列研究?
在进行人群队列研究中,可以进行eQTL分析,通过比较不同人群中eQTL的差异,充实人群队列研究结果,寻找不同 人群中基因差异与基因表达调控的关系。
from http://www.novogene.com/tech/service/eQTL/QA/
=====================================================
=====================================================
=====================================================
以上是关于如何获得基因组CpG位点信息 —— Package:BSgenome.Hsapiens.UCSC.hg19的主要内容,如果未能解决你的问题,请参考以下文章
简单易懂的在线网站datamonkey:支系基因位点的选择压力检测
eQTL | Expression quantitative trait loci | 表达数量性状基因座 | QTL | 数量性状位点