利用PCA进行数据降维
Posted Babyface Killer
tags:
篇首语:本文由小常识网(cha138.com)小编为大家整理,主要介绍了利用PCA进行数据降维相关的知识,希望对你有一定的参考价值。
PCA原理
在介绍PCA之前首先要熟悉一下数学推导过程。
特征多项式:
设A为一个方阵,则该方阵的特征多项式就为该方阵减去 倍的单位矩阵后构成的矩阵的行列式。而该多项式的所有解即为
倍的单位矩阵后构成的矩阵的行列式。而该多项式的所有解即为 的值,也就是该方阵的特征值。
的值,也就是该方阵的特征值。
解得特征值之后如何求得特征向量:

找到特征值后,根据上式定义我们可推出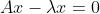 ,即
,即 ,该式中
,该式中 即为特征向量,
即为特征向量,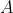 与
与 已知,解出上式即可求出方阵A的特征向量,且每个特征值
已知,解出上式即可求出方阵A的特征向量,且每个特征值 对应一个特征向量。
对应一个特征向量。
特征分解:

对于任一方阵A,其可根据上式被分解,P为由方阵的特征向量构成的方阵,D为一对角矩阵,其中当方阵A的特征向量是n维的基向量时对角矩阵D的值为方阵A的特征值。
注:特征分解和奇异值分解的区别
特征分解公式:
奇异值分解公式:
- SVD对任意矩阵都适用,而特征分解只适用于方阵
- 特征分解中的P矩阵不一定正交,而SVD中的U和V矩阵一定正交
- SVD中的U和V代表不同含义因此大部分情况下两者不互为对方的逆矩阵
实践中PCA应用的关键步骤:
- 标准化,对于数据的每一个特征减去其对应均值并除以其对应标准差
- 计算协方差矩阵,
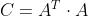
- 对协方差矩阵做特征分解得到其特征值和特征分解(在使用大部分机器学习库时这一步的输出为按照特征值由高到低排序后的结果)
- 根据选取的特征向量做投影,即把标准化后的数据投影到选取的特征向量上,Projection=

- 在下面的例子中我对投影后的数据进行了重建,即把投影后的数据转化为和原始数据相同的维度:Reconstruction=
 ,这一步在大部分实践中是不需要的,这一步的目的是为了比较使用PCA前后数据的变化
,这一步在大部分实践中是不需要的,这一步的目的是为了比较使用PCA前后数据的变化
PCA的应用
介绍完了PCA的原理下面我们来看看PCA在实际应用中的效果。我使用的数据集是不同国家每天的新冠确诊人数,在进行PCA之前我已经对数据集做了标准化处理。
我这里写了一个自定义函数,输入为每个国家对应的字符串,输出为对应国家每日确诊病例变化曲线(标准化后),依次使用不同数量的PC重建数据集的图像(max 10),使用不同数量PC重建数据与原始数据的残差图像(max 10),使用不同数量PC的RMSE曲线(max10),RMSE降低到1,0.1,0.01分别需要的PC数量。(这里PC为principal component,也就是特征向量)
from sklearn.metrics import mean_squared_error
import numpy as np
def pca(country):
#找出输入国家对应的index
country_index=np.where(cases_standardized.index==country)
fig,axes=plt.subplots(4,1,figsize=[10,20])
#提取出该国家对应的数据
country_data=cases_standardized.iloc[country_index[0][0],:]
axr=axes.ravel()
#在第一张图中画出对应国家的每日确诊病例曲线
axr[0].plot(country_data)
axr[0].set_title('Standardized Time Series')
labels=[x for x in range(0,265,30)]
axr[0].set_xticks(labels)
axr[0].set_xticklabels(dates[labels],rotation=30)
n,m=data_standardized.shape
#计算协方差矩阵
C = np.dot(data_standardized.T, data_standardized) / (n-1)
#使用numpy.linalg.eigh()计算协方差矩阵的特征值和特征向量
eigenValues, eigenVectors = np.linalg.eigh(C)
#提取排序后的index
args = (-eigenValues).argsort()
#把特征值和特征向量按照从高到低的数据排序
eigenValues = eigenValues[args]
eigenVectors = eigenVectors[:, args]
#提取该国家原始数据
true_value=data_standardized[country_index]
RMSE=[]
#使用不同数量的特征向量进行十次投影和重建
for i in range(10):
W=eigenVectors[:,:i+1]
projX=np.dot(data_standardized,W)
ReconX = np.dot(projX, W.T)
result=ReconX[country_index][0]
residual_error=true_value-result
#计算RMSE
RMSE.append(np.sqrt(mean_squared_error(true_value[0],result)))
#在第二张图中绘制重建后的数据图像
axr[1].plot(ReconX[country_index][0],label='CumPC'.format(i+1))
#在第三张图中绘制残差图像
axr[2].plot(residual_error[0],label='CumPC'.format(i+1))
axr[1].set_title('Cumulative Reconstruction')
labels=[x for x in range(0,265,30)]
axr[1].set_xticks(labels)
axr[1].set_xticklabels(dates[labels],rotation=30)
axr[1].legend()
axr[2].set_title('Residual Error')
axr[2].set_xticks(labels)
axr[2].set_xticklabels(dates[labels],rotation=30)
axr[2].legend()
#在第四张图中绘制RMSE曲线
axr[3].plot(RMSE)
axr[3].set_title('RMSE')
axr[3].set_xlabel('Number of component')
#找出使RMSE低于1需要的最少的特征向量数
for i in range(10):
if RMSE[i] < 1:
print('Number of PCs required for RMSE < 1: '.format(i+1))
break
#找出使RMSE低于0.1需要的最少的特征向量数
for i in range(10):
if RMSE[i] < 0.1:
print('Number of PCs required for RMSE < 0.1: '.format(i+1))
break
#找出使RMSE低于0.01需要的最少的特征向量数
for i in range(10):
if RMSE[i] < 0.01:
print('Number of PCs required for RMSE < 0.01: '.format(i+1))
break
#如果十个特征向量不能满足RMSE低于0.01的条件则继续使用更多的特征向量进行投影和重建直到找到能使RMSE低于0.01的特征向量数
if RMSE[9] >= 0.01:
for i in range(10,265):
W=eigenVectors[:,:i+1]
projX=np.dot(data_standardized,W)
ReconX = np.dot(projX, W.T)
result=ReconX[country_index][0]
RMSE=np.sqrt(mean_squared_error(true_value[0],result))
if RMSE < 0.01:
print('Number of PCs required for RMSE < 0.01: '.format(i+1))
break
我们把‘China’输入这个函数来看一下输出。

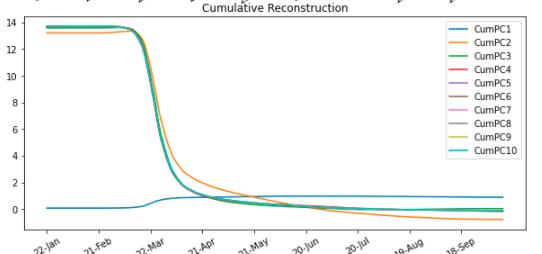

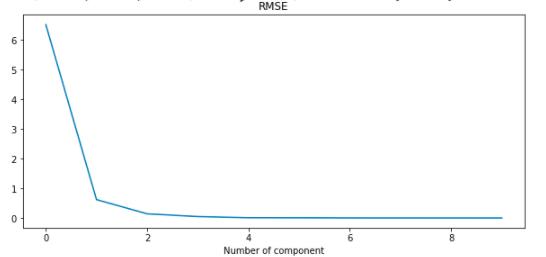

可以看到只使用一个PC时对于原数据的还原不是很理想,但是在使用两个PC时重建后的数据已经和原始数据非常相似了,而使用5个PC就可以使原数据与重建数据之间的RMSE低于0.01。
通过以上的例子我们可以发现,使用PCA可以把一个非常高维的数据集降为低维,在上面的例子中原数据集中包含了189个国家265天的数据,而我们只使用5个主成分就可以几乎完美地拟合原数据集。但PCA最大的缺陷就在于把原数据集投影到主成分后数据的可解释性就变得极低,因此在使用PCA前就需要考虑特征的可解释性对于之后的建模和分析到底是不是必要的。
本文中公式推导部分来自于:Mathematics for Machine Learning https://github.com/mml-book/mml-book.github.io
以上是关于利用PCA进行数据降维的主要内容,如果未能解决你的问题,请参考以下文章